
Colloques du
Groupement des Anthropologistes de Langue Française (GALF)
Cerny V., Hajek M., Bruzek J., Cmejla R., Brdicka R., 2004, Relations génétiques des populations de langues tchadiques parmi les populations péri-sahariennes révélées par l’étude des séquences de l’ADN mitochondriale. Antropo, 7, 123-131. www.didac.ehu.es/antropo
Relations génétiques des populations de langues tchadiques parmi les populations péri-sahariennes révélées par l’étude des séquences de l’ADN mitochondriale
Genetic relations of Chadic speaking populations in subsaharian mtDNA sequences populations variability
Cerny V.1, Hajek M.2, Bruzek J.3, Cmejla R.4, Brdicka R.5
1Département d’anthropologie et d’écologie, Institut d’archéologie, Académie des sciences, Letenská 4, 118 01, Prague 1, République tchèque, e-mail: cerny@arup.cas.cz
2Département des sciences de l’environnement, Institut d’archéologie, Académie des sciences, Letenská 4, 118 01, Prague 1, République tchèque, e-mail: hajek@arup.cas.cz
3UMR 5199 – PACEA - Laboratoire d’anthropologie des populations du passé, Université de Bordeaux I, Avenue des facultés, 33405 Talence, France, e-mail: j.bruzek@anthropologie.u-bordeaux1.fr ; et Laboratoire d’Anthropologie, Faculté des Sciences Sociales, Université de la Bohême de l’Ouest, Plzen, République tchèque.
4Département de physiologie celulaire, Institut d’hématologie et de transfusion sanguine, U Nemocnice 1, 128 20 Prague 2, République tchèque, e-mail : racm@centrum.cz
5Département de génétique moléculaire, Institut d’hématologie et de transfusion sanguine, U Nemocnice 1, 128 20 Prague 2, République tchèque, e-mail : molgen@uhkt.cz
Mots-clés: Tchadique, mtDNA, diversité, distance génétique, Afrique, populations
Keywords: Chadic, mtDNA diversity, genetic distances, Africa, populations
Résumé
Le but principal de cet article est de présenter les nouvelles séquences de l’ADN mitochondrial (mtDNA) des populations parlant des langues tchadiques, d’analyser leur diversité génétique et d’établir leurs relations dans la zone géographique péri-saharienne. Les séquences de mtDNA de quatre populations (Hidé, Kotoko, Mafa et Masa) du Cameroun du Nord ont été obtenues par prélèvement des frottis buccaux des sujets vivant dans leur milieu d’origine. Les relations génétiques des populations parlant des langues tchadiques ont été étudiées à partir de leur séquences mitochondriales puis comparées à celles des populations péri-sahariennes dèjà publiées. Les résultats de ces analyses nous ont permis de préciser plusieurs points. Tout d’abord concernant les séquences HVS-I, les populations vivant aujourd’hui autour du Sahara se distinguent en deux groupes différents. Le premier est composé principalement de populations de l’Afrique du Nord mais aussi de quelques groupes de l’Afrique occidentale (Mauritaniens, Saharawi, Wolof, Serer). Le deuxième groupe est composé seulement de populations vivant au sud du Sahara. Pour ce groupe, nous pouvons constater une bonne corrélation entre les distances génétiques et géographiques, alors que l’appartenance ethnique ou linguistique ne joue pas un rôle très important. Les analyses statistiques et l’étude phylogéographiques des séquences mitochondriales des populations parlant des langues tchadiques montrent davantage de relations génétiques avec les populations de l’Afrique orientale qu’avec celles de l’Afrique centrale. Cette observation va dans le sens des études linguistiques et archéologiques démontrant les relations assez anciennes entre ces régions séparées aujourd’hui par les vastes étendus du Sahara.
Abstract
The main aims of this paper are to report new mitochondrial DNA (mtDNA) sequences of Chadic-speaking populations, to analyse their genetic diversity and to establish their relationships within the peri-Saharian area in respect of geography and languages. mtDNA sequences of four Chadic-speaking populations (Hide, Kotoko, Mafa and Masa) from Northern Cameroon were obtained from buccal swabs which were collected during anthropological research in the area. According to the molecular analyses, we can conclude some points. First, the HVS-I of the populations living today around Sahara distinguish themselves in two different groups. The first one consists mainly in populations of North Africa but also of some groups of western Africa (Mauritanian, Saharawi, Wolof, Serer). The second group consists only in populations living in the South of Sahara. For this group, we can notice a good correlation between the genetic and geographic distances, while the ethnic or linguistic membership plays a very important role. The statistical analyses and the phylogeographic analysis of mtDNA sequences of Chadic-speaking populations show more genetic relations with the populations of Eastern Africa than with those of central Africa. This observation goes in the direction of the linguistic and archaeological studies which demonstrate the old relations between these regions, separates nowadays by vast land of Sahara.
Introduction
Cela fait plus de dix ans que l’on sait que les populations subsahariennes montrent un plus grand degré d’hétérogénéité génétique que toutes les autres. Cette constatation a servi d’argument pour conforter la théorie monophylétique de l’origine africaine de l’homme anatomiquement moderne (Cann et al. 1987 ; Excoffier et al. 1987 ; Vigilant et al. 1991). En ce qui concerne les populations africaines elles-mêmes, diverses études ont montré que leur histoire démographique plus récente (migrations sur le continent africain) peut être révélée par des études de la diversité génétique des populations autochtones (Watson et al. 1996, 1997 ; Mateu et al. 1997 ; Krings et al. 1999). Il existe deux façons d’échantillonner une population pour l’étude génétique. La première consiste à recueillir des échantillons d’ADN dans les hôpitaux ou les centres de transfusion sanguine, des grandes agglomérations, auprès des personnes le désirant. Malheureusement, l’origine géographique et l’appartenance ethnique de ces individus sont données sans être réellement vérifiables. Ceci concerne la majorité des données publiées. La seconde façon d’obtenir des échantillons est d’aller sur le terrain à la rencontre directe de la population à étudier. Dans ce cas-là, la localisation géographique exacte d’individus examinés ainsi que leurs parentés peuvent être repérées d’une manière beaucoup plus fiable. Alors que le premier mode d’échantillonnage est bien adapté à l’étude des différences génétiques entre les continents, les grands espaces ou les phénomènes démographiques plus anciens, le deuxième est indispensable pour analyser l’histoire démographique plus récente ou d'une région limitée.
L’histoire démographique et l’origine biologique des populations de langues tchadiques n’ont pas été encore abordées d’une manière systématique. Il s’agit d’un groupe linguistique dont les représentants sont aujourd’hui installés dans la partie méridionale du Bassin du Tchad, au Nigeria (branche occidentale), au Cameroun (branche central) et au Tchad (branche orientale). Ce groupe appartient, selon la classification linguistique, à la famille afro-asiatique. Aussi, il paraît intéressant de voir quelles sont ses relations biologiques avec les autres populations de cette famille et de chercher si elles recoupent les informations linguistiques. Autrement dit, nous voudrions trouver de quelle population péri-sahariennes actuelles, les populations tchadiques se rapprochent le plus.
Nous avons choisi l’ADN mitochondrial afin de répondre à notre interrogation. Il présente plusieurs avantages – elle n'est héritée que par le voie matrilinéaire (pas de recombinaison) et les mutations dans le premier segment hypervariable (HVS-I) de région de contrôle sont accumulés à une vitesse plus importante que dans les autres parties du génome humaine (différentiation possible entre les populations plus proches).
Matériel et méthodes
Nous avons visité les représentants de branche centrale qui habitent dans les deux types de l’environnement soudano-sahélienne. Alors que les Mafa et les Hidé – des montagnards – vivent dans les Monts Mandara près de la frontière Cameroun/Nigeria, les Kotoko et les Masa – des riverains – vivent autour des fleuves Chari et Logone près de la frontière Cameroun/Tchad (Figure 1).
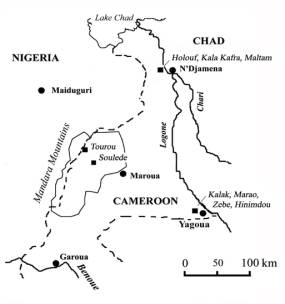
Figure 1. La situation géographique des populations de langues tchadiques au Cameroun du Nord (les Hidé de Tourou, les Mafa de Souledé, les Kotoko de Houlouf, Kala Kafra et Maltam et les Masa de Kalak, Marao, Zebe et Hinimdou).
Figure 1. Geographic localisation of the Chadic speaking populations from North Cameroon (the Hide from Tourou, the Mafa from Soulede, the Kotoko from Houlouf, Kala Kafra and Maltam and the Masa from Kalak, Marao, Zebe and Hinimdou).
Pendant les missions dans ces régions, nous avons prélevé des frottis buccaux (VC) et nous avons mesuré les principales dimensions anthropométriques (JB). Les représentants de différentes familles, des clans et des tribus ont été sélectionnés pour que seulement les individus non apparentés figurent dans notre échantillon. Avec l’assistance des interprètes locaux, nous avons obtenu un certain nombre d’échantillons biologiques dans les villages mono-ethniques séparés géographiquement entre eux approximativement 200 km, sauf pour les montagnards Mafa et Hidé qui sont les voisins plus proches (50 km).
L’extraction
de l’ADN a été effectuée à partir du tampon de lyse (50mM Tris pH 8.0, 50mM
EDTA pH 8.0, 50mM saccharase, 100mM NaCl, 1% SDS) où les échantillons ont été
conservés au terrain. L'isolation a été réalisée en laboratoire par QIAquick
PCR purification kit (QIAGEN) selon le protocole
recommandé dans le mode d’emploi. L’amplification génique du HVS-I a été menée
en utilisant les amorces L15995 (5’-CTC CAC CAT TAG CAC CCA AAG) et H16401
(5’-TTG ATT TCA CGG AGG ATG GTG) dans le volume total de 25 ![]() .
Les conditions ont été suivantes : 94°C 5 minutes, 35 cycles de 94°C 50
secondes, 51°C 30 secondes et 72°C 45 secondes, l’extension finale a été faite
á 72°C pour 5 minutes.
.
Les conditions ont été suivantes : 94°C 5 minutes, 35 cycles de 94°C 50
secondes, 51°C 30 secondes et 72°C 45 secondes, l’extension finale a été faite
á 72°C pour 5 minutes.
La re-isolation de fragments amplifiés du gel a été faite à l’aide du QIAquick gel extraction kit (QIAGEN) selon le protocole recommandé dans le mode d’emploi. En ce qui concerne le séquençage du HVS-I, nous avons appliqué la chimie BigDye Terminator. Nous avons ensuite aligné les séquences à l’aide du logiciel BioEdit qui nous a facilité l’identification des mutations (ou bien des différences) par rapport à la séquence de référence (CRS - Anderson et al. 1981). Les échantillons contenants de la heteroplasmie de longueur (en majorité de cas ceux avec la chaîne homopolymeric de cytosines du 16184 à 16193) ont été complétés par le séquençage de la chaîne opposée.
La région codante de l’ADN mitochondriale, où les mutations sont rares mais phylogénétiquement plus importantes, a été analysé par la digestion enzymatique des amplicons dont les amorces ont été projetées pour inclure les régions voisines. Le polymorphisme a été classé comme présent ou absent selon la séparation éléctrophorétique des fragments clivés. Nous avons concentré notre attention sur trois sites (le 3592 clivée par HpaI, le 2349 clivé par MboI et le 16390 clivée par AvaII) rapportés récemment comme informatives pour les populations d’origine subsaharienne (Alves-Silva et al. 2000, Pereira et al. 2001, Salas et al. 2002). La partie englobante le site 3592 a été amplifiée par les amorces L03526 (5´-CAT CAC CCT CTA CAT CAC CG et H03706 (5´-ATT GTT TGG GCT ACT GCT CG), la partie englobante le site 2349 par les amorces L02224 (5´-TTC AAG CTC AAC ACC CAC TAC) et H02526 (5´-AGG CGG TGC CTC TAA TAC TG) et la partie englobante le site 16390 par les amorces L16259 (5´-AAC TGC AAC TCC AAA GCC AC et (H16529 5´-GGG AAC GTG TGG GCT ATT TA). Les conditions pour le 3592 ont été suivantes : 94°C 5 minutes denaturation initiale, 35 cycles de 94°C 50 secondes (denaturation), 55°C 30 secondes (annelation) et 72°C 30 secondes (extension), l’extension finale a été faite á 72°C pour 5 minutes. Les conditions pour le 2349 ont été suivantes : 94°C 5 minutes denaturation initiale, 35 cycles de 94°C 50 secondes (denaturation), 53°C 30 secondes (annelation) et 72°C 1 minute (extension), l’extension finale a été faite á 72°C pour 5 minutes. Les conditions pour le 16390 ont été suivantes : 94°C 5 minutes denaturation initiale, 35 cycles de 94°C 50 secondes (denaturation), 55°C 30 secondes (annelation) et 72°C 1 minute (extension), l’extension finale a été faite á 72°C pour 5 minutes.
Selon les informations obtenues (le motif du HVS-I et les mutations de points dans la région codante), nous avons pu diviser notre échantillon en quatre haplogroupes du type L, dont la répartition actuelle sur le continent africaine et l’origine ancienne ont été proposées par les études récentes (Watson et al. 1996, 1997; Krings et al. 1999; Torroni et al. 2001; Mateu et al. 1997; Rando et al. 1998; Bandelt et al. 2001; Salas et al. 2002).
Comme matériel de comparaison, nous avons choisi les séquences HVS-I des populations péri-sahariennes publiées auparavant et disponibles sur l’internet. Pour toutes les populations, nous avons calculé des indices de la diversité moléculaire à l’aide du logiciel ARLEQUIN (Schneider et al. 2000). L’application de l’analyse de variance moléculaire (AMOVA) a été ajustée d’une manière suivante: nous avons regroupé les populations dans les cinq groupements régionaux et cherché quelle combinaison d’un groupe ainsi défini avec le groupe tchadique augmente la variance parmi les groupes par rapport à l’analyse où les populations tchadiques ont été prises comme un groupement indépendant. Pour mieux aborder les différentiations génétiques entre les populations analysées, nous avons également calculé les distances FST qui ont servi comme une matrice pour des analyses ultérieures – classification hiérarchique et analyse de proximité faites par le logiciel STATISTICA.
Pour étudier les relations phylogénétiques au sein des séquences mitochondriales (haplotypes), nous avons utilisé l’analyse Reduced Median Network disponible dans le logiciel Network (Bandelt et al. 1995). Puisque le HVS-I (région de contrôle) contient des sites à fort taux de mutation (mutations appelées aussi hotspots), nous avons été obligés de minimaliser leurs poids. A l’opposé, nous avons augmenté le poids des mutations de la région codante.
Résultats
Les différents paramètres génétiques (le nombre des haplotypes différentes, le nombre de positions polymorphiques, la diversité des séquences, la diversité nucléique et le nombre moyen de différences de paires entre chaque individu) montrent que les populations parlant des langues tchadiques sont plus proches des populations d’Afrique orientale qui donnent en même temps le plus grand degré de la diversité génétique (Tableau 1).
Nous pouvons aussi constater que notre type d’échantillonnage fournit le même niveau de diversité génétique que les échantillonnages faits dans les hôpitaux et qu’il n’est donc pas influencé par l’inclusion d’individus apparentés. Nous avons aussi testé le modèle de population évolué en effectif constant par la statistique de Tajima et de Fu. Toutes les valeurs sont négatives et le test Fs de Fu donne les résultats hautement significatifs. Cela peut être interprété comme l’indication de l’expansion démographique passée des populations examinées. Cette observation a été constatée chez les autres populations sédentaires ou nomades mais non chez certaines populations chasseurs-collecteurs (Pereira et al. 2000).
L’analyse de variance moléculaire a fourni des résultats intéressants (Tableau 2). L’augmentation de la variance parmi les groupes a été relevée seulement pour le groupement tchadiques – Afrique orientale (groupement A+D,B,C,E,F) et non pour le groupement tchadiques – Afrique centrale (A+B,C,D,E,F) qui se situe géographiquement plus proche. A l’opposé, la réduction drastique de la variance parmi les groupes est produite pour le groupement des tchadiques avec les populations maghrébines (groupement A+E,B,C,D,F).
La représentation des distances génétiques (FST) entre les populations étudiées a été réalisée par l’analyse de proximité considérée généralement comme une alternative a l’analyse factorielle (Figure 2). La contrainte assez basse (0,078) rapporte que la configuration reproduit assez bien la matrice des distances observées. Sur ce graphique nous pouvons voir non seulement les distances entre les populations de deux côtés du Sahara (les populations subsahariennes en bas, les autres en haut) mais aussi les relations de l’ouest à l’est chez les premières. Il faut souligner que les populations tchadiques se situent proches des populations orientales (comme les Turkana, les Kikuyu, les Nilotic et les Somali) ce qui confirme les résultats de l’AMOVA. On voit ici que les distances génétiques que nous avons obtenues par l’étude de l’ADN mitochondrial (HVS-I) suivent dans la région subsaharienne assez bien les distances géographiques alors que la repartition des familles linguistiques est assez aléatoire.
|
Population |
reference |
LF |
n |
k |
S |
hs |
π |
Dii |
IH |
pIH |
|
Hide |
Cette étude |
AA |
25 |
23 |
47 |
0,993 |
0,026 |
8,863 |
0,012 |
0,610 |
|
Kotoko |
Cette etude |
AA |
16 |
11 |
39 |
0,942 |
0,024 |
8,175 |
0,024 |
0,724 |
|
Mafa |
Cette etude |
AA |
32 |
23 |
55 |
0,980 |
0,024 |
8,260 |
0,019 |
0,108 |
|
Masa |
Cette etude |
AA |
31 |
28 |
42 |
0,991 |
0,024 |
8,092 |
0,007 |
0,734 |
|
Turkana |
Watson et al. 1997 |
NS |
37 |
35 |
65 |
0,996 |
0,032 |
10,887 |
0,007 |
0,558 |
|
Somali |
Watson et al. 1997 |
AA |
27 |
24 |
48 |
0,992 |
0,024 |
8,399 |
0,008 |
0,852 |
|
Kikuyu |
Watson et al. 1997 |
NC |
25 |
23 |
50 |
0,993 |
0,026 |
8,890 |
0,009 |
0,898 |
|
Nubian |
Krings et al. 1999 |
NS |
78 |
51 |
74 |
0,973 |
0,025 |
8,721 |
0,005 |
0,876 |
|
Nilotic |
Krings et al. 1999 |
NS |
72 |
61 |
78 |
0,995 |
0,026 |
8,765 |
0,004 |
0,808 |
|
Hausa |
Watson et al. 1997 |
AA |
20 |
19 |
35 |
0,995 |
0,019 |
6,447 |
0,010 |
0,938 |
|
Kanuri |
Watson et al. 1997 |
NS |
14 |
13 |
36 |
0,938 |
0,022 |
7,593 |
0,018 |
0,750 |
|
Fulbe |
Watson et al. 1997 |
NC |
61 |
39 |
48 |
0,973 |
0,021 |
7,282 |
0,009 |
0,557 |
|
Songhai |
Watson et al. 1997 |
NS |
10 |
9 |
32 |
0,978 |
0,028 |
9,600 |
0,041 |
0,559 |
|
Tuareg |
Watson et al. 1997 |
AA |
26 |
21 |
42 |
0,985 |
0,022 |
7,375 |
0,012 |
0,598 |
|
Yoruba |
Watson et al. 1997 |
NC |
33 |
31 |
48 |
0,996 |
0,023 |
7,864 |
0,009 |
0,716 |
|
Serer |
Watson et al. 1997 |
NC |
23 |
20 |
37 |
0,988 |
0,015 |
5,233 |
0,047 |
0,093 |
|
Wolof |
Watson et al. 1997 |
NC |
48 |
40 |
75 |
0,992 |
0,020 |
7,033 |
0,013 |
0,355 |
|
Mandenka |
Graven et al. 1995 |
NC |
79 |
29 |
44 |
0,947 |
0,019 |
6,361 |
0,011 |
0,650 |
|
Saharawi |
Rando et al. 1998 |
AA |
25 |
20 |
32 |
0,973 |
0,013 |
4,547 |
0,075 |
0,020 |
|
Mauritanian |
Rando et al. 1998 |
AA |
30 |
23 |
40 |
0,975 |
0,015 |
4,949 |
0,032 |
0,136 |
|
Egyptian Lower |
Krings et al. 1999 |
AA |
59 |
53 |
68 |
0,994 |
0,022 |
7,628 |
0,008 |
0,671 |
|
Egyptian Upper |
Krings et al. 1999 |
AA |
35 |
32 |
59 |
0,995 |
0,024 |
8,247 |
0,004 |
0,976 |
|
Moroccan Berber |
Rando et al. 1998 |
AA |
60 |
38 |
53 |
0,963 |
0,016 |
5,434 |
0,011 |
0,737 |
|
Moroccan Arabs |
Rando et al. 1998 |
AA |
32 |
29 |
44 |
0,988 |
0,020 |
6,683 |
0,018 |
0,272 |
|
Berber Souss |
Brakez et al. 2001 |
AA |
50 |
34 |
38 |
0,961 |
0,014 |
4,604 |
0,010 |
0,863 |
|
Mozabite |
Côrte-Real et al. 1996 |
AA |
85 |
30 |
37 |
0,943 |
0,016 |
4,822 |
0,018 |
0,298 |
Tableau 1. Les différents paramètres génétiques du HVS-I de 26 populations péri-sahariennes. LF – famille linguistique (NC: Nigero-Congolais, AA: Afro-Asiatique, NS: Nilo-Saharien); n – effectif ; k – le nombre des haplotypes différentes, S – le nombre de positions polymorphiques, hs – la diversité des séquences, π – diversité nucléique, Dii – le nombre moyen de différences de paires entre chaque individu, IH – indice d’Harpending, pIH – probabilité de l’indice d’Harpending.
Table 1. Diversity indices for HVS-I in 26 African populations. LF - language family (NC: Niger-Congo, AA: Afro-Asiatic, NS: Nilo-Saharan); n - sample size ; k - number of different sequences; S - number of polymorphic sites; hs - gene (sequence) diversity; π - nucleotide diversity; π - nucleotide diversity; Dii - mean number of pairwise differences (mismatch observed mean); V(Dii) - mismatch observed variance; IH - Harpending Raggedness index; pIH - probability of Harpending Raggedness index.
|
% de la variance parmi les groupes |
% de la variance dans les groupes |
% de la variance dans les populations |
|
|
A,B,C,D,E,F |
5,09 |
4,46 |
90,44 |
|
A+B,C,D,E,F |
5,04 |
4,67 |
90,29 |
|
A+C,B,D,E,F |
4,09 |
5,50 |
90,41 |
|
A+D,B,C,E,F |
5,15 |
4,65 |
90,20 |
|
A+E,B,C,D,F |
2,96 |
6,45 |
90,59 |
|
A+F,B,C,D,E |
4,15 |
5,36 |
90,49 |
Tableau 2. L’analyse de variance moléculaire.
Groupements (Groupings)
B) Central (Central): Kanuri, Fulbe, Hausa, Yoruba, Songhai, Tuareg
C) Occidental (Western): Serer, Wollof, Mandenka, Mauritanian, Saharawi
D) Oriental (Eastern): Nilotic, Somali, Kikuyu, Nubian, Turkana
E) Nord-ouest (North-western): Moroccan Arabs, Moroccan Berbers, Berbers Souss, Mozabite
F) Nord-est (North-eastern): Egyptians Lower, Egyptians Upper
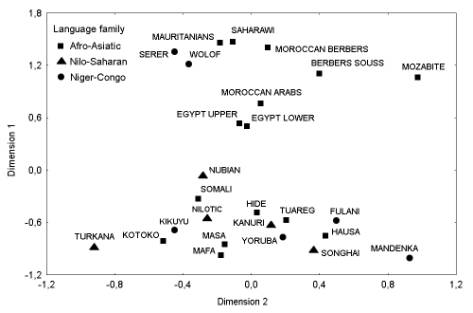
Figure 2. L’analyse de proximité de 26 populations analysées pour le HVS-I. de mtADN
Figure 2. Multidimensional scaling analysis of 26 populations analyzed for mtDNA (HVS-I).
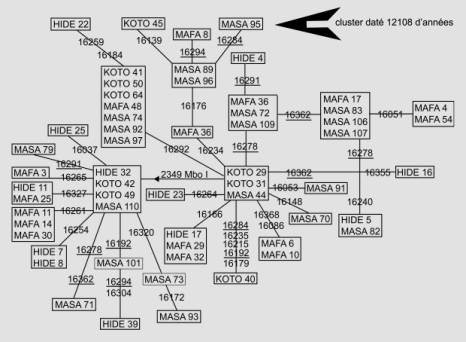
Figure 3. Le réseau d’haplotypes L3 révélés chez les populations de langues tchadiques.
Figure 3. The network of L3 haplotypes revealed in the Chadic speaking populations.
Selon la résolution moléculaire que nous avons obtenue jusqu’à nos jours, nous pouvons attribuer les haplotypes des échantillons tchadiques aux haplogroupes publiées par les travaux phylogénétiques. Pour les 106 haplotypes du groupe tchadique, nous n’en trouvons qu’un seul d’origine européenne. Il s’agit d’une séquence qui s’arrange avec le CRS (séquence de référence de Cambridge), donc avec le haplogroupe H. Tous les autres peuvent être classés parmi les haplogroupes d’origine subsaharienne de type L. Il faut souligner que l’haplogroupe L3 dont l’origine et la plus grande diversité se trouvent en Afrique orientale est également le plus fréquent (57%) dans notre échantillon. A partir de logiciel Network, nous avons construit un réseau d’haplotypes L3 (Figure 3).
Dans ce réseau, nous pouvons identifier un cluster commencant chez les séquences «masa 89» et «masa 96» qui n’a pas été encore publié ailleurs. La date de l’expansion démographique qui pouvait être responsable pour cette diversification a été estimée par le Network à 12108 ± 6990 d’années.
Discussion
Comment expliquer les relations des populations tchadiques du Cameroun septentrional avec les populations d’Afrique orientale? Une explication peut être apportée par la linguistique historique et l’archéologie. La reconstruction des langues de la famille afro-asiatique a abouti à une hypothèse sur l’origine des langues tchadiques (Blench 1999). Selon cette hypothèse, l’origine des ancêtres des populations de langues couchitiques et tchadiques se trouverait dans la région du Nil moyen, dans la région du Khartoum, il y a approximativement 6000 ans. A partir de ce centre commun, deux vagues migratoires se seraient succédées ensuite – la première vers le sud aurait abouti à l’origine et la diversification des langues couchitiques, par contre, la seconde vers l’ouest aurait donné naissance aux langues tchadiques. Les traces archéologiques (céramiques) de la migration occidentale sont visibles le long de Wadi Howar au Soudan (Kuper 1981 ; Keding 1993). Malheureusement, la région du Tchad n’a pas été encore prospectée. Il est possible que les premiers groupes des pasteurs de langues tchadiques soient venus au bord du Lac Tchad il y a 4000 ans. Il est pour l’instant prématuré de voir dans la diversification du cluster révélé parmi les haplotypes L3 une preuve évidente d’une expansion des populations de langues tchadiques. Néanmoins, il parait évident que l’étude à l’échelle locale de la structure génétique des populations vivant actuellement dans la zone de migration présumée peut nous fournir des informations supplémentaires concernant le peuplement de cette partie du monde.
Références bibliographiques
Anderson, S., Bankier, A.T., Barrell, B.G., Bruijn, M.H.L., Coulson, A.R., Drouin, J., Eperon, I.C., Nierlich, D.P., Roe, B.A., Sanger, F., Schreier, P.H., Smith, A.J.H., Staden, R. and Young, I.G., 1981, Sequence and organization of the human mitochondrial genome. Nature, 290, 457-465.
Bandelt, H.-J., Alves-Silva, J., Guimaraes, P.E., Santos, M.S., Brehm, A., Pereira, L., Coppa, A., Larruga, J.M., Rengo, C., Scozzari, R., Torroni, A., Prata, M.J., Amorim, A., Prado, V.F., Pena, S.D., 2001, Phylogeography of the human mitochondrial haplogroup L3e: a snapshot of African prehistory and Atlantic slave trade. Annals of Human Genetics, 65, 549-563.
Bandelt, H.-J., Forster, P., Sykes, B.C., Richards, M.B., 1995, Mitochondrial portraits of human populations using median networks. Genetics, 141, 743-753.
Blench, R., 1999, The westward wandering of Cushitic pastoralists. Explorations in the prehistory of Central Africa. In L’Homme et l’animal dans le bassin du lac Tchad, edited by C. Baroin and J. Boutrais (Paris: Editions IRD) p. 39-80.
Cann, R., Stoneking, M. and Wilson, A., 1987, Mitochondrial DNA and human evolution. Nature, 325, 31-36.
Excoffier, L., Pellegrini, B., Sanchez-Mazas, A., Simon, Ch., and Langaney, A., 1987, Genetics and history of sub-Saharan Africa. Yearbook of Physical Anthropology, 30, 151-194.
Keding, B., 1993, Leiterband sites in the Wadi Howar, North Sudan. In Environmental Change and Human Culture in the Nile Basin and Northern Africa until the Second Millennium B.C., edited by L. Krzyzaniak, L. Kobusiewicz and J. Alexander (Poznan: Poznan Archaeological Museum) p. 371-380.
Krings, M., Salem, A.H., Bauer, K., Geisert, H., Malek, A.K., Chaix, L., Simon, Ch., Welsby, D., Di Rienzo, A., Utermann, G., Sajantila, A., Pääbo, S. and Stoneking, M., 1999, mtDNA analysis of Nile river valley populations: a genetic corridor or a barrier to migration? American Journal of Human Genetics, 64, 1166-1176.
Kuper, R., 1981, Untersuchungen zur Besiedlunsgeschiche der östlichen Sahara. Vorbericht über die Expedition 1980. Beiträge zur Allgemeinen und Vergleichenden Archeologie, 3, 215-275.
Rando, J.C., Pinto, F., Gonzalez, A.M., Hernandez, M., Larruga, J.M., Cabrera, V.M. and Bandelt, H.-J., 1998, Mitochondrial DNA analysis of northwest African populations reveals genetic exchanges with European, near-eastern, and sub-Saharan populations. Annals of Human Genetics, 62, 531-550.
Salas, A., Richards, M., de la Fe, T., Lareu, M.V., Sobrino, B., Sanchez-Diz, P., Macaulay, V., Carracedo, A., 2002, The making of the African mtDNA landscape. American Journal of Human Genetics, 71, 1082-1111.
Schneider, S., Roessli, D., and Excoffier, L., 2000, Arlequin ver. 2.000: A Software for Population Genetics Data Analysis (Genetics and Biometry Laboratory, University of Geneva, Switzerland).
Torroni, A., Chiara, R., Guida, V., Cruciani, F., Sellitto, D., Coppa, A., Calderon, F.L., Simionati, B., Valle, G., Richards, M., Macaulay, V., Scozzari, R., 2001, Do the four clades of the mtDNA haplogroup L2 evolve at different rates ? American Journal of Human Genetics, 69, 1348-1356.
Vigilant, L., Stoneking, M., Harpending, H., Hawkes, K. and Willson, A.C., 1991, African human populations and the evolution of human mitochondrial DNA. Science, 253, 1503-1507.
Watson, E., Bauer, K., Aman, R., Weiss, G., von Haeseler, A., and Pääbo, S., 1996, mtDNA sequence diversity in Africa. American Journal of Human Genetics, 59, 437-444.
Watson, E., Forster, P., Richards, M. and Bandelt, H.-J., 1997, Mitochondrial footprints of human expansions in Africa. American Journal of Human Genetics, 61, 691-704.