
Calò, C.M., Corrias, L., Bachis, V.,
Vona, G., Brandas, A., Scudiero, C.M., Di Fede, C., Mameli, A., Robledo, R., 2013. Analisi di due
isolati della Sardegna (Italia) attraverso lo studio dei polimorfismi del
cromosoma Y. Antropo, 29, 1-7. www.didac.ehu.es/antropo
Analisi di due isolati della
Sardegna (Italia) attraverso lo studio dei polimorfismi del cromosoma Y
Analysis of two
Sardinian isolates (Italy) through Y chromosome polymorphisms
C. M. Calò1, L. Corrias1,
V. Bachis1, G. Vona1, A. Brandas1, C. M. Scudiero2,
C. Di Fede2, A. Mameli2, R. Robledo3
1Dip. Scienze della vita e dell’Ambiente, Università di
Cagliari
2RIS, Raggruppamento Investigazioni Scientifiche dei
Carabinieri, Cagliari
3Dip. Scienze Mediche, Università di Cagliari
Corrispondenza: Carla Maria Calò. Dip. Scienze della Vita e
dell’Ambiente, Università di Cagliari, SS 554 km4.500 – 09042 Monserrato
(Ca). E-mail: cmcalo@unica.it
Parole chiave: Sardegna, isolati genetici, Y-STR.
Key words: Sardinia, genetic isolates, Y-STR.
Riassunto
Nel presente lavoro sono stati analizzati due isolati della Sardegna (Italia): l’isolato linguistico di Carloforte e l’isolato geografico di Benetutti attraverso gli Y-STRs e gli aplogruppi derivati, con lo scopo di valutare il grado di isolamento e le diverse influenze delle barriere geografiche e culturali sull’isolamento e la struttura genetica. Gli individui campionati sono stati selezionati con il metodo dei cognomi fondatori. Sono stati selezionati anche due campioni di confronto provenienti dalle aree geografiche più vicine agli isolati studiati. Il DNA è stato estratto da sangue periferico o tampone buccale e amplificato mediante il kit AmpFISTR Yfiler con il ciclatore termico Geneamp®PCR System 9700. La separazione e l’individualizzazione dei prodotti della PCR multiplex sono state realizzate con il sistema ABI Prism 3100 Genetic Analyzer. L’analisi dei dati è stata infine realizzata con GeneScan v. 3.1. Gli aplogruppi sono stati stimati mediante Haplogroup predictor. Attraverso l’analisi dei 17 Y-STRs entrambi gli isolati appaiono significativamente differenziati dalle aree circostanti. L’analisi degli aplogruppi rivela una distribuzione caratteristica: la popolazione di Carloforte è caratterizzata dalla bassissima frequenza dell’aplogruppo I2a1. Questo aplogruppo è identificato dalla mutazione M26, caratteristica della Sardegna, e quasi assente nel resto d’Italia. Il confronto con altre popolazioni italiane, realizzato con l’albero Neighbor joining, ha confermato la peculiarità genetica della Sardegna. Carloforte, nonostante sia in una branca separata, si trova nello stesso cluster delle popolazioni italiane, mentre Benetutti, sempre in un ramo separato, si colloca più vicina alle popolazioni sarde.
In conclusione, sembra che le barriere linguistiche e culturali abbiano la stessa influenza delle barriere geografiche sull’isolamento e come conseguenza sulla struttura genetica di una popolazione.
Abstract
In this paper we analyzed two Sardinian isolates (Italy): the linguistic isolate of Carloforte and the geographic isolate of Benetutti through Y-STRs and Y-haplogroups with the aim to evaluate the degree of isolation and the different influence of cultural and geographical boundaries on isolation. Individuals from Carloforte and Benetutti were all selected with the method of founder surnames. We also selected two comparison samples from the geographical areas closer to studied isolates. DNA was extracted from peripheral blood and buccal swab and amplified with the AmpFISTR Yfiler PCR amplification kit in a Geneamp®PCR System 9700 thermal cycler. Separation and detection of Y-STR 17plex PCR products were accomplished with the ABI Prism 3100 Genetic Analyzer sixteen-capillary array system. Analysis of the data was performed using GeneScan v. 3.1. Haplogroups were inferred by Haplogroup predictor. Through the analysis of Y-STRs, both the isolates appear significantly differentiated from the neighboring areas. The analysis of haplogroup reveals a peculiar distribution: Carloforte population is characterized by the very low frequency of haplogroup I2a1. This haplogroup is identified by the mutation M26, characteristic of Sardinia, and almost absent in the rest of Italy. Comparison with other Italian populations, carried out with genetic tree, confirms the genetic peculiarity of Sardinian populations. Carloforte, despite being on a separate branch, is in the same cluster of Italian populations, while Benetutti, closer to the other two Sardinian populations, is collocated on a separate branch as an outlier.
In conclusion, it seems that cultural and linguistic barriers have the same influence of the geographic boundaries on isolation and as a consequence on the genetic structure of a population.
Introduzione
Le popolazioni isolate sono campioni ideali per lo studio di fenomeni evolutivi (migrazioni, popolamento) e per la ricostruzione di genealogie (Colonna et al., 2007).
Inoltre, gli isolati genetici rappresentano
eccezionali risorse per gli studi di associazione e per il mappaggio di tratti
complessi.
La popolazione sarda e i suoi villaggi sono stati proposti come popolazioni adatte per il mappaggio di geni coinvolti in malattie multifattoriali (Zavattari et al., 2000, Angius et al., 2001), a causa della loro peculiare struttura genetica e dell’isolamento. La popolazione sarda è caratterizzata da una forte variabilità dovuta a: (1) barriere geografiche e linguistiche; (2) alto tasso di endogamia e consanguineità; (3) presenza in passato della malaria che ha esercitato una forte pressione selettiva (Cappello et al., 1996; Gatti, 1990; Kosoy et al., 2011; Calò et al., 2008). All’interno dell’isola, le differenze genetiche sono state determinate dalla deriva genetica, poiché le barriere naturali e l’orografia della regione hanno mantenuto alcune aree estremamente isolate.
In questo lavoro sono stati analizzati due isolati della Sardegna (Fig. 1): l’isolato linguistico di Carloforte e l’isolato geografico di Benetutti attraverso 17 Y-STRs e gli aplogruppi derivati, con l’intento di valutare il grado di isolamento e la differente influenza delle barriere culturali e geografiche sull’isolamento.
Carloforte
(6.444 abitanti), localizzata nella piccola isola di San Pietro, è una comunità
alloglotta fondata nel 1738 da rifugiati originari della Liguria che provenivano
dall’isola tunisina di Tabarka. Ancora oggi il linguaggio parlato è l’antico
dialetto di Pegli (Liguria).
Benetutti, un piccolo villaggio di 2.181 abitanti, è localizzato in un’area del nord Sardegna, il Goceano, caratterizzato da un forte isolamento. I primi dati demografici documentati (Quinque Libri) risalgono al 1618.
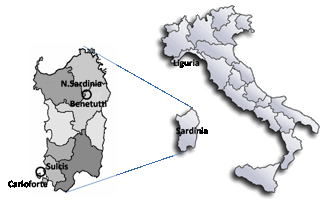
Figura 1. Localizzazione geografica di Carloforte e Benetutti.
Figure 1. Geographical location of Carloforte and Benetutti.
Materiali e Metodi
Gli individui campionati di Carloforte (N=42) e Benetutti (N=48) sono stati tutti selezionati con il metodo dei cognomi fondatori. Sono stati identificati i cognomi presenti al momento della fondazione del comune di Carloforte (Vallebona, 1974), mentre per Benetutti, dove non era possibile conoscere i cognomi dei fondatori per mancanza di dati storici, sono stati identificati i cognomi più diffusi nel periodo più antico documentato (1600). Sono stati selezionati anche due campioni di confronto (popolazioni aperte): una del Sulcis-Iglesiente (N=48) e una del Nord Sardegna (N=48), rispettivamente le regioni più vicine a Carloforte e Benetutti.
Il DNA è stato estratto da sangue intero periferico o da tampone boccale ed è stato quantificato tramite fluorimetro (Qubit). L’amplificazione di 0,5 ng di DNA è stata realizzata con l’AmpFlSTR Yfiler PCR amplification kit (Applied Biosystems) dal ciclatore termico Geneamp®PCR System 9700 (Applied Biosystems), utilizzando le condizioni di PCR raccomandate dalla ditta. La separazione e l’identificazione dei prodotti della PCR multiplex è avvenuta tramite il sistema array a sedici capillari ABI Prism 3100 Genetic Analyzer (Applied Biosystems). I genotipi sono stati determinati con GeneScan v. 3.1. Gli aplogruppi sono stati stimati con Haplogroup predictor (http://www.hprg.com/hapest5/). Le analisi statistiche sono state realizzate attraverso i programmi Arlequin (ver. 3.11) e Phylip (ver. 3.76).
Risultati e discussione
I principali indici di diversità calcolati per i 17 Y-STRs sono mostrati in tabella 1.
|
|
Carloforte |
Sulcis |
Benetutti |
N.Sardegna |
|
N. di aplotipi |
39/42 |
45/48 |
43/48 |
47/48 |
|
Diversità genica |
0,975 |
0,978 |
0,975 |
0,980 |
|
Diversità genica media |
0,603 |
0,622 |
0,593 |
0,633 |
|
Pairwise differences |
9,650 |
9,962 |
9,481 |
10,135 |
Tabella 1. Diversità genetica tramite i 17 Y-STRs nelle
popolazioni analizzate.
Table 1. Genetic diversity through 17 Y-STRs in the analyzed populations.
Per ogni popolazione, ma soprattutto per le popolazioni aperte, è stato riscontrato un elevato grado di diversità genica, considerando, infatti, che la diversità genica media calcolata per l’Europa (0,498, Jorde et al., 2000) è nettamente inferiore a quella calcolata per le popolazioni sarde.
Attraverso l’analisi degli STRs, entrambi gli isolati sono risultati significativamente differenziati dalle regioni circostanti (p<0,05). Carloforte condivide con il Sulcis solo un aplotipo, mentre nessun altro aplotipo è condiviso tra le popolazioni sarde esaminate.
Nelle popolazioni sarde sono stati trovati alleli off-ladder in 4 individui: 2 del Sulcis per il locus DYS458 (allele 13) e due di Benetutti per il YGATAH4 (allele 14), nonostante questi alleli siano stati trovati anche in altre popolazioni (Redd et al., 2002) risultano molto rari per le popolazioni italiane.
Un’ interessante osservazione riguarda gli alleli non-consensus osservati per il locus DYS458: tre individui (due di Benetutti e uno del Nord Sardegna) presentavano l’allele non-consensus 18.2 del marcatore DYS458, e questi erano gli unici individui appartenenti all’aplogruppo J1. Questo sembra confermare che questo polimorfismo può essere usato efficacemente per discriminare l’aplogruppo, come precedenti ricerche avevano suggerito (Alves et al., 2007; Myers et al., 2007; Ghiani et al., 2009).
L’analisi degli aplogruppi (Tab. 2) ha rivelato una distribuzione caratteristica: La popolazione di Carloforte si caratterizza per una bassissima frequenza dell’aplogruppo I2a1, presente in tutte le altre aree della Sardegna. Questo aplogruppo è identificato dalla mutazione M26, caratteristica della Sardegna, ma assente o presente a bassa frequenza nel resto d’Italia (Onofri et al., 2007; Rootsi et al., 2004; Semino et al., 2000; Scozzari et al., 2001) e in Corsica (Francalacci et al., 2003).
|
|
Carloforte |
Sulcis Iglesiente |
Benetutti |
Nord Sardegna |
|
R1a* |
- |
- |
- |
4% |
|
R1b* |
55% |
25% |
4% |
21% |
|
G2a* |
10% |
13% |
10% |
15% |
|
E1b1* |
7% |
4% |
15% |
4% |
|
E2a1 |
- |
- |
2% |
- |
|
J1* |
- |
- |
4% |
4% |
|
J2a1 |
5% |
6% |
10% |
10% |
|
J2b* |
- |
4% |
- |
2% |
|
T* |
2% |
- |
2% |
- |
|
I1* |
5% |
- |
- |
4% |
|
I2a* |
2% |
4% |
- |
- |
|
I2a1 |
2% |
40% |
51% |
34% |
|
I2b1 |
12% |
4% |
- |
- |
|
L* |
- |
- |
2% |
2% |
Tabella 2. Frequenze aplotipiche nelle popolazioni esaminate.
Table 2. Haplotype frequencies in the examined populations.
La frequenza dell’aplogruppo R1b raggiunge a Carloforte valori del 55%, simili ai valori riscontrati nel Nord Italia (Ferri et al., 2009; Semino et al., 2000), mentre nel resto della Sardegna esso mostra frequenze più basse (intorno al 20%), con il minimo della frequenza a Benetutti (4%).
E’ stato effettuato un confronto con altre popolazioni italiane tramite le distanze genetiche di Slatkin (1995) utilizzando unicamente 7 Y-STRs (DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393) per mancanza di dati reperibili in letteratura (Presciuttini et al., 2001; Di Gaetano et al., 2009). L’albero genetico ottenuto (Fig.2) ha confermato la peculiarità genetica delle popolazioni sarde. Carloforte, nonostante sia collocata in una branca separata, si trova molto più vicina alle popolazioni italiane, mentre Benetutti, che clusterizza con le altre popolazioni sarde, si posiziona comunque in un ramo più distante.
E’ importante notare che la popolazione ligure non clusterizza con Carloforte. Questo risultato potrebbe essere dovuto a differenti eventi: a) la popolazione fondatrice di Carloforte è stata soggetta a effetto collo di bottiglia che quindi ha modificato la composizione genetica rispetto alla popolazione madre; b) la differenziazione e l’evoluzione genica della popolazione della Liguria; c) in letteratura non è specificata l’esatta provenienza del campione ligure e non si può escludere una eterogeneità genetica della popolazione della Liguria.
Quando la Tunisia viene aggiunta al confronto (Frigi et al., 2006) (dati non mostrati), essa si colloca in un ramo isolato, come un outlier, dimostrando che non ha contribuito al pool genico di Carloforte.
In conclusione, i nostri dati suggeriscono che sia le barriere culturali e linguistiche sia le barriere geografiche hanno la stessa influenza sull’isolamento e, quindi, sulla struttura genetica di una popolazione. Inoltre, questo lavoro conferma l’eterogeneità genetica della popolazione sarda, già evidenziata da ricerche precedenti. (Gatti, 1990; Walter et al., 1991; Morelli, 1993; Moral et al., 1994; Zavattari et al., 2000; Angius et al., 2001; Calò et al., 2008; Pistis et al., 2009), e l’alto grado di isolamento delle popolazioni analizzate, che, come conseguenza, potrebbero rappresentare un’importante risorsa per il futuro mappaggio di tratti complessi.
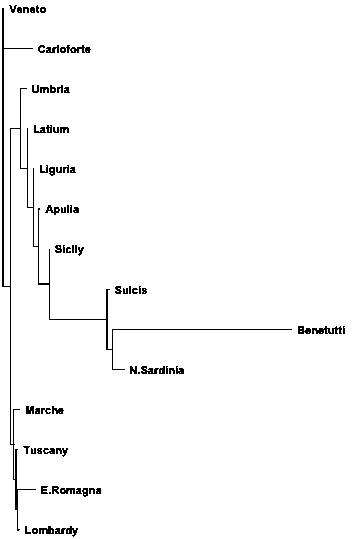
Figura 2. Albero Neighbor-joining costruito mediante il
confronto tra Carloforte, Benetutti e altre popolazioni Italiane (7 Y-STRs).
Figure 2.Neighbor-joining tree built with the Carloforte and
Benetutti samples and Italian populations (7 Y-STRs).
Bibliografia
Alves, V., Gomes, M.J., Prata, A., Amorim, L., Gusmao,
2007, Population data for Y-chromosome haplotypes defined by 17 STRs (AmpFlSTR
YFiler) in Portugal, Forensic Sci Int, 171, 250-255.
Angius, P.M., Melis, L., Morelli, E., Petretto, G., Casu,
G.B., Maestrale, C., Fraumene, D., Bebbere, P., Forabosco, M., Pirastu, 2001,
Archival, demographic and genetic studies define a Sardinian sub-isolate as a
suitable model for mapping complex traits. Hum Genet, 109, 198-209.
Calò, C.M., Melis, A., Vona, G., Piras, I.S., 2008,
Sardinian population (Italy) a Genetic Review. Int. J. Modern Anthropology, 1,
39-64.
Cappello, N., Rendine, S., Griffo, R., Mameli, G.E., Succa,
V., Vona, G., Piazza, A., 1996, Genetic analysis of Sardinia: I. data on 12
polymorphisms in 21 linguistic domains. Ann. Hum. Genet, 60, 125-141.
Colonna, V., Nutile, T., Astore, M., Guardiola, O.,
Antoniol, G., Ciullo, M., Persico, M.G., Campora, 2007, A young genetic isolate
in South Italy. Hum Hered, 64, 123-135.
Di Gaetano, N., Cerutti, F., Crobu, C., Robino, S.,
Inturri, S., Gino, S., Guarrera, P.A., Underhill, R.J., King, V., Romano, F.,
Cali, M., Gasparini, G., Matullo, A., Salerno, C., Torre, A., Piazza,
2009, Differential Greek and northern African migrations to Sicily are
supported by genetic evidence from the Y chromosome. Eur. J. Hum. Genet, 17,
91-99.
Ferri, G., Alù, M., Corradini, B., Radheshi, E., Beduschi,
G., 2009, Slow and fast evolving markers typing in Modena males (North Italy),
Forensic Sci. Int. Genet, 3, 3: e31-e33.
Francalacci, P., Morelli, L., Underhill, P.A., Lillie,
A.S., Passarino, G., Useli, A., Madeddu, R., Paoli, G., Tofanelli, S., Calò,
C.M., Ghiani, M.E., Varesi, L., Memmi, M., Vona, G., Lin, A.A., Oefner, P.,
Cavalli-Sforza, L.L., 2003, Peopling of three Mediterranean islands (Corsica,
Sardinia, and Sicily) inferred by Y-chromosome biallelic variabilità. Am. J.
Phys. Anthrop, 121, 270-279.
Frigi, S., Pereira, P., Pereira, L., Yacoubi, B., Gusma˜o,
L., Alves, C., Khodjet el Khil, H., L., Cherni, A., Amorim, A., El Gaaied,
2006, Data for Y-chromosome haplotypes defined by 17 STRs (AmpFLSTR1 YfilerTM)
in two Tunisian Berber communities. Forensic Sci. Int, 160, 80–83.
Gatti, A.M., 1990,
L'area di scambi matrimoniali in Sardegna tra il XVII e XX secolo. In AA. VV.:
Famiglia e matrimonio nella Societa' Sarda Tradizionale, Nuoro: La tarantola
Edizioni, pp 170-191.
Ghiani, M.E.,
Mameli, A., Piras, G., Berti, A.,
Calò, C.M., Vona, G., 2009, Population Data for Y-chromosome haplotypes defined
by AmpFISTR YFiler PCR Amplification kit in North Sardinia (Italy), Coll.
Antropol, 33, 643-651.
Haplogroup predictor. http://www.hprg.com/hapest5/
Jorde, L. B., Watkins, W. S., Bamshad, M. J., Dixon, M. E.,
Ricker, C. E., Seielstad, M. T., Batzer, M. A., 2000, The Distribution of Human
Genetic Diversity: A Comparison of Mitochondrial, Autosomal, and Y-Chromosome
Data. Am. J. Hum. Genet. 66:979–988.
Kosoy, R., Ransom,
M., Chen, H., Marconi, M., Macciardi,
F., Glorioso, N., Gregersen,
P.K., Cusi, D., Seldin, M.F., 2011, Evidence for malaria selection of a CR1
haplotype in Sardinia. Genes Immun, 12(7):582-8. doi: 10.1038/gene.2011.33.
Moral, P., Marogna, G., Salis, M., Succa, V., Vona, G.,
1994, Genetic data on Alghero population (Sardinia): contrast between
biological and cultural evidence. Am. J. Phys. Anthropol, 93(4): 441-453.
Morelli, L., Vona, G., 1993, Studio genetico della
popolazione di Villanova Monteleone (Sardegna). Nota I. Struttura matrimoniale.
Rend. Semin. Fac. Sci. Univ. Cagliari, 63: 75-86.
Myres, N.M., Ekins, J.E., Lin, A.A., Cavalli-Sforza, L.L., Woodward, S.R., Underhill, P.A., 2007,
Y-chromosome short tandem repeat DYS458.2 non-consensus alleles occur independently
in both binary haplogroups J1-M267 and R1b3-M405. Croat Med J, 48(4):450-9.
Onofri, V., Alessandrini, F., Turchi, C., Fraternale, B.,
Buscemi, L., Pesaresi, M., Tagliabracci, A., 2007, Y-chromosome genetic
structure in sub-Apennine populations of Central Italy by SNP and STR
analysis. Int. J. Legal Med, 121,
234-237.
Pistis, G., Piras, I.,
Pirastu, N., Persico, I., Sassu, A.,
Picciau, A., Prodi, D., Fraumene, C., Mocci, E., Manias, M.T.,
Atzeni, R., Cosso, M., Pirastu, M., Angius, A., 1991, High differentiation among eight villages in
a secluded area of Sardinia revealed by genome-wide high density SNPs analysis.
PLoS One, 4, e4654.
Presciuttini, S., Caglià, A., Alù, M., Asmundo, A.,
Buscemi, L., Caenazzo, L., Carnevali, E., Carra, E., Battisti, Z., De Stefano,
De F., Domenici, R., Piccinini, A., Resta, N., Ricci, U., Pascali, V.L., 2001,
Y-chromosome haplotypes in Italy: the GEFI collaborative database. For. Sc.
Int. 122, 184-188.
Redd, A.J., Agellon, A.B., Kearney, V.A., Contreras, V.A.,
Karafet, T., Park, H., de Knijff, P.,
Butler, J.M., Hammer, M.F., 2002,
Forensic value of 14 novel STRs on the human Y chromosome. Forensic Sci Int,
4;130(2-3):97-111.
Rootsi, S., Magri, C., Kivisild, T., Benuzzi, G., Help, H.,
Bermisheva, M., Kutuev, I., Barać, L., PericˇIc´, M., Balanovsky, O.,
Pshenichnov, A., Dion, D., Grobei, M., Zhivotovsky, L.A., Battaglia, V.,
Achilli, A., Al-Zahery, N., Parik, J., King, R., Cinniog˘Lu, C.,
Khusnutdinova, E., Rudan, P., Balanovska, E., Scheffrahn, W., Simonescu, M.,
Brehm, A., Goncalves, R., Rosa, A., Moisan, J. P., Chaventre A., Ferak, V.,
Furedi, S., Oefner, P.J., Shen, P., Beckman, L., Mikerezi, I., Terzic, R.,
Primorac, D., Cambon-Thomsen, A., Krumina, A., Torroni, A., Underhill, P.A.,
Santachiara-Benerecetti, A.S., Villems, R., Semino, O, 2004, Phylogeography of
Y-Chromosome Haplogroup I Reveals Distinct Domains of Prehistoric Gene Flow in
Europe. Am. J. Hum. Genet. 75:128–137
Scozzari, R., Cruciani, F., Pangrazio, A., Santolamazza,
P., Vona, G., Moral, P., Latini, V., Varesi, L., Memmi, M.M., Romano, V., De
Leo, G., Gennarelli, M., Jaruzelska, J., Villems, R., Parik, J., Macaulay, V.,
Torroni, A, 2001, Human Y-chromosome variation in the western Mediterranean
area: implications for the peopling of the region. Hum. Immunol.
62:871–884.
Semino O, Passarino G, Oefner PJ, Lin AA, Arbuzova S,
Beckman LE, De Benedictis G, Francalacci P, Kouvatsi A, Limborska S, Marcikiae
M, Mika A, Mika B,Primorac D, Santachiara-Benerecetti AS, Cavalli-Sforza LL,
Underhill PA, 2000, The genetic legacy of Paleolithic Homo sapiens sapiens in
extant Europeans: a Y chromosome perspective. Science, 10;290(5494):1155-9.
Slatkin, M., 1995, A Measure of Population Subdivision
Based on Microsatellite Allele Frequencies. Genetics, 139(1): 457–462.
Vallebona, G., 1974, Carloforte. Storia di una
colonizzazione (Cagliari: Edizioni della Torre).
Walter, H., Matsumoto, H., De Stefano, G.F., 1991, Gm and
Km allotypes in four Sardinian population samples. Am. J. Phys. Anthrop, 86,
45-50.
Zavattari, P., Deidda,
E., Whalen, M., Lampis, R., Mulargia, A., Loddo, M., Eaves,
I., Mastio, G., Todd, J.A., Cucca, F., 2000,
Major factors influencing linkage disequilibrium by analysis of
different chromosome regions in distinct populations: demography, chromosome
recombination frequency and selection. Hum. Mol. Gen, 9, 2947-2957.