
Dumas, F., 2012, Riarrangiamenti cromosomici: i nuovi centromeri evolutivi nei
primati. Antropo,
28, 1-7. www.didac.ehu.es/antropo
Riarrangiamenti
cromosomici: i nuovi centromeri evolutivi nei primati
Chromosomal rearrangements: evolutionary new centromeres in primates
Francesca Dumas
Dipartimento di Biologia ambientale e biodiversità, sezione
di Biologia animale ed Antropologia biologica, Università degli Studi di
Palermo, via Archirafi 18. Palermo, Italy
e-mail address: francesca.dumas@unipa.it
Parole chiave: cromosomi, riposizionamento centromeri, mapppaggio di BACs, citogenetica molecolare
Keywords: chromosomes, centromere
repositioning, BACs mapping, molecular cytogenetics
Abstract
Molecular
cytogenetics by mapping cloned DNA fragments such as BACs (Bacterial Artificial
Chromosomes) on primates metaphases permitted researchers to show that the centromeres
location is not conserved as previously thought. The analysis of data present
in literature shows that new evolutionary centromeres (centromeres shifts) are
not rare and have to be considered as other traditional chromosomal
rearrangements (fission, fusion and inversion) as responsible of chromosomal
evolutions. The comparison between neocentromere in humans and evolutionary new
centromeres in primates permits to define their relationship in clinics and
evolution.
Riassunto
La citogenetica
molecolare mediante il mappaggio di sonde di DNA clonato in BACs (Bacterial
Artificial Chromosomes) su metafasi di Primati ha permesso di dimostrare che la
posizione dei centromeri non è
conservata come in precedenza ipotizzato. L’analisi dei dati presenti in
letteratura dimostra che la formazione di un nuovo centromero evolutivo (o
“shift” del centromero) non è un evento raro e deve essere considerato alla
stregua degli altri riarrangiamenti (fissione, fusione e inversione) come
responsabile dell’evoluzione cromosomica. Il confronto tra i neocentromeri nell’uomo e
i nuovi centromeri evolutivi nei primati permette di definire il loro rapporto
nei casi clinici e nell'evoluzione.
Introduzione
Tutti
i cromosomi hanno in una certa posizione una costrizione chiamata centromero,
composta da blocchi di DNA satellite, responsabile della segregazione dei
cromosomi e cromatidi durante la mitosi e meiosi. Tradizionalmente la posizione
dei centromeri è stata considerata altamente conservata, recentemente mediante
la citogenetica molecolare è stato dimostrato il contrario. Questo approccio ha
permesso di enfatizzare il ruolo delle fissioni, fusioni di Robertson, così
come delle inversioni pericentriche, negli eventi di trasformazione delle
specie. In relazione a queste trasformazioni è stato attribuito al centromero
un ruolo fondamentale nei cambiamenti cromosomici sia nel corso dell'evoluzione
sia in campo clinico (Villasante et al., 2007).
I
nuovi centromeri evolutivi e i neocentromeri nell’uomo emergono per epigenesi
(non come conseguenza di una sequenza specifica) in “hot spot” cromosomici; i
primi sono centromeri che si spostano (“shift”) lungo il cromosoma durante
l’evoluzione genomica senza alcuna variazione dell’ordine dei marcatori (senza
l’intervento di inversioni o di altri riarrangiamenti strutturali) Fig. 1; i
secondi sono centromeri che emergono, nei cromosomi umani, in regioni
cromosomiche ectopiche, prive di
sequenze alfoide, cioè di DNA satellite presente nei centromeri dei primati. Il
dominio in cui si raggruppano i neocentromeri spesso corrisponde a centromeri
ancestrali disattivati, per questo motivo i nuovi centromeri evolutivi e i neocentromeri possono essere considerati
come le due facce dello stesso fenomeno (Capozzi et al., 2008, Rocchi et al.,
2009).
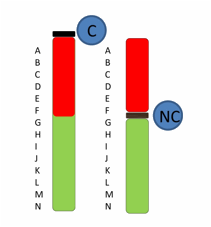
Figura 1. Nuovi centromeri evolutivi (NC): riposizionamento del centromero in
una regione cromosomica diversa senza alcun cambiamento nell’ordine dei
marcatori (es. senza intervento di inversioni)
Figure
1. Evolutionarily new
centromeres (NC). Evolutionarily New centromeres (centromere shifts) are
centromeres found in a different genomic context without a change in marker
order (i.e.no inversion)
Prospettiva storica sui nuovi centromeri evolutivi nei Primati
La
diversa posizione del centromero lungo un cromosoma è stata quasi sempre
interpretata come il risultato di una inversione pericentrica o di
riarrangiamenti complessi. Tuttavia, in uno studio sull'evoluzione dei
cromosomi umani mediante la citogenetica classica (bandeggio dei cromosomi) in
60 specie di primati è stato definito, per la prima volta, il concetto di
”traslocazione del centromero” come possibile meccanismo dell'evoluzione del
cromosoma 11 in alcuni Cercopithecidae (Dutrillaux, 1979). Si è inoltre
ipotizzato che in gruppi caratterizzati da un elevato numero diploide, a causa
del verificarsi di fissioni non centromeriche, si acquisiscono nuovi
centromeri. Più tardi, Clemente e colleghi (1990) ipotizzarono, studiando
l’evoluzione dei cromosomi umani, che la differente posizione del centromero
negli omologhi dei cromosomi umani 4, 6 e 10 non fosse risultato di inversioni,
ma derivasse dall'attivazione ed
inattivazione di nuovi centromeri.
L’avvento
del painting comparativo di cromosomi o zoo-FISH (fluorescent in-situ
hybridization) ha permesso la comparazione di genomi poco studiati, delineando
l'omologia cromosomica a livello di DNA ed identificando le sintenie genomiche
e i riarrangiamenti intercromosomici (Wienberg et al., 1990). Tale approccio ha
permesso di acquisire una considerevole quantità di dati sui riarrangiamenti
inter-cromosomici (traslocazioni, fissioni e fusioni) nei mammiferi, fornendo solo limitate informazioni sull'orientamento
delle sintenie e sui riarrangiamenti intra-cromosomici come inversioni, duplicazioni
e sul riposizionamento dei centromeri (Dumas et al., 2007). Tale limite viene superato mediante il
mappaggio di sonde sub-cromosomiche, locus specifiche e di DNA clonato
all’interno di vettori (Romagno et al., 2000, Ventura et al., 2004, Dumas e
Sineo 2010).
La rivelazione dei nuovi centromeri evolutivi: il mappaggio di sonde di
DNA clonato
Sonde
molecolari costituite da inserti di DNA in vettori come fagi, cosmidi, YAC
(Yeat Artificial Chromosomes) e BACs (Bacterial Artificial Chromosomes) sono usate
per identificare accuratamente i riarrangiamenti intra-cromosomici, i punti di
rottura cromosomici (Muller et al., 2000), l'ordine dei marcatori nelle
sintenie cromosomiche e i nuovi centromeri evolutivi (ENC) (Stanyon et al., 2008)
Fig.2.
I
nuovi centromeri evolutivi (ENC), come i neocentromeri, sono stati trovati in
una nuova regione cromosomica funzionale senza che ciò influenzi in alcun modo
l'ordine dei marcatori (es. BACs) e la loro formazione risulta essere sempre
accompagnata dall'inattivazione di un vecchio centromero anche se è stato
ipotizzato che per un certo tempo il
vecchio e il nuovo centromero possono coesistere come polimorfismi.
Montefalcone
et al. (1999) fu il primo a dimostrare in modo inequivocabile mediante
FISH con sonde di DNA clonato
l'esistenza del fenomeno del riposizionamento del centromero tracciando la
storia evolutiva e filogenetica del cromosoma IX nei primati.
Da quel momento in poi
nuovi centromeri evolutivi sono stati dimostrati mediante questo approccio in molti
primati e in altri mammiferi (Ferreri et a., 2005, Cardone et al., 2006,
Carbone et al., 2006, Kobayashi et al., 2008, Wade et al., 2009).
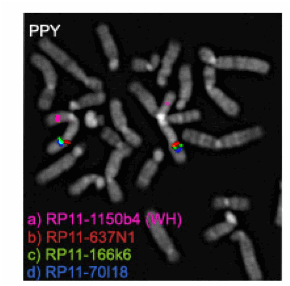
Figura 2. Ibridazione (FISH) con sonde di DNA clonato; in questo caso sonde
BACs su metafasi di Pongo pygmaeus (PPY)
Figure
2. Hybridization (FISH) with
cloned DNA; in this case BACs (Bacterial artificial chromosomes) on Pongo pygmaeus (PPY) metaphases
Nuovi centromeri evolutivi (ENC) nei
Primati
Molti nuovi
centromeri evolutivi sono stati individuati studiando la storia evolutiva dei
cromosomi umani nei primati (Stanyon e Bigoni, 2010, Rocchi et al., 2012): 3 (Ventura
et al., 2004), 6 ( Edere t al.,2003), 10 (Carbone et al., 2002), 11 (Cardone et
al, 2007), 13 (Cardone et al., 2006), 14,15 (Ventura et al., 2003), 20 (Misceo
et al., 2005), X (ventura et al., 2001).
Altri nuovi centromeri evolutivi sono stati identificati mediante il
mappaggio di sonde BACs al fine definire l’ordine dei marcatori all’interno di
specifici cromosomi nei primati (Stanyon et al., 2008, Rocchi et al., 2012).
Il
caso più noto riguarda il cromosoma 2 dell’uomo che deriva dalla fusione di due
cromosomi acrocentrici e dalla disattivazione di un centromero in posizione
2q21.1 (Yunish and Prakash 1982, Jauch et al. 1992, Avarello et al., 1992).
Particolare
attenzione merita la sintenia cromosomica X in quanto nei mammiferi risulta uno dei cromosomi più
conservati (Chowdhary et al., 1998) eccetto in alcune proscimmie (Ventura et
al., 2001); in questo contesto risultò strana l’ipotesi di Schempp et al. (1989)
secondo cui il cromosoma X avesse subito qualche complesso riarrangiamento
intracromosomico in Saimiri sciureus. Successivamente Dumas e colleghi
(2007) ipotizzarono che il cromosoma X di Saimiri sciureus avesse subito
o una inversione pericentrica o un riposizionamento del centromero. Rocchi e
colleghi (2012) mediante il mappaggio di sonde BACs dimostrano l’occorrenza di
un’inversione in concomitanza di uno “shift” del centromero nel cromosoma X di Saimiri
sciureus.
Ventura
e colleghi (2007) confrontando i cromosomi umani e di Macaca hanno
dimostrato che il 50% dei cromosomi di macaca presenta nuovi centromeri
evolutivi. In totale in macaca e uomo sono presenti 14 nuovi centromeri,
nove nella scimmia del vecchio mondo e cinque in Homo, tutti costituiti
da DNA satellite e duplicazioni pericentromeriche (presenti normalmente nei
centromeri funzionali).
Persino
a livello di popolazione è stato dimostrato, di recente, un polimorfismo sul
cromosoma 9 di Orango frutto di un nuovo centromero evolutivo (Rocchi et
al., 2012).
Relazione tra i Nuovi Centromeri Evolutivi e i Neocentromeri
In
ambito clinico sono stati descritti più di 100 casi di neocentromeri (Marshall
et al. 2008) la cui apparizione è
generalmente un evento secondario e opportunistico, concomitante al
riarrangiamento che ha generato un frammento acrocentrico.
I
neocentromeri sono centromeri ectopici, che si possono formare spontaneamente
in regioni eucromatiniche del genoma umano prive di sequenze ripetute. La
migrazione dei centromeri e l'esistenza dei neocentromeri umani al di fuori del
DNA alfa-satellite sono un'evidenza del fatto che i centromeri sono determinati
da un meccanismo epigenetico piuttosto che sequenza-specifico. I neocentromeri
clinici tendono a raggrupparsi in "punti caldi" in determinati
cromosomi come per esempio sul 3q, 8p, 13q, e 15q particolarmente proni a
formare neocentromeri (Marshall et al., 2008, Ventura et al., 2004).
La conoscenza degli ENC,
fornisce una buona spiegazione per l'inusuale “clustering” dei neocentromeri; lo
studio della storia dei cromosomi umani ha permesso di identificare i nuovi
centromeri evolutivi e nel caso dei cromosomi 3, 13 e 15 è stata dimostrata
l’esistenza di una stretta correlazione tra la posizione dei nuovi centromeri
evolutivi e i neocentromeri. In particolare nel caso del cromosoma 3 i
neocentromeri si formano in un’area dove un nuovo centromero evolutivo si è
formato nelle Catarrhinae (3q26). Lo stesso dicasi per il cromosoma 13 dove i
neocentromeri si formano in corrispondenza del nuovo centromero evolutivo
formatosi nel braccio lungo dell’omologo del cromosoma 13 nelle Catarrhinae
(13q12). Nel caso del cromosoma 15, in posizione q24-26, dove si riscontrano
tutti i casi di neocentromeri clinici è presente la regione dominio di un
centromero ancestrale inattivato (Fig. 3).
Un caso di nuovo centromero
funzionale è stato identificato durante uno “screening prenatale” in 6
individui di una famiglia, nel corso di tre generazioni, in posizione 6p22. Lo
studio della storia evolutiva del cromosoma 6 ha permesso di dimostrare che 17
milioni di anni fa, il dominio 6p22, fosse la normale posizione del centromero
(Capozzi et al., 2009).
Questi studi dimostrano
che i nuovi centromeri evolutivi non
sono rari e devono essere considerati alla stregua dei riarrangiamenti
tradizionali (inversioni, traslocazioni, delezioni ed inserzioni) come
meccanismi responsabili dell’evoluzione cromosomica.
La citogenetica
molecolare fornisce gli strumenti necessari per l’identificazione dei nuovi
centromeri evolutivi non individuabili mediante il sequenziamento “shot-gun”di
DNA (Rocchi et al., 2009). Infatti le tecniche di sequenziamento presentano dei
limiti, nell’assemblaggio nei cromosomi delle sequenze e nell’identificare i
centromeri; tale limite può essere superato solo attraverso l’implementazione
dei metodi della citogenetica.
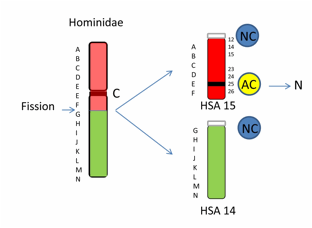
Figura 3. Storia evolutiva dei
cromosomi 14 e 15 dell’uomo mediante il mappaggio di sonde BAC (marcatori
rappresentati con le lettere A-N) e relazione tra neocentromeri e nuovi centromeri
evolutivi. I cromosomi 14 e 15 derivano
dalla fissione di un cromosoma ancestrale presente nel comune antenato degli Hominidae
(umani e grandi scimmie). Il mappaggio delle sonde BAC indica che l’ordine dei
marcatori è conservato in macaca e nei due cromosomi umani. La
fissione è avvenuta nella regione tra il marcatore F e G (Ventura et al., 2003).
Si formano due nuovi centromeri: uno nella regione telomerica del cromosoma 15,
l’altro in corrispondenza del punto di fissione sul cromosoma 14. Il centromero
ancestrale mappa in corrispondenza dell’apparente suddivisione del marcatore E.
In corrispondenza del centromero ancestrale 15q24-26 si riscontrano i
neocentromeri umani. C= centromero; AC=centromero ancestrale; NC= nuovo
centromero; N=Neocentromeri clinici
Figure 3. Evolutionary history of human chromosomes 14 and 15 by mapping BAC
probes (markers A-N) and relationship between neocentromere and new
evolutionary centromeres. Human chromosomes 15 and 14 derive from the fission
of an ancestral chromosome in the hominidae ancestor (humans and great apes).
The mapping of the BACs probes shows that the marker order is perfectly
conserved in Macaca and on the two
human chromosomes. The fission occurred in the region between markers F and G
(Ventura et al., 2003). Two novel centromeres emerged: one on the telomeric
region of chromosome 15 and the second in correspondence to the fission point
on the chromosome 14. The ancestral chromosome is signed by the apparent split of marker E. Human
neocentromeres cluster overlapping the ancestral centromeres (15q24-26). C=centromeres; AC=
ancestral centromeres; NC= new centromeres; N=clinical neocentromere.
Bibliografia
Avarello , R., Pedicini,
A., Caiulo, A., Zuffardi, O., Fraccaro M., 1992, Evidence for an ancestral
alphoid domain on the long arm of human chromosome 2. Human genet, 89; (2)
335-339.
Capozzi, O., Purgato, S., D’Addabbo, P.,
Archidiacono, N., Battaglia, P., Baroncini, A. Capucci A e Stanyon, R., 2009,
Evolutionary descent of a human chromosome 6 neocentromere: a jump back to 17
million years ago. Genome Res 19: 778–784.
Capozzi, O., Purgato, S., Verdun di Cantogno, L., Grosso, E.,
Ciccone, R., Zuffardi, O. della Valle G., e Rocchi, M., 2008, Evolutionary and clinical
neocentromeres: two faces of the same coin? Chromosoma 117: 339–344.
Carbone, L., Ventura, M., Tempesta, S., Rocchi, M., Archidiacono,
N. Evolutionary history of chromosome 10 in primates. Chromosoma (2002)
111:267–272
Carbone, L., Nergadze, SG., Magnani, E., Misceo, D., Francesca
Cardone, M, Roberto, R, Bertoni, Attolini C, Francesca Piras M., de jong P.,
Raudsepp T., Chowdhary BP, Guerin G., Archidiacono, N., Rocchi, M., e Giulotto
E., 2006, Evolutionary movement of centromeres in horse, donkey, and zebra. Genomics 87: 777–782.
Cardone, MF., Alonso, A., Pazienza M.,
Ventura, M., Montemurro, G., Carbone, L., de jong P, Stanyon, R., D’Addabbo, P.,
Archidiacono, N., She, X., Eichler EE, Warburton PE, Rocchi, M., 2006,
Independent centromere formation in a capricious, gene-free domain of
chromosome 13q21 in old world monkeys and pigs. Genome Biol 7: R91.
Cardone, MF., Lomiento, M., Teti, MG.,
Misceo, D., Roberto, R., Capozzi, O., D’Addabbo, P., Ventura, M., Rocchi, M., Archidiacono, N., 2007,
Evolutionary history of chromosome 11 featuring four distinct centromere repositioning events in Catarrhini. Genomics
90:35-43.
Chowdhary, BP., Raudsepp, T., Fronicke,
L., e Scherthan, H., 1998, Emerging patterns of comparative genome organization in some
mammalian species as revealed by Zoo- FISH. Genome Res 8: 577–589.
Clemente, IC., Ponsa, M., Garcia, M., e Egozcue,
J., 1990, Evolution of the Simiiformes and the phylogeny of human chromosomes.
Hum Genet 84: 493–506.
Dumas, F., Stanyon, R.,
Sineo, L., Stone ,G., e Bigoni, F., 2007, Phylogenomics of species from four
genera of new world monkeys by flow sorting and reciprocal chromosome painting.
BMC Evol Biol 7(Suppl 2): S11
Dumas, F., Sineo, L., 2010,
Chromosomal dinamycs in Cercopithecini studied by Williams Beuren probe
mapping. Caryologia 3, 4:435-442.
Dutrillaux, B., 1979, Chromosomal
evolution in primates: tentative phylogeny from Microcebus murinus (Prosimian)
to man. Hum Genet 48: 251–314.
Eder, V., Ventura, M., Ianigro, M., Teti,
M., Rocchi, M., e Archidiacono, N., 2003, Chromosome 6 phylogeny in primates
and centromere repositioning. Mol Biol Evol 20: 1506–1512.
Ferreri, GC., Liscinsky, DM., Mack, JA.,
Eldridge, MD., e O’Neill, RJ., 2005, Retention of latent centromeres in the
mammalian genome. J Hered 96: 217–224.
Kobayashi, T., Yamada,.F, Hashimoto, T.,
Abe, S., Matsuda, Y., e Kuroiwa, A., 2008, Centromere repositioning in the X
chromosome of XO/XO mammals, Ryukyu spiny rat. Chromosome Res 16:
587–593.
Jauch, A., Wienberg, J., Stanyon, R.,
Arnold, N., Tofanelli, S., Ishida, T., e Cremer, T., 1992, Reconstruction of
genomic rearrangements in great apes and gibbons by chromosome painting. Proc.
Natl. Acad. Sci. 89: 8611–8615.
Marshall, O.J., Chueh, AC,
Wong, LH., e Choo, KH., 2008, Neocentromeres: new insights into centromere
structure, disease development, and karyotype evolution. Am J Hum Genet, 2008.
82(2): p.261-82.
Misceo D, Cardone MF, Carbone L,
D’Addabbo P, de Jong PJ, Rocchi M., e Archidiacono, N., 2005, Evolutionary
history of chromosome 20. Mol Biol Evol 22: 360–366.
Montefalcone, G., Tempesta, S., Rocchi, M., e Archidiacono, N., 1999,
Centromere repositioning. Genome Res 9: 1184–1188.
Rocchi, M., Stanyon, R. e Archidiacono, N., 2009, Evolutionary
new Centromere in Primates. Prog Mol Subcell Biol.;48:103-52.
Rocchi, M., Archidiacono, N., Schempp, W., Capozzi, O.,e Stanyon,
R., 2012. Centromere repositioning in mammals.
Heredity 108: 59-67.
Romagno, D., Chiarelli, B., Guarducci, S.,
Giovannucci-Uzielli, ML., e Sineo, L., 2000, chromosome mapping of GABRB3 and
PML loci in Macaca and Cercopithecus indicates the mechanism of evolution of human
chromosome 15. Chromosome Res 8:747–749.
Schempp, W., Weber, B., e Muller, G.,
1989, Mammalian sex-chromosome evolution: a conserved homoeologous segment on
the X and Y chromosomes in primates. Cytogenet Cell Genet 50: 201–205.
Stanyon, R., e Bigoni, F., 2010, Primate
chromosome evolution: with reference to marker order and Neocentromeres. Russian
journal of genetics 46: 1087-1093.
Stanyon, R, Rocchi, M, Capozzi, O,
Roberto, R, Misceo, D, Ventur,a M, Cardone, MF, Bigoni, F e Archidiacono, N,
2008, Primate chromosome evolution: Ancestral karyotypes, marker order and
neocentromeres. Chromosome Research, 16:17-39.
Ventura, M., Antonacci, F., Cardone, MF., Stanyon, R.,
D’Addabbo, P., Cellamare, A., Sprague, LJ., Eichler, EE., Archidiacono,
N.,
e Rocchi, M, 2007, Evolutionary formation of new centromeres in macaque. Science 316:
243–246.
Ventura, M., Archidiacono, N., e Rocchi, M., 2001, Centromere
emergence in evolution. Genome Res 11: 595–599.
Ventura M, Mudge JM, Palumbo V, Burn S, Blennow E, Pierluigi M,
Giorda, R., Zuffardi, O., Archidiacono, N., Jackoson, MS., e Rocchi M., 2003. Neocentromeres in 15q24-26 map to duplicons which
flanked an ancestral centromeres in 15q25. Genome Res
13: 2059–2068.
Ventura,
M., Weigl, S., Carbone, L., Cardone, MF., Misceo, D., Teti, M. D'Addabbo, P., Wandall, A., Björck, E., de Jong, PJ., She, X., Eichler ,EE., Archidiacono,
N., e Rocchi, M., 2004, Recurrent sites for new centromere seeding. Genome Res 14:
1696–1703.
Villasante, A., J.P. Abad, e
Mendez-Lago, M., 2007, Centromeres were derived from telomeres during the
evolution of the eukaryotic chromosome. Proc Natl Acad Sci U S A,. 104(25): p.
10542-7.
Wade, CM., Giulotto, E.,
Sigurdsson, S., Zoli, M., Gnerre, S., Imsland, F., Lear, TL., Adelson, DL.,
Bailey ,E., Bellone, RR., Blöcke,r H., Distl, O., Edgar, RC., Garber, M., Leeb,
T., Mauceli, E., MacLeod, JN., Penedo, MC., Raison, JM., Sharpe, T., Vogel, J.,
Andersson, L., Antczak, DF., Biagi, T., Binns, MM., Chowdhary, BP., Colema,n SJ.,
Della Valle, G., Fryc, S., Guérin, G., Hasegawa, T., Hill, EW., Jurka, J.,
Kiialainen, A., Lindgren, G., Liu, J., Magnani, E., Mickelson, JR., Murray, J.,
Nergadze, SG., Onofrio, R., Pedroni, S., Piras, MF., Raudsepp, T., Rocchi, M.,
Røed, KH., Ryder, OA., Searle, S., Skow, L., Swinburne, JE., Syvänen, AC.,
Tozaki, T., Valberg, SJ., Vaudin, M., White, JR., e Zody, MC., 2009, Genome
sequence, comparative analysis, and population genetics of the domestic horse.
Science 326: 865–867.
Wienberg, J., Jauch, A., Stanyon, R., e Cremer,
T., 1990, Molecular cytotaxonomy of primates by chromosomal in situ suppression
hybridization. Genomics 8: 347–350.
Yunis, J.J. and Prakash, O., 1982, The
origin of man: A chromosomal pictorial legacy. Science 215: 1525–1530.