
Dumas, F., 2011, Studio sull’origine e l’evoluzione del cromosoma 4 dell’uomo
mediante la citogenetica molecolare comparata. Antropo, 25,
43-64. www.didac.ehu.es/antropo
Studio sull’origine e l’evoluzione del
cromosoma 4 dell’uomo mediante la citogenetica molecolare comparata
Study on the origin and evolution
of the human chromosome 4 through comparative molecular cytogenetics
F. Dumas
Dipartimento di
Biologia Ambientale e biodiversità, sezione Biologia Animale ed Antropologia biologica
“G. Reverberi”, via Archirafi 18, 90123
Palermo, Italy. fdumas@unipa.it
Parole chiave:
sintenia umana 4, painting cromosomico, mammiferi, primati
Keywords: human synteny 4,
chromosome painting, mammals, primates
Riassunto
La citogenetica molecolare permette uno studio accurato sull'evoluzione
e sull'origine
dei cromosomi umani. Il
painting comparativo di cromosomi o zoo-FISH (Fluorescent In-Situ
Hybridization) è un efficace mezzo di comparazione di genomi di specie in
quanto permette di delineare l'omologia cromosomica, di definire la sintenia
genomica e i riarrangiamenti intercromosomici. Il presente lavoro è uno studio
di revisione di dati di painting cromosomico sugli omologhi del cromosoma umano
4 nei mammiferi con particolare attenzione
ai primati al fine di delineare
l’origine e la storia evolutiva del cromosoma umano 4. I dati presenti in
letteratura indicano che la sintenia 4 dell’uomo è in associazione con il braccio
piccolo del cromosoma umano 8p nel cariotipo ancestrale di tutti i mammiferi
euteri. Nel presente lavoro a partire dalla forma ancestrale 4/8p si sono
ricostruite le tappe principali e i
riarrangiamenti intercromosomici che hanno caratterizzato la storia evolutiva
della sintenia 4 lungo ciascuna linea filogenetica dei mammiferi. Questa indagine
ha permesso di dimostrare che la sintenia 4 è meno conservata di quanto si fosse
sempre pensato.
Abstract
Molecular cytogenetics allows
the accurate study of the evolution and origin of human chromosomes. The comparative
chromosome painting or zoo-FISH (Fluorescent In-Situ Hybridization) is
an efficient means of comparing genomes, delineating chromosome homology,
defining genomic synteny and interchromosomal rearrangements. The present work
is a review of comparative chromosome painting data on the homologs to human chromosome
4 throughout the mammals
with special emphasis on primates. The aim is to define the origin and the
history of human chromosome 4. The analyses of
literature show that human synteny 4 is
in association with synteny 8p in the hypothetic ancestral karyotype of all
eutherian mammals. In the present work the principal steps from this
ancestral 4/8p syntenic association are traced along each phylogenetic branch.
This review shows that the synteny is less conserved then frequently thought.
Introduzione
La citogenetica molecolare comparata mediante lo “ZOO-Fluorescence In Situ Hybridization” (ZOO-FISH) che consiste nell’ibridazione di sonde cromosomiche umane e non umane su matafasi di specie target, permette di individuare le omologie cromosomiche e i riarrangiamenti intercromosomici (traslocazioni, fusioni e fissioni) che si verificano nel corso dell’evoluzione genomica (Wienberg et al., 1992). L’applicazione dei principi della scuola cladista e della massima parsimonia, nella comparazione delle omologie cromosomiche, permette di discriminare le sintenie ancestrali dalle associazioni cromosomiche derivate, formatesi a seguito di riarrangiamenti, utili nello studio filogenetico e nella ricostruzione degli ipotetici cariotipi ancestrali (Wienberg and Stanyon, 1995).
Il contenuto dell’ipotetico cariotipo ancestrale dei mammiferi euteri ricostruito mediante la citogenetica molecolare ha la seguente costituzione (AEK, Ancestral Eutherian karyotype, 2n=46): 1, 2p-q, 2q, 3/21, 4/8p, 5, 6, 7a, 7b/16p, 8q, 9, 10q, 10p/12/22, 11, 12/22, 13, 14/15, 16q/19q, 17, 18, 19p, 20, X e Y (Ferguson-Smith and Trifonov, 2007, Robinson and Ruiz-Herrera 2008). A partire dal cariotipo ancestrale di Mammiferi placentati, è possibile, determinare i passi più importanti che negli ultimi 90 milioni di anni hanno portato alla formazione dei cromosomi umani (Stanyon et al., 2008) anche se per molti cromosomi alcune tappe sono ancora oscure.
Il cromosoma umano 4 (HSA 4) è stato poco studiato ed ha ricevuto attenzione da un punto di vista medico, principalmente per le ricerche correlate al gene della malattia di Huntington, per i geni associati alla sindrome di Wolf-Hirschhorn e del gene associato alla distrofia muscolare degenerativa (FSHD) (Walker et al. 2007, Wright et al., 1997, Zollino et al., 2004, Bodega et al., 2007).
Con il nome di cromosoma 4 si indica, per convenzione, il quarto cromosoma umano in ordine di grandezza. Questo largo cromosoma submetacentrico del gruppo B, contiene 186 milioni di coppie di basi, 796 geni codificanti per proteine ed enormi "deserti genetici" (parti del genoma prive di sequenze che codificano per proteine) e regioni molto concentrate (Hillier et al., 2005).
Mediante la genomica comparata è stato dimostrato che il cromosoma 4 è conservato nel cariotipo ancestrale dei Mammiferi euteri (AEK- Ancestral Eutherian Karyotype), in associazione con il braccio piccolo del cromosoma umano 8p (Ferguson-Smith and Trifonov, 2007, Stanyon et al., 2008). Questa associazione ancestrale 4/8p la ritroviamo in tutti i mammiferi eccetto in alcuni Afrotheria e nei Primati. Inoltre la sintenia 4 è andata incontro a diversi riarrangiamenti intercromosomici formando nuovi segmenti e associazioni con altre sintenie umane. A tal proposito per esempio, sono stati dimostrati diversi riarrangiamenti nel cane e nei muridi (Graphodasky et al., 2002, 2008).
Nel presente lavoro sono stati raccolti dati cromosomici presenti in letteratura, ottenuti mediante la citogenetica molecolare, relativi all’omologo del cromosoma 4 dell’uomo, in 176 specie di Mammiferi Euteri appartenenti a 18 ordini al fine di chiarirne la storia evolutiva (Tab.1). L’analisi dei dati raccolti ha permesso di individuare le tappe principali che hanno caratterizzano la storia di questo cromosoma nel corso dell’evoluzione genomica dei Mammiferi con particolare riguardo ai Primati.
I
dati relativi all’omologo del cromosoma 4 umano nei mammiferi placentati sono stati
raccolti per i super ordini: Afrotheria (Tubulidentata, Macroscelidea,
Afrosoricidae, Sirenia, Hyracoidea, Proboscidea) che comprende i lamantini,
tenrecs, oritteropi, toporagni elefante, iraci, elefanti; Xanarthra (Pilosa e
Cingulata) con i bradipi, formichieri e armadilli; Laurasiatheria (Eulipotyphla,
Carnivora, Pholidota, Cetartiodactyla, Perissodactyla, Chiroptera) con gli
insettivori, carnivori, pangolini, cetacei, artiodattili, perissodattili, pipistrelli; Euarchontoglires (Primates, Dermoptera, Scandentia, Lagomorpha,
Rodentia) con i primati, lemuri volanti, tupaia, lepri e conigli e roditori. In
particolare sono stati analizzate informazioni riguardanti i Tubulidentata
(Yang et al., 2003) i Macroscelidea (Robinson et al., 2004, Svartman et al., 2004), gli Afrosoricidae
(Robinson et
al., 2004; Gilbert et al., 2006), i Sirenia (Kellogg et al., 2007), i Proboscidea
(Yang et al., 2003), gli Hyracoidea (Pardini et al., 2007), i Pilosa (Yang et
al., 2006; Svartman et al., 2006), i Ciungulata
(Svartman et al., 2006), gli Eulipotyphla
(Dixkens et al., 1998, Volleth and Muller 2006, Ye et al., 2006, Yang et al.,
2006), i Carnivora (Nash et al., 1998; Froenicke et al., 1997; Wienberg et al.,
1997; Yang et al, 1999, 2000; Hameister, et al., 1997; Nie et al., 2002; Perelman et al.,
2008; Cavagna et al., 2000, Graphodatsky et al., 2000, 2001, 2002 Tian et al.,
2004), i Pholidota (Yang et al., 2006, Nie et al., 2009), i Cetartiodactyla
(Kulemzina et al., 2009; Froenicke e Wienberg, 2001; Chaves et al., 2004;
Bielec et al.,1998; Kulemzina et al., 2009, Balmus et al., 2007, Huang et al.,
2008), i Perissodactyla (Richard et al., 2001, Trifonov et al., 2008; Yang et
al., 2004), i Chiroptera (Volleth et
al., 2002, 1999; Mao et al. 2007, 2008), i Dermoptera (Nie et al., 2008), gli Scandentia (Muller et al.,
1999), i Lagomorpha (Korstanje et al., 1999, Hayes et al., 2002) e i Rodentia (Graphodatsky et al., 2008; Li et al., 2004;
Stanyon et al., 2003, 2004, Richard et al., 2003).
Relativamente all’ordine dei Primati, sono stati considerati dati riguardanti le Proscimmie (Cardone et al., 2002; Muller et al., 1997; Richard et al., 2000, Stanyon et al., 2002; Stanyon et al., 2006, Nie et al., 2006; Warter et al., 2005, Rumpler et al., 2008), le Platyrrhinae (Morescalchi et al., 1997; Neusser et al., 2001; Muller et al., 2001, Garcia et al., 2002; Richard et al., 1996; Barros et al., 2003, De Oliveira et al., 2005, 2002, Consigliere et al., 1996, 1998; Stanyon et al., 2000, 2001, 2003, 2004; Serreau et al., 2004, Dumas et al., 2005, 2007, Ruiz-Herrera et al., 2005; Amaral et al., 2008, Finotelo et al., 2010) e le Catarrhinae (Wienberg et al., 1992; Ruiz-Herrera et al., 2002; Bigoni et al., 1997a, 1997b, 2003, 2004; Finelli et al., 1999; Nie et al.,1998; Stanyon et al., 2005, Moulin et al., 2008; Muller e Wienberg 2001, Nie et al., 2001, Koehler et al., 1995; Muller et al., 2003; Jauch et al., 1992).
|
Ordine |
Specie |
Riferimento bibliografico |
|
TUBELIDENTATA |
Orycteropus afer |
Robinson et al., 2004 |
|
MACROSCELIDA |
Elephantulus rupestris |
Robinson et al., 2004 |
|
Macroscidelis proboscidens |
Svartman et al., 2004 |
|
|
SIRENIA |
Trichechus manatus |
Kellogg et al., 2007 |
|
PROBOSCIDEA |
Loxodonta africana |
Yang et al., 2003 |
|
Elephas maximum |
Yang et al., 2003 |
|
|
HYRACOIDEA |
Procavia capensis |
Pardini et al., 2007 |
|
AFROSORICIDA |
Chrysochloris asiaticus |
Robinson et al., 2004 |
|
Amblysomus hottentotus |
Gilbert et al., 2006 |
|
|
Chrysospalax trevelyani |
Gilbert et al., 2006 |
|
|
Cryptochloris zyli |
Gilbert et al., 2006 |
|
|
Eremitalpa granti |
Gilbert et al., 2006 |
|
|
PILOSA |
Tamandua tetradactyla |
Yang et al.,
2006 Svartman et al.,
2006 |
|
Choloepus didactylus |
Yang et al., 2006 |
|
|
Choloepus hoffmanni |
Svartman et al., 2006 |
|
|
CINGULATA |
Dasypus novemcinctus |
Svartman et al., 2006 |
|
CARNIVORA |
Bassariscus astutus |
Nash et al., 2008 |
|
Procyon lotor |
Perelman
et al., 2008 |
|
|
Conepatus leuconotus |
Perelman
et al., 2008 |
|
|
Mephitis
macroura |
Perelman
et al., 2008 |
|
|
Mephitis mephitis |
Perelman
et al., 2008 |
|
|
Ailuropoda melanoleuca |
Nash et al., 1998 |
|
|
Vulpes volpe |
Yang et al., 1999 |
|
|
Graphodasky et al., 2000 |
||
|
Phoca vitulina |
Froenicke et al., 1997 |
|
|
Felis catus |
Wienberg 1997 |
|
|
Yang et al., 2000 |
||
|
Murphy et al., 2007 |
||
|
Nie et al., 2002 |
||
|
Canis familiaris |
Breen et al., 1999 |
|
|
Yang et al., 2000 |
||
|
Graphodatsky et al., 2001 |
||
|
Graphodasky et al., 2002 |
||
|
Martes foina |
Nie et al., 2002 |
|
|
|
|
Perelman
et al., 2008 |
|
Mustela vison |
Hameister et al., 1997 |
|
|
Graphodasky et al., 2000 |
||
|
Graphodasky et al., 2001 |
||
|
Nie et al., 2002 |
||
|
Mustela putorius |
Cavagna et al., 2000 |
|
|
Tremarctos ornatus |
Tian et al., 2004 |
|
|
PHOLIDOTA |
Manis javanica |
Yang et al., 2006 |
|
Manis pentadactyla |
Nie et al., 2009 |
|
|
CETARTIODACTYLA |
Damaliscus hunter |
Chaves et al., 2004 |
|
Sus scrofa |
Balmus et al., 2007 |
|
|
Froenicke and Wienberg 2001 |
||
|
Ovies aries |
Froenicke and Wienberg 2001 |
|
|
Camel dromedaries |
Balmus et al., 2007 |
|
|
Bos Taurus |
Balmus et al., 2007 |
|
|
Hippopotamus amphibious |
Kulemzina et al., 2009 |
|
|
Giraffa camelopardalis |
Kulemzina et al., 2009 |
|
|
Huang et al., 2008 |
||
|
Okapia johnstoni |
Kulemzina et al., 2009 |
|
|
Tursiops truncatus |
Bielec et al., 1998 |
|
|
Globicephala melas |
Kulemzina et al., 2009 |
|
|
Moschus moschifera |
Kulemzina et al., 2009 |
|
|
PERISSODACTYLA |
Equus caballus |
Yang et al., 2004 |
|
Malayan tapir |
Trifonov et al., 2008 |
|
|
Diceros bicornis |
Trifonov et al., 2008 |
|
|
Equus zebra |
Trifonov et al., 2008 |
|
|
Equus burcelli |
Yang et al., 2004 |
|
|
Equus asinum |
Trifonov et al., 2008 |
|
|
Equus grevyi |
Trifonov et al., 2008 |
|
|
Equus zebra harmannae |
Trifonov et al., 2008 |
|
|
Equus hemionus onager |
Trifonov et al., 2008 |
|
|
Equus przewalskii |
Trifonov et al., 2008 |
|
|
Ceratotherium simum |
Trifonov et al., 2008 |
|
|
Tapirus indicus |
Trifonov et al., 2008 |
|
|
Tapirus terestris |
Trifonov et al., 2008 |
|
|
Tapirus bairdii |
Trifonov et al., 2008 |
|
|
Tapirus pinchaque |
Trifonov et al., 2008 |
|
|
CHIROPTERA |
Asellicus stolickanus |
Mao et al., 2007 |
|
Megaderma Spasma |
Mao et al., 2008 |
|
|
Taphozous melanopon |
Mao et al., 2008 |
|
|
Myotis myotis |
Volleth et al., 2002 |
|
|
Mormopterus planiceps |
Volleth et al., 2002 |
|
|
Hipposideros larvatus |
Volleth et al., 2002 |
|
|
Mao et al., 2007 |
||
|
Rhinolophus mehelyi |
Volleth et al., 2002 |
|
|
Eonycteris spelaea |
Volleth et al., 2002 |
|
|
Glassophaga soricina |
Volleth et al., 1999 |
|
|
Mormopterus jugularis |
Richard et al., 2010 |
|
|
Miniopterus griveaudi |
Richard et al., 2010 |
|
|
Myotis goudoti |
Richard et al., 2010 |
|
|
Myzopoda aurita |
Richard et al., 2010 |
|
|
PRIMATI |
||
|
Prosimii |
Galago moholi |
Stanyon et al., 2002 |
|
Nie et al., 2006 |
||
|
Cardone et al., 2002 |
||
|
Otolemur crassicaudatus |
Stanyon et al., 2002 |
|
|
Otolemur garnettii |
Stanyon et al., 2006 |
|
|
Eulemur macaco macaco |
Muller et al., 1997 |
|
|
Cardone et al., 2002 |
||
|
Eulemur fulvus mayottensis |
Muller et al., 1997 |
|
|
Lemur catta |
Cardone et al., 2002 |
|
|
Nycticebus coucang |
Stanyon et al., 2006 |
|
|
Nie et al., 2006 |
||
|
Warter et al., 2005 |
||
|
Hapalemur griseus |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur mustelinus |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur jamesi |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur leucopus |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur ruficaudatus |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur edwardisi |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Lepilemur microdon |
Warter et al., 2005 |
|
|
Rumpler et al., 2008 |
||
|
Deubentonia madagascariensis |
Warter et al., 2005 |
|
|
Microcebus murinus |
Warter et al., 2005 |
|
|
Avahi laniger |
Warter et al., 2005 |
|
|
Propithecus verreauxi |
Warter et al., 2005 |
|
|
Indri indri |
Warter et al., 2005 |
|
|
Platyrrhinae |
Ateles geoffroy |
Morescalchi et al., 1997 |
|
Ateles paniscus chamek |
Suanez et al., 2001 |
|
|
Ateles belzebuth hybridus |
Garcia et al., 2002 |
|
|
Ateles belzebuth marginatus |
De oliveira et al., 2005 |
|
|
Brachyteles arachnoides |
De oliveira et al., 2005 |
|
|
Lagothrix lagothrica |
Stanyon et al., 2001 |
|
|
Alouatta belzebul |
Consigliere et al., 1998 |
|
|
Alouatta caraya |
de Oliveira et al., 2005 |
|
|
Stanyon et al., 2011 |
||
|
Alouatta seniculus arctoidea |
Consigliere et al., 1998 |
|
|
Alouatta seniculus sara |
Consigliere et al., 1998 |
|
|
Alouatta seniculus macconelli |
de Oliveira et al., 2002 |
|
|
Alouatta fusca clamitans |
de Oliveira et al., 2002 |
|
|
Alouatta fusca fusca |
de Oliveira et al., 2002 |
|
|
Alouatta guariba guariba |
Stanyon et al., 2011 |
|
|
Callicebus moloch |
Stanyon et al., 2003 |
|
|
Callicebus lugens |
Stanyon et al., 2003 |
|
|
Callicebus cupreus |
Dumas et al., 2005 |
|
|
Callicebus donacophilus pallescens |
Barros et al., 2003 |
|
|
Cebus nigrivitatus |
Garcia et al., 2002 |
|
|
Cebus apella |
Garcia et al., 2002 |
|
|
C. apella olivaceus |
Amaral et al., 2008 |
|
|
C. apella robustus |
Amaral et al., 2008 |
|
|
C. apella paraguayanus |
Amaral et al., 2008 |
|
|
Cebus capucinus |
Richard et al., 1996 |
|
|
Cebus albifrons |
Garcia et al., 2002 |
|
|
Saimiri sciureus |
Stanyon et al., 2000 |
|
|
Dumas et al., 2007 |
||
|
Cebuella pygmaea |
Neusser et al., 2001 |
|
|
Dumas et al., 2007 |
||
|
Callithrix jacchus |
Neusser et al., 2001 |
|
|
Callithrix argentata |
Neusser et al., 2001 |
|
|
Saguinus oedipus |
Muller et al., 2001 |
|
|
Neusser et al., 2001 |
||
|
Callimico Goeldii |
Neusser et al., 2001 |
|
|
Dumas et al., 2007 |
||
|
Pithecia irrorata |
Filotelo et al., 2010 |
|
|
Cacajao calvus rubicundus |
Filotelo et al., 2010 |
|
|
Chiropotes utahicki |
Stanyon et al., 2004 |
|
|
Leontopithecus crysomales |
Serreau et al., 2004 |
|
|
Aotus lemurinus griseimembra |
Stanyon et al., 2011 |
|
|
Aotus nancymae |
Ruiz-Herrera et al., 2005 |
|
|
Catarrhinae |
Macaca fascicularis |
Ruiz-Herrera et al., 2002 |
|
Macaca arctoides |
Ruiz-Herrera et al., 2002 |
|
|
Mandrillus sphinx |
Ruiz-Herrera et al., 2002 |
|
|
Macaca silvana |
Wienberg et al., 1992 |
|
|
Macaca fuscata |
Wienberg et al., 1992 |
|
|
Macaca nemestrina |
Muller and Wienberg 2001 |
|
|
Chlorocebus aethiops |
Finelli et al., 1999 |
|
|
Cercopithecus erythrogaster |
Moulin et al., 2008 |
|
|
Cercopithecus neglectus |
Stanyon et al., 2005 |
|
|
Cercopithecus stampfii |
Moulin et al., 2008 |
|
|
Cercopithecus albogularis labiatus |
Dumas and Sineo 2010 |
|
|
Cercopithecus mitis mitis |
Dumas and Sineo 2010 |
|
|
Erythrocebus patas |
Stanyon et al., 2005 |
|
|
Colobus guereza |
Bigoni et al., 1997 |
|
|
Trachipithecus cristatus |
Bigoni et al., 1997b |
|
|
Presbytys Cristata |
Bigoni et al., 1997 |
|
|
Nasalys larvatus |
Bigoni et al., 2003 |
|
|
Pygathrix nemaeus |
Bigoni et al., 2004 |
|
|
Semnopithecus francoisi |
Nie et al., 1998 |
|
|
Semnopithecus phayrei |
Nie et al., 1998 |
|
|
Hylobates lar |
Jauch et al., 1992 |
|
|
Hylobates moloch |
Jauch et al., 1992 |
|
|
Hylobates klossi |
Jauch et al., 1992 |
|
|
Hylobates concolor |
Koehler et al., 1995 |
|
|
Muller and Wienberg 2001 |
||
|
Symphalangus syndactylus |
Muller and Wienberg 2001 |
|
|
Muller et al., 2003 |
||
|
Nomascus leucogenesis |
Nie et al., 2001 |
|
|
Nomascus concolor |
Muller et al., 2003 |
|
|
Bunopithecus hoolock |
Muller et al., 2003 |
|
|
Pongo pygmaeus |
Jauch et al., 1992 |
|
|
Gorilla gorilla |
Jauch et al., 1992 |
|
|
Pan troglodytes |
Jauch et al., 1992 |
|
|
DERMOPTERA |
Galeopterus variegatus |
Nie et al., 2008 |
|
SCANDENTIA |
Tupaia belangeri |
Muller et al., 1999 |
|
LAGOMORPHA |
Oryctolagus cuniculus |
Korstanje et al., 1999 |
|
Hayes et al., 2002 |
||
|
RODENTIA |
Sciurus carolinensis |
Stanyon
et al., 2003 |
|
Li et al., 2004 |
||
|
Sicista betulina |
Graphodasky et al., 2008 |
|
|
Petaurista albiventer |
Li et al., 2004 |
|
|
Tamias sibiricus |
Li et al., 2004 |
|
|
Castor fiber |
Graphodasky et al., 2008 |
|
|
Pedetes capensis |
Graphodasky et al., 2008 |
|
|
Callosciurus erythracheus |
Li et al., 2004 |
|
|
Menetes berdmorei |
Richar et al., 2003 |
|
|
Mus musculus |
||
|
Rattus norvegicus |
||
|
Rattus rattus |
Cavagna et al., 2002 |
|
|
Apodemus sylvaticus |
Stanyon et al., 2004 |
|
|
EULIPOTYPHLA |
Sorex araneus |
Ye et al., 2006 |
|
Blarinella griselda |
Ye et al., 2006 |
|
|
Hemiechinus auritus |
Yang et al., 2006 |
|
|
Neotetracus sinesi |
Ye et al., 2006 |
|
|
Talpa europea |
Volleth and Muller 2006 |
|
|
Dickens et al., 1998 |
Tabella 1. Lista di specie analizzate
mediante painting cromosomico e prese in considerazione nel presente lavoro con
relativo riferimento bibliografico.
Table 1. Lists the
species studied through
chromosome painting considerate in the present study and their relative
bibliography.
Mammiferi
euteri
La sintenia 4 dell’uomo è in associazione con il braccio piccolo dell’omologo del cromosoma 8p umano (4/8p) in tutti i mammiferi euteri, eccetto nei primati (Stanyon et. al., 2008), negli elefanti (Yang et al., 2003) e nei sirenidi (kellogs et al., 2007), motivo per cui l’associazione è stata inclusa nel cariotipo ancestrale di tutti i mammiferi euteri (2n=46) (Ferguson-Smith and Trifonov 2007, Robinson and Ruiz-Herrera 2008).
L’esistenza di tale associazione nel cariotipo ancestrale dei mammiferi placentati inizialmente era stata alquanto dibattuta; fu ipotizzato che la sintenia umana fosse presente nel cariotipo ancestrale dei mammiferi come singolo cromosoma da Chowdhary (et al. 1998); infatti mediante i primi studi di painting l’associazione 4/8p non era stata mappata per esempio nello scoiattolo (Volleth et al., 2002), nel delfino (Bielec et al., 1998), in tupaia (Muller et al., 1999) e nel cane (Breen et al., 1999) probabilmente a causa di problemi tecnici dovuti alla bassa efficienza di ibridazione del piccolo segmento HSA 8p. Successivamente il painting cromosomico comparativo ha fornito la prova che l’associazione 4/8p è presente nei mammiferi Boroeuteri (Richard et al., 2003, Svartman et al., 2004, Wienberg et al., 2004). Uno studio recente sulla sintenia 4 ha permesso di individuare le bande cromosomiche e i punti di rottura coinvolti nei principali riarrangiamenti che si sono verificati nel corso dell’evoluzione genomica nei mammiferi (Picone et al., 2010).
Mediante la genomica comparativa, in particolare con il sequenziamento del DNA e l’analisi di blocchi sintenici è stato dimostrato che nel cariotipo ancestrale dei mammiferi euteri dell’emisfero boreale (Laurasiatheria e Euarchontoglires) e nel cariotipo dei Ferungulati (Cetartiodactyla, Perissodactyla, Carnivora e Pholidota) è presente l’associazione 4/8p/4 (Murphy et al., 2001, Froenicke et al., 2005). Recentemente è stata dimostrata la presenza dell’associazione 4/8p nell’opossum (Monodelphis domestica- 4qt/8p/4pq, Mikkelsen et al., 2007) e nel gallo (Gallus gallus- 4/8p/4/8p/4/8p, Murphy et al., 2005, Robinson e Ruiz-Herrera 2008), “outgroups” degli euteri, ciò ha permesso di confermare che la stessa è presente non solo nell’antenato comune dei Metateria (mammiferi placentati) ma anche nell’antenato comune degli Amnioti (rettili, uccelli e mammiferi).
L’analisi comparata dei dati cromosomici pubblicati, ottenuti mediante citogenetica molecolare, permette di ricostruire, a partire dall’organizzazione dell’omologo del cromosoma 4 nel cariotipo ancestrale dei mammiferi euteri, le tappe evolutive principali che la sintenia umana 4 ha subito durante l’origine e la diversificazione di ciascun clade dei mammiferi a partire da circa 90 milioni di anni fa. Le tappe, individuate mediante il presente lavoro di revisione, sono state riportate su un albero filogenetico che tiene in considerazione la suddivisione dei mammiferi in quattro superordini (Murphy et al., 2001, Ferguson-Smith and Trifonov, 2007) (Fig.1).
Afrotheria
In Orycteropus afer (Tubulidentata), in Elephantulus
rupestris (Macroscelidae) e in Chrysochloris
asiaticus (Afrosoricida)
è presente l’associazione 4/8p/2, carattere
derivato condiviso tra Afrosoricidae, Tubelidentata e Macroscelidea (Robinson et al., 2004, Swartman et al., 2004 ). La stessa associazione è
presente anche in due Boroeuteri: Manis
javanica (Pholidota) e Oryctalagus
cuniculus (Lagomorpha) (Yang et al., 2006, Korstanje et al., 1999), anche se il painting
reciproco tra armadillo (Tubelidentata) e pangolino (Pholidota) ha permesso di
dimostrare che non esiste una reale omologia nell’associazione che è frutto di
una convergenza evolutiva (Robinson
et al., 2004); la stessa cosa è vera presumibilmente anche per Oryctalagus cuniculus.
In
Trichechus manatus (Sirenia) l’associazione ancestrale 4/8p si è persa e la mancanza di
tale associazione rappresenta un tratto derivato (Kellogg et al., 2007). Anche
nell’ordine Proboscidea (Loxodonta
africana e Elephas maximus)
l’associazione ancestrale 4/8p si è persa per fissione e sono inoltre presenti
due nuove associazioni umane 4/16 e 15/4 che rappresentano caratteri derivati
(Yang et al., 2003). La mancanza dell’associazione 4/8p nei sirenii e
negli elefanti potrebbe essere un carattere derivato nei Paenungulata
(Sirenia, Proboscidea, e Hyracoidea), anche se non è stato ancora pubblicato
nessun lavoro di painting con sonde umane in Hyracoidea. Altrettanto probabile
è che il segnale dell’8p non sia stato rivelato per problemi tecnici data la
presenza di tale associazione nella maggior parte dei mammiferi (Kellogg et
al., 2007, Swartman et al., 2004). In realtà quest’ultima ipotesi sembra essere
meno probabile in quanto mediante estrapolazioni di dati di “cross species
hybridization” ottenuti utilizzando sonde di elefanti e iracoidei su tricheco
risulterebbe mancare l’associazione 4/8p anche negli Hyracoidea (Pardini et
al., 2007).
Xanarthra
L’associazione sintenica 4/8p è inclusa nel cariotipo ancestrale degli
Xanarthra ed è conservata in C.
hoffmannii (Svartman et al., 2006); inoltre la
sintenia 4 presenta da 2 a 5 frammenti in Dasypus novemcinctus (Ciungulata), Choloepus didactylus e in Tamandua tetradactyla (Pilosa) (Svartman et al., 2006,
Yang et al., 2006).
Laurasiatheria
Tra i carnivori in Phoca vitulina (Froenicke et al., 1997), Procyon lotor (Perelman et al., 2008), Mustela vison (Hameister et al., 1997) e Felis catus (Yang et al., 2000) troviamo l’associazione 4/8p/4 su un singolo cromosoma. In Mephitis mephitis, Ailuropoda melanoleuca, Mustela putorius e Canis familiaris la sintenia 4 ha subito diversi riarrangiamenti (fissioni e traslocazioni) che hanno formato vari blocchi di sintenia e l’associazione 4/8p è ripetuta due volte (Breen et al., 1999, Yang et al., 2000, Graphodatsky et al., 2000, 2001, 2002).
Nel cariotipo
ancestrale dei Cetartiodactyla è presente l’associazione 4/8p una volta sola (4/8p, 4) più un ulteriore frammento (Kulemzina et al. 2009).
Questa organizzazione è presente in Camel
dromedaries (Balmus et al., 2007) Tursiops
truncatus e Globicephala melas (Kulemzina et al. 2009). L’associazione 4/8p ha subito
una fissione in Sus scrofa e in Bos
taurus, infatti sono presenti due blocchi di associazione 4/8p ed un
ulteriore frammento di sintenia 4 (4/8p,
4/8p, 4) (Froenicke et Wienberg 2001, Balmus et al., 2007). Ulteriori
riarrangiamenti si sono verificati a carico di questi blocchi di sintenia nelle
varie specie dell’ordine.
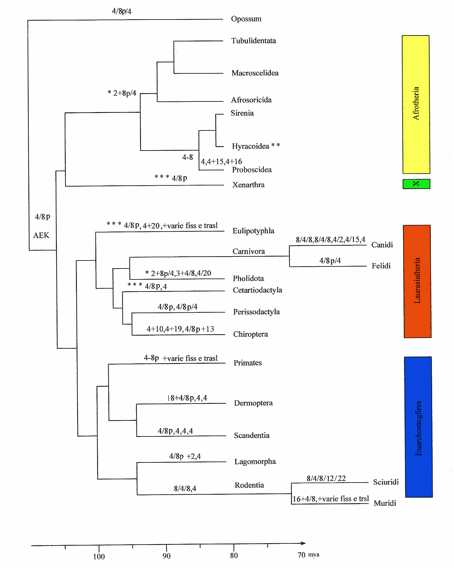
Figura
1.
Presunti blocchi ancestrali di sintenia 4 dell’uomo nei
mammiferi placentati e riarrangiamenti che si sono verificati a carico di
questa sintenia nelle varie linee filogenetiche; rielaborazione della figura di
Ferguson Smith and Trifonov (2007); AEK= Ancestral
Eutherian karyotype- cariotipo
ancestrale degli euteri; * Convergenza evolutiva; **Dati non ancora prodotti;
*** diversi frammenti di sintenia 4 nelle varie specie.
Figure 1. Hypothetical ancestral blocks
of the synteny 4 in eutherian mammals and rearrangements that occurred on this
synteny in the various phylogenetics lineages; figure modified from Ferguson
Smith and Trifonov (2007); AEK= Ancestral
Eutherian karyotype; *Convergence; **
data not jet produced; *** different segments of the synteny 4 in the various
species.
Nel cariotipo ancestrale dei Perissodattili sono
presenti due blocchi di sintenia 4/8p e 4/8p/4 (Trifonov et al., 2008).
L’associazione 4/8p/4 è intatta nel tapiro Malesiano, nella zebra e nell’asino
(Malayan Tapir, Equus asinus e Equus
zebra) (Yang et al. 2004),
o distribuita in due cromosomi negli altri tapiri, nei rinoceronti neri e
bianchi, in onagro e nel cavallo (T. bairdii, Diceros bicornis, Equus
caballus, E. hemionus onager) (Trifonov et al., 2008).
Nei Chiroptera (Asellicus stolickanus, Megaderma spasma, Taphozous melanopon, Myotis myotis, Mormopterus planiceps, Hipposideros larvatus, Rhinolophus mehelyi, Eonycteris spelaea, Glassophaga soricina) oltre all’associazione 4/8p/13 altri due frammenti di sintenia 4 formano due nuove associazioni tipiche di questo ordine: l’associazione HSA 4/10 e l’associazione HSA 4/19 (Volleth et al., 2002, 1999, Mao et al., 2007, 2008).
L’associazione 4/8p negli Eulipotyphla ha subito diversi riarrangiamenti incluse fissioni e traslocazioni. Una fissione ha rotto l’associazione 4/8p in Blarinella e in Neotetracus sinensis. In Sorex araneus, Blarinella griselda e Neotetracus sinesi l’associazione condivisa derivata 4/20 (Ye et al., 2006) risulta un buon marker citogenetico che lega gli Eulipotyphla (Ye et al. 2006) ai Pholidota (Yang et al., 2006).
Nell’ordine Pholidota oltre l’associazione 4/20 è presente l’associazione 2/4q+8p in Manis javanica (Yang et al., 2006) che, come detto in precedenza, è apparsa molto simile in un primo momento all’associazione 2/8p/4 trovata nell’armadillo (Tubulidentata). Un’associazione apomorfica è la 7/4q+8 in Manis pentadactyla (Nie et al., 2009).
Euarchontoglires
In Galeopterus variegatus (Dermopthera) troviamo l’associazione 4/8p in associazione con la sintenia umana 18 (18/4/8p) e altri 2 blocchi di sintenia 4 (Nie et al., 2008).
In Oryctolagus cunuculus (Lagomorpha) riscontriamo l’associazione 4/8p/2 e un ulteriore frammento omologo al cromosoma 4 umano (Korstanje et al., 1999, Hayes et al., 2002).
In Tupaia belangeri (Scandentia) mediante i primi studi di painting cromosomico (Muller et al., 1999) sono stati trovati quattro segmenti della sintenia umana 4 ma non l’associazione 4/8p; ciò permise di ipotizzare l’esistenza di un legame filogenetico tra primati e scadentia. Recentemente studi di painting reciproco su Tupaia minor (Scandentia) dimostrerebbero la presenza di una associazione 4/8p smentendo la precedente ipotesi (Stanyon comunicazione personale).
Nel cariotipo
ancestrale dei roditori si trova l’associazione 8p/4/8p ed un frammento di 4
(Graphodasky et al., 2008, Romanenko et al., 2010). Una organizzazione più
conservata la si riscontra in Sciurus carolinensis, Petaurista albiventer e Tamias sibiricus (Sciuridae) (Stanyon et al., 2003, Li et al
2004; 2006), in Castor fiber (Castoridae), Pedetes
capensis (Pedetidae) e Sicista betulina (Graphodasky et al., 2008). Invece
nel ratto e topo (Rattus norvegicus e Mus musculus - Muridae) pur avendo l’associazione 4/8p conservata la
sintenia 4 ha subito diverse fissioni, formando fino a 9 blocchi sintenici, e
traslocazioni, formando nuove
associazioni (Stanyon
et al., 2004, Graphodasky et al., 2008, Ensembl.org).
Ordine
dei Primati
L’associazione umana 4/8p si è persa mediante fissione, prima della divergenza delle proscimmie e scimmie tra i 90 e 60 milioni di anni fa; infatti il painting cromosomico comparativo ha permesso di dimostrare che le sonde umane 4 e 8 mappano in cromosomi differenti nei primati (Haig 1999), per tale motivo l’associazione 4/8p non è inclusa nel cariotipo ancestrale dei primati (Stanyon et al., 2008).
L’omologo del cromosoma 4 umano, è nei Primati, tranne qualche eccezione, un grande cromosoma submetacentrico. Questo cromosoma è conservato nella maggior parte dei primati, ma è soggetto ad un elevato tasso di riarrangiamenti, soprattutto fissioni, inversioni pericentriche e formazione di nuovi centromeri, nei taxa caratterizzati da un’alta dinamica cromosomica, come per esempio nelle Proscimmie, nelle Platyrrhinae e nei Gibboni. Sulla base di evidenze fossili, dati molecolari e costituzione cromosomica delle specie esistenti si possono ipotizzare le tappe principali che hanno caratterizzano l’evoluzione della sintenia umana 4 nei Primati (Fig. 2).
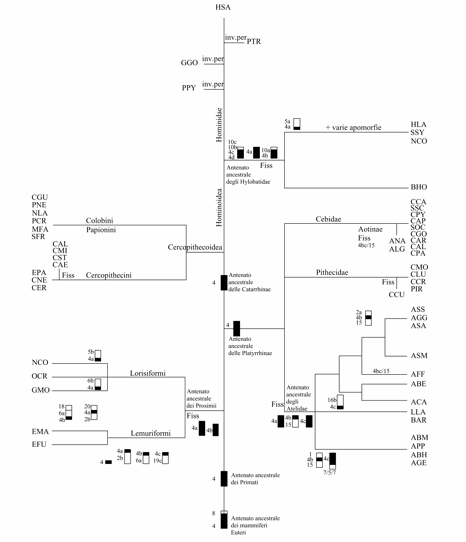
Figura 2. Presunti cromosomi ancestrali omologhi al cromosoma 4
umano nei primati e riarrangiamenti che si sono verificati a carico di questa
sintenia nei vari taxa.
Figure 2. Hypothetical
ancestral chromosomes homologous to the human chromosome 4 in primates and
rearrangements that occurred on this synteny in the various taxa.
Fiss= Fission, Trasl=
Traslocation, Inv per= pericentric Inversion
Nycticebus coucang
(NCO), Galago moholi (GMO),
Otolemur crassicaudatus (OCR), Eulemur macaco (EMA), Eulemur
fulvus (EFU), Lagothrix lagothrica (LLA), Alouatta caraya (ACA),
Alouatta belzebul (ABE), Alouatta fusca fusca (AFF), Alouatta
seniculus macconelli (ASM), Alouatta seniculus arctoidea (ASA), Alouatta
seniculus sara (ASS), Alouatta guariba guariba (AGG), Ateles
geoffri (AGE), Ateles belzebuth marginatus (ABM), Ateles paniscus
paniscus (APP), Pithecia irrorata (PIR), Brachyteles arachnoides (BAR),
Callicebus moloch (CMO), Callicebus cupreus (CCU),Callimico
Goeldii (GGO), Saguinus oedipus (SOE), Cebus capucinus (CAP),
Cebus robustus (CRO), Cebus albifrons (CAL), Cebus paraguyanus (CPA),
Cebuella pygmaea (CPY), Saimiri sciureus (SSC), Aotus nacymae (ANA),
Aotus lemurinus griseimembras (ALG),Cacajao calvus rubicundus (CCA), Cebus nigrivitatus (CNI).
Macaca fascicularis (MFA), Semnopithecus francoisi (SFR),
Cercopithecus aethiops (CAE), Cercopithecus mitis (CMI), Cercopithecus
albogularis labiatus (CAL), Cercopithecus erithrogaster (CER)
Cercopithecus stampfii (CST),
Erythrocebus patas (EPA) Colobus guereza (CGU), Presbytys
Cristata (PCR), Nasalys larvatus (NLA), Pygathrix nemacus
(PNE) Hylobates lar (HLA), Symphalangus syndactylus (SSY), Buonophitecus
hoolock (BHO), Pongo pygmaeus (PPY), Gorilla gorilla (GGO),
Pan troglodytes (PTR).
Proscimmie o Prosimii
Nelle Proscimmie la sintenia HSA 4 è andata incontro ad una fissione, nel loro cariotipo ancestrale troviamo due blocchi di sintenia (4a e 4b) (Stanyon et al., 2002, Stanyon et al., 2006), questi a loro volta sono andati incontro nelle diverse specie ad alcune traslocazioni che hanno formato nuove associazioni con altre sintenie umane (Nie et al., 2006, Muller et al., 1997, Warter et al., 2005, Rumpler et al., 2008). Nel cariotipo ancestrale dei lemuri sono presenti quattro frammenti omologhi al cromosoma 4 umano di cui tre sono in associazione con gli omologhi dei cromosomi umani HSA 2, 6 e 19. Nel cariotipo ancestrale dei loriformi sono presenti due blocchi di sintenia 4 (4a, 4b) di cui uno in associazione con un diverso segmento di cromosoma umano (Nie et al., 2006). In Nycticebus coucang (Lorisidae) (4a/5, 4) il blocco 4a forma l’associazione sintenica 4a/5, prodotto di una traslocazione con la sintenia umana 5 (Stanyon et al., 2006, Nie et al., 2006). In Galago moholi (4a/6, 4) (Galagidae) avviene invece una traslocazione tra il 4a e la sintenia umana 6 dando luogo all’associazione sintenica 4a/6.
Platyrrhinae o Scimmie del Nuovo
Mondo
Il nodo evolutivo della divergenza tra Platyrrhinae e Catarrhinae datato circa 40 milioni di anni fa è stato segnato, nella sintenia 4, da un’inversione pericentrica, dimostrata da Dutrillaux (1979) mediante bandeggio cromosomico. Stanyon e colleghi (2008) recentemente hanno ipotizzato, invece, che la sintenia umana 4 è stata caratterizzata in questo nodo evolutivo da uno spostamento (shift) del centromero. Tale riarrangiamento è stato dimostrato, a livello molecolare, mediante FISH con sonde di DNA clonate in BACs (Bacterial Artificial Chromosomes) in Callithrix jacchus e Homo sapiens.
Nella specie
analizzate dell’infraordine Platyrrhinae, appartenenti alle famiglie Cebidae e
Phitecidae, la sintenia 4 è conservata come unico cromosoma: in Callicebus moloch sul cromosoma 1
(Stanyon et al., 2000); in Callicebus
lugens (Stanyon et al., 2003), Saimiri
sciureus sul cromosoma 3 (Stanyon et al., 2000); in Cebus nigrivitatus (Garcia et al., 2002), in Cebus
capucinus (Richard et al., 1996), Cebus albifrons, C. robustus e C. paraguayanus sul 2 (Amaral et al., 2008);
in Saguinus oedipus sul
cromosoma 7; in
Cebuella pygmaea, Callithrix argentata, Callimico
goeldii sul cromosoma 9 (Neusser et al., 2001, Dumas
et al., 2007); in Phitecia irrorata sul cromosoma 5 e in Cacajao calvus rubicundus sul cromosoma
9 (Finotelo et al ., 2010). Tra i Pithecidae le eccezioni si riscontrano in Callicebus cupreus in cui la sintenia 4
è frammentata in due blocchi (Dumas et al., 2005) e nelle Aotinae, Aotus nancymae e A. lemurinus griseimembras dove sono presenti due blocchi di cui uno in
associazione con la sintenia umana 15 (4, 4/15) (Ruiz-herrera et al., 2005,
Stanyon et al., 2011).
L’organizzazione della sintenia 4 del cariotipo ancestrale degli Atelidae (de Oliveira et al., 2002, 2005) (4a, 4b/15, 4c), viene mantenuta in Lagothrix lagothrica (Stanyon et al., 2001) e Brachyteles aracnoidea (de Oliveira et al., 2005); l’associazione 4b/15 è una sinapomorfia che lega tutte le specie. In Ateles geoffroy (Morescalchi et al., 1997) ed Ateles belzebuth hybridus (Garcia et al.., 2002), l’ulteriore associazione sintenica 15/4/1 è una condizione apomorfa. In Alouatta caraya (Consigliere et al., 1998, Stanyon et al., 2011) e Alouatta belzebul (De Oliveira et al., 2002) (4b/15, 4a, 4c/16), il frammento 4c subisce una traslocazione sul 16c per cui l’associazione 4c/16c costituisce una sinapomorfia che lega queste specie. In Alouatta seniculus macconnelli (De Oliveira et al., 2002), Alouatta seniculus sara, Alouatta seniculus arctoidea (Consigliere et al. 1996) e in Alouatta guariba guariba (Stanyon et al., 2011) è presente l’associazione sintenica 2a/4b/15a che è una sinapomorfia condivisa. Nelle due sottospecie Alouatta fusca clamitas e fusca fusca i frammenti 4b e 4c hanno subito una fusione formando l’associazione sintenica 4bc/15 (de Oliveira et al., 2002). In Ateles belzebuth marginatus, A paniscus paniscus, Ateles belzebuth l’associazione 4b/15 ed il blocco 4c subiscono delle traslocazioni formando altre associazioni (De Oliveira et al., 2005).
Catarrhinae o Scimmie del Vecchio
Mondo
Nell’infraordine delle Catarrhinae, nella superfamiglia delle Cercopithecoidea e nella superfamiglia degli Hominoidea, l’omologo della sintenia umana 4 è un singolo cromosoma conservato in tutte le specie studiate tranne rispettivamente in alcuni Cercopithecini e negli Hylobatidae. Nelle Cercopithecoidea per esempio la sintenia 4 in Macaca fascicularis si riscontra sul cromosoma 4 (Ruiz-Herrera et al., 2002), in Erythrocebus patas sul cromosoma 12, in Cercopithecus neglectus sul cromosoma 4 (Stanyon et al., 2005), in Colobus guereza sul cromosoma 1, in Presbytys cristata sul cromosoma 3 (Bigoni et al., 1997), in Nasalys larvatus sul cromosoma 2 (Bigoni et al., 2003), in Pygathrix nemaeus sul 7 (Bigoni et al., 2004), in Semnopithecus francoisi sul cromosoma 3 (Nie et al., 1998) e in Cercopithecus erythrogaster (Moulin et al., 2008); Le eccezioni rispetto a questo “trend” sono state riscontrate nei Cercopithecini, Cercopithecus aethiops o Chlorocebus (Finelli et al., 1999), Cercopithecus stampfii (Moulin et al., 2008), C. mitis mitis e in C. albogularis labiatus (Dumas et Sineo 2010) dove sono presenti due frammenti.
La sintenia 4 nel cariotipo ancestrale dei gibboni (Hylobatidae) è costituita da tre blocchi, di cui due in associazione con frammenti di sintenia umana 10cb (10cb/4cd, 4a, 10a/4b); questa organizzazione ancestrale è conservata in Bunopithecus hoolock. In Hylobates lar, Symphalangus syndactylus e Nomascus concolor, il frammento 4a forma l’associazione sintenica derivata condivisa (sinapomorfia) 4a/5, prodotto di una traslocazione con la sintenia umana 5 (Muller et al., 2003).
Negli Hominidae gli omologhi della sintenia dell’uomo
4 in Pongo
pygmaeus Gorilla gorilla, Pan troglodytes, (Jauch et al., 1992) differiscono
a causa di inversioni (Nickerson
and Nelson 1998) i cui punti di rottura, diversi
gli uni dagli altri, sono stati identificati attraverso
la mappatura con sonde BACs (Marzella et al., 2000). Inoltre mediante il metodo
del sequenziamento è stato identificato il punto di rottura (4p14 e 4q21.3)
responsabile della inversione pericentrica verificatasi durante la divergenza
di Pan e homo (5-6 milioni di anni fa) (Keher- Sawatziki et al., 2005).
Conclusione
L’origine evolutiva della sintenia 4 dell’uomo la si può far risalire a 340 Milioni di anni fa per la presunta presenza dell’associazione 4/8p nei cariotipi ancestrali dei Metateria e degli Amnioti. L’analisi comparativa dei dati sugli omologhi del cromosoma 4 umano nei mammiferi euteri ha permesso di chiarire le tappe evolutive della sintenia umana 4 negli ultimi 90 milioni di anni, dimostrando che il cromosoma umano 4 è conservativo, ma meno di quanto si credesse in precedenza, in quanto ha subito diversi riarrangiamenti soprattutto nei taxa caratterizzati da un elevato tasso evolutivo.
La sintenia dell’uomo 4 è in associazione con il braccio piccolo del cromosoma 8p in tutti i mammiferi con qualche eccezione, motivo per cui l’associazione è stata inclusa nel cariotipo ancestrale di tutti i mammiferi (4/8p). Questa associazione conservata subisce però vari riarrangiamenti nei diversi raggruppamenti di mammiferi. Nel super ordine Xanarthra l’associazione 4/8p è conservata anche se sono presenti più blocchi di sintenia 4. Negli Afrotheria l’associazione 4/8p, seppur con alcune eccezioni, è conservata ed ha subito la prima traslocazione formando una nuova associazione con la sintenia 2 (4/8p/2); Le eccezioni negli Afrotheria si riscontrano nei Sirenia e negli Proboscidae dove l’associazione 4/8p è stata scissa per fissione e di conseguenza le sintenie 4 e 8 formano due cromosomi separati. Nei Laurasiatheria sono state individuate due forme alternative polimorfiche ancestrali della stessa associazione, una su un singolo cromosoma (4/8p/4) e l’altra su due (4/8p, 4/8p) (Trifonov et al., 2008). Negli Euarchontoglires l’associazione è diversamente riarrangiata nei vari ordini, tranne nei Primati, dove per fissione si sono formati due cromosomi separati, il 4 e 8.
Nei Primati, la sintenia dell’uomo 4 è un unico cromosoma submetacentrico con qualche eccezione; infatti nelle proscimmie, Atelidi, Cercopithecini e nei gibboni, la sintenia 4 ha subito diverse fissioni e traslocazioni. Nelle proscimmie sono presenti quattro blocchi sintenici prodotto di fissioni e nuove associazioni sinteniche risultato di traslocazioni; nelle Platyrrhianae la sintenia 4 è conservata tranne negli Atelidi dove sono presenti tre blocchi di sintenia e nuove associazioni sinteniche. Nelle Catarrhinae la sintenia è conservata tranne nei Cercopithecini, dove sono presenti due blocchi sintenici, e nei gibboni dove sono presenti tre blocchi di sintenia e varie nuove associazioni. Nell’uomo e nelle antropomorfe superiori è presente un unico blocco di sintenia equivalente alla forma ancestrale dei primati anche se le diverse forme omologhe differiscono per inversioni pericentriche diverse tra loro.
Ulteriori studi condotti grazie alla disponibilità di dati di sequenza insieme ad informazioni relative ai blocchi di sintenia permetterà di ampliare gli studi di genomica comparata fornendo una prospettiva più dettagliata sull’origine e l’evoluzione dei cromosomi umani.
Bibliografia
Amaral PJS, Finotelo LFM, De Oliveira, EHC,
Pissinati, A, Nagamachi, CY and Piezarcka J.C., 2008, Phylogenetic studies of the genus Cebus (Cebidae-Primates) using
chromosome painting and G-banding. BMC Evolutionary Biology, 8:169-178.
Balmus, G, Trifonov VA,
Biltueva LS, O’Brien, PCR, Alkalaeva, ES, Fu, B, Skidmore, JA, Allent,
Graphodatdsky, AS and Yang F., et al. 2007, cross-species
chromosome painting among camel, cattle pig and human: further insights into
the putative cetartiodactyla ancestral karyotype. Chromosome Research,
15: 499-514.
Barros, RM, Nagamachi, CY,
Pieczarka, JC, Rodrigues, LRR, Neusser, M., De Oliveira, EHC, Wienberg, J,
Muniz, JAPC. Rissino DJ and Muller S., 2003, Chromosomal studies in Callicebus donacophilus pallescens, with classic
and molecular cytogenetic approaches: multicolour FISH using human and Saguinus
oedipus painting probes. Chromosome Research, 11: 327-334.
Bielec, PE, Gallagher, DS,
Womack, JE, Busbee, DL 1998, Homologies between human and dolphin chromosome
detected by heterologous chromosome painting. Cytogenet Cell Genet. 81: 18-25.
Bigoni ,F, Stanyon, R,
Wimmer, R and Schempp, W, 2003 — Chromosome
painting shows that the proboscis monkey (Nasalis larvatus) has a derived
karyotype and is phylogenetically nested within Asian Colobines.
American Journal of Primatology, 60: 85-93.
Bigoni, F, Koehler U,
Stanyon, R, Morescalchi, AM and Wienberg, J, 1997b Mapping homology between human and black and white colobine monkey
chromosomes by fluorescent in situ hybridization. American Journal of
Primatology, 42: 289-298.
Bigoni, F, Koehler, U,
Stanyon, R, Ishida T and Wienberg, J, 1997a, Fluorescence in situ hybridization establishes homology between human
and silvered leaf monkey chromosomes, reveals reciprocal translocations between
chromosomes homologous to human Y/5, 1/9, and 6/16, and delineates an X1X2Y1Y2/
X1X1X2X2 sex-chromosome system. American Journal of Physical
Anthropology, 102: 315-327.
Bigoni, F., Houck, ML,
Ryder OA, Wienberg, J and Stanyon R., 2004, Chromosome painting shows that Pygathrix nemaeus has the most basal
karyotype among Asian Colobinae. International Journal of Primatology,
25: 679-688.
Bodega, B, Cardone, MF,
Müller, S, Neusser, M, Orzan, F, Rossi, E. Battaglioli, E. Marozzi, A Riva, P
and Rocchi, M, et al. 2007 — Evolutionary genomic remodelling of the human 4q subtelomere (4q35.2).
BMC Evolutionary Biology, 7: 39-52.
Breen, M, Thomas, R, Binns,
MM, Carter, NP And Langford, CF, 1999 Reciprocal chromosome painting reveals
detailed regions of conserved synteny between the karyotypes of the domestic
dog (Canis familiaris) and human. Genomics, 61: 145-155.
Cardone, MF, Ventura, M,
Tempesta, S, Rocchi, M And Archidiacono, N, 2002, Analysis of chromosome conservation in Lemur catta studied by
chromosome paints and BAC/PAC probes. Chromosoma, 111: 267-272.
Cavagna, P, Menotti, A,
Stanyon, R 2000, Genomic homology of the domestic ferret with cats and humans.
Mamm Genome. 11: 866-70
Chaves, R, Fronicke, L,
Guedes-Pinto,Wienberg, J, 2004, Multidirectional chromosome painting between
the Hirola antelope (Damaliscus hunteri, Alcelaphini, Bovidae), sheep and
human. Chromosome Res. 12: 495–503
Chowdhary, BP, Raudsepp, T,
Fronicke, L, and Scherthan, H 1998, Emerging patterns of comparative genome
organization in some mammalian species as revealed by Zoo-FISH. Genome Res.8:
577–589.
Consigliere,
S, Stanyon, R, Koelher, U, Agoramoorthy G, Wienbreg J, 1996. Chromosome
painting defines genomic rearrangements between red howler monkey subspecies. Chromosome Res. 4(4): 264-70.
Consigliere, S, Stanyon, R,
Koelher, U, Arnold, N, And Wienberg J, 1998, In situ hybridization (FISH)
maps chromosomal homologies between Alouatta belzebul (Platyrrhini, Cebidae)
and other primates and reveals extensive interchromosomal rearrangements
between howler monkey genomes. American Journal of Primatology, 46 (2):
119-133.
De Oliveira, EHC, Neusser,
M, Figueireido, WB, NagamachiC, Pieczarca, JC, Sbalquiero, IJ, Wienberg, J, and
Muller, S, 2002, The phylogeny of
howler monkeys (Alouatta, Platyrrhini): reconstruction by multicolor
cross-species chromosome painting. Chromosome Research,10 (8): 669-683.
De Oliveira, EHC, Neusser,
M, Nagamachi, C, Pieczarca, JC, Sbalquiero, IJ and Muller, S, 2005, Phylogenetic inferences of Atelinae
(Platyrrhini) based on multi-directional chromosome painting in Brachyteles
arachnoides, Ateles paniscus paniscus and Ateles b. marginatus.
Cytogenetic and Genome Research, 108 (1-3): 183-190.
Dixkens, C, Klett, C,
Bruch, J et al, 1998, Zoo-FISH analysis in insectivores: FEvolution extols the
virtue of the status quo_. Cytogenet Cell Genet 80: 61-67.
Dumas,
F, and Sineo, L, 2010, Chromosomal dinamycs in Cercopithecini studied by
Williams beuren probe mapping. Caryologia (vol 3, 4:435-442).
Dumas, F, Bigoni, F, Stone,
G, Sineo, L and Stanyon R, 2005, Mapping genomic rearrangements in titi
monkeys by chromosome flow sorting and multidirectional in-situ hybridization.
Chromosome Research, 13 (1): 85-96.
Dumas, F, Bigoni, F, Stone,
G, Sineo, L and Stanyon, R, 2007, Phylogenomics of species from four genera of
New World monkeys by flow sorting and reciprocal chromosome painting.
BMC Evolutionary Biology, 7 (Suppl 2): 11-25.
Dutrillaux, B, 1979,
Chromosomal evolution in primates: tentative phylogeny from Microcebus murinus
(Prosimian) to man. Hum Genet 48:251–314.
Ferguson-Smith, MA and
Trifonov, V, 2007, Mammalian karyotype
evolution. Nature Reviews Genetics, 8: 950-962.
Finelli, P, Stanyon, R,
Plesker, R, Ferguson-Smith, Ma, O’brien, PCM. and Wienberg, J, 1999, Reciprocal
chromosome painting shows that the great difference in diploid number between
human and African green monkey is mostly due to non- Robertsonian fissions.
Mammalian Genome, 10: 713–718.
Finotelo, L, Amaral, P,
Pieczarka, J, De Oliveira, E, Pissinati, A, Neusser, M, Muller, S, Nagamachi,
C, 2010, Chrmosome phylogeny of the subfamily Pitheciinae (Platyrrhini,
Primates) by classic cytogenetics and chromosome painting. BMC Evolutionary
Biology 10:189
Froenicke, L, 2005, Origins of primate chromosomes – as
delineated by Zoo-FISH and alignments of human and mouse draft genome sequences.
Cytogenetic and Genome Research, 108 (1-3):122- 138.
Froenicke, L, Caldes, M,
Graphodatsky, A, Müller, S, Lyons, L, Robinson,T, Volleth, M, Yang, F,
Wienberg, J, 2006, Are molecular cytogenetics and bioinformatics suggesting
diverging models of ancestral mammalian genomes? Genome Res 16:306–310
Froenicke, L, Muller-Navia,
J, Romanakis, K, Scherthan, H, 1997, Chromosomal homologies between human,
harbor seal (Phoca vitulina) and the putative ancestral carnivore Karyotipe
revelead by Zoo-FISH. Chromosoma. 106: 108-13.
Froenicke, L, Wienberg, J,
2001. Compartive chromosome painting defines the high rate of Karyotype changes
between pigs and bovids. Mamm Genome. 12: 442-9.
Gandelman, KY, Gibson, L,
Meyn, MS and Yangfeng, TL, 1992, Molecular
definition of the smallest region of deletion overlap in the Wolf-Hirschhorn
syndrome. American Journal of Human Genetics, 51: 571– 578.
Garcia F, Ruiz-Herrera A,
Egzocue M, Ponsa’ M and Garcia M, 2002, Chromosomal homologies between Cebus and
Ateles (Primates) based on ZOO-FISH and G-banding comparison. American
Journal of Primatology, 57: 177-188.
Gilbert, C, O'Brien, PC, Bronner,G, Yang, F, Hassanin, A, Ferguson-Smith, M A and Robinson TJ, 2006, Chromosome
painting and molecular dating indicate a low rate of chromosomal evolution in
golden moles (Mammalia, Chrysochloridae). Chrom Res 14(8):793-803
Graphodatsky, A,S, Yang, F,
Perelman, P, O’brien, PCM, Serdukova, N, Milne, BS, Biltueva, LS, Fu, B,
Vorobieva ,NV and Kawada, SI, et al, 2002 Comparative
molecular cytogenetic studies in the order Carnivora: mapping chromosomal
rearrangements onto the phylogenetic tree. Cytogenetic and Genome Research, 96: 137-145.
Graphodatsky, AS, Yang, F,
Dobigny, G, Romanenko, SA, Biltueva, LS, Perelman, PL, Bekleshieva, VR,
Alkalaeva, EZ, Serdukova, NA and Ferguson-Smith, MA, et a,. 2008, Tracking the evolution of genome
organization in rodents by ZOO-FISH. Chromosome Research, 16: 261-274.
Graphodatsky, AS, Yang, F,
O’brien, PCM, Perelman, P, Milne, BS, Serdukova, N, Kawada, SI and
Ferguson-Smith, MA, 2001, Phylogenetic implications of the 38 putative
ancestral chromosome segments for four canid species. Cytogenetics and
Cell Genetics, 92: 243-247.
Graphodatsky, AS, Yang, F,
O’brien, PCM, Serdukova, N, Milne, BS, Trifonov, V, and Ferguson-smith, M,
2000a, A comparative chromosome map of
the Arctic fox, red fox and dog defined by chromosome painting and high
resolution G-banding. Chromosome Research, 8: 253-263.
Haig, D, 1999, A brief history of Human autosomes.
Trans. Soc. London B 354 : 1447-1470.
Hameister, H, Klett, C,
Bruch, J, Dixkens ,C, Vogel, W and Christensen K, 1997, Zoo-FISH analysis: the American mink (Mustela vison) closely resembles
the cat karyotype. Chromosome Research, 5: 5-11.
Hayes, H, Rogel-Gaillard
,C, Zijlstra, C, De Haan, NA, Urien, C, Bourgeaux, N, Bertaud, N Andbosma, AA,
2002, Establishment of an R-banded
rabbit karyotype nomenclature by FISH localizationof 23 chromosome-specifi c
genes on both G- and R-banded chromosomes. Cytogenetic and Genome
Research, 98: 199-205.
Hillier, LW, Graves, TA,
Fulton, RS, Fulton, LA, Pepin, KH, Minx, P, Wagner-Mcpherson C, Layman, D,
Wylie, K And Sekhon, M, et al. 2005,
Generation and annotation of
the DNA sequences of human chromosomes 2 and 4. Nature, 434 (7034):
724-731.
Huang, L, Nesterenko, A,
Nie, W, Wang, J, Su ,W, Graphodatsky, AS and Yang, F, 2008, Karyotype
evolution of giraffes (Giraffa camelopardalis) revealed by cross-species
chromosome painting withChinese muntjac (Muntiacus reevesi) and human (Homo
sapiens) paints. Cytogenetic and Genome Research, 122: 132-138.
Iannuzzi, L, Di Meo, GP,
Peruccati, A and Incarnato, D, 1999, Comparison
of the human with the sheep genomes by use of human chromosomespecific painting
probes. Mammalian Genome, 10:719-723.
Jauch, A, Wienberg, J, Stanyon,
R, Arnold, N, Tofanelli, S, Ishida, T and Cremer, T, 1992, Reconstruction
of genomic rearrangements in great apes and gibbons by chromosome painting. Proceedings
of the National Academy of Sciences of the United States of America, 89:
8611-8615.
Kehrer-Sawatzki, H.,
Sandig, C,A,, Goidts, V, and Hameister, H, 2005, Breakpoint analysis of the
pericentric inversion between chimpanzee chromosome 10 and the homologous
chromosome 12 in humans. Cytogenet Genome Res 108:91–97
Kellogg, M, Burket,t S,
Dennis, TR, Stone, G, Gray, BA, Mcguire, PM, Zori, RT and Stanyon, R, 2007, Chromosome painting in the manatee supports
Afrotheria and Paenungulata. BMC Evolutionary Biology, 7: 6-13.
Kemkemer, C, Kohn, ,M,
Cooper, DN, Froenicke, L, Hügel, J, Hamaister, H and Keher-Sawatzki, H,
2009 Gene synteny comparisons between different vertebrates provide new
insights into breakage and fusion events during mammalian karyotype evolution.
BMC Evolutionary Biology, 9: 84-108.
Koelher, U, Arnold, N,
Wienberg, J, Tofanelli, S and Stanyon, R, 1995, Genomic reorganizationand disrupted chromosomal synteny in the siamang
(Hylobates syndactylus) revealed by fluorescence insitu hybridization.
American Journal of Physical Anthropology, 97(1): 37-47.
Korstanje, R, O’brien, PCM,
Yang, F, Rens, W, Bosma A.A., Van Lith H.A., Van Zupthen L.F.M. And
Ferguson-Smith M.A., 1999 — Complete homology maps of the rabbit
(Oryctolagus cuniculus) and human by reciprocal chromosome painting.
Cytogenetics and Cell Genetics, 86: 317-322.
Kulemzina, AI, Trifonov,
VA, Perelman, PL, Rubtsova, NV, Volobuev, V, Ferguson-Smit, MA, Stanyon, R,
Yang, F and Graphodatsky, AS, 2009, Cross-species
chromosome painting in Cetartiodactyla: Reconstructing the karyotype evolution
in key phylogenetic lineage. Chromosome Research, 17: 419-436.
Li, T, O’brien, PCM,
Biltueva, L, Fu, B, Wang, J, Nie ,W, Ferguson-Smith, MA, Graphodatsky, AS and
Yang, F, 2004, Evolution of genome organizations of squirrels (Sciuridae) revealed by
cross-species chromosome painting. Chromosome Research, 12: 317-335.
Li, T, Wang, J, Su, W, Nie,
W, Yang, F, 2006, Karyotypic evolution of the family Sciuridae: inferences from
the genome organizations of ground squirrels. Cytogenet Genome Res 112:
270-276.
Ma, J, Zhang, L, Suh, BB,
Raney, BJ, Burthans, RC, Kent, WJ, Blanchette, M, Hauslerr, D and Miller, W,
2006 Reconstructing contiguous regions
of an ancestral genome. Genome Research, 16: 1557-1565.
Mao, X, Nie, W, Wang, J,
Su, W, Feng, Q, Wang, Y, Volleth, M and Yang, F, 2007, Karyotype evolution in Rhinolophus bats (Rhinolophidae, Chiroptera)
illuminated by cross-species chromosome painting and G-banding comparison.
Chromosome Research, 15: 835-847.
Mao, X, Nie, W, Wang, J,
Su, W, Feng, Q, Wang, Y, Volleth, M, Dobigny, G and Yang, F, 2008, Comparative cytogenetics of bats
(Chiroptera): The prevalence of Robertsonian translocations limits the power of
chromosomal characters in resolving interfamily phylogenetic relationships.
Chromosome Research, 16: 155-170.
Marzella, R, Viggiano, L,
Miolla, V, Storlazzi, CT, Ricco, A, Gentile, E, Roberto, R, Surace, C,
Fratello, A and Mancini, M, et al. 2000, Molecular cytogenetic resources for chromosome 4 and comparative
analysis of Phylogenetic Chromosome IV in Great Apes. Genomics, 63:
307–313.
Mikkelsen, Ts, Wakefield, Mj,
Aken, B, Amemiya,Ct, Chang, Jl, Duke, S, Garber, M, Gentles,Aj, Goodstadt, L,
Heger, A, Jurka, J, Kamal, M, Mauceli, E, Searle, Sm, Sharpe T, BakerMl, Batzer
Ma, Benos Pv, Belov K, Clamp M, Cook A, Cuff, J, Das, R, Davidow, L, Deakin,
Je, Fazzari, Mj, Glass, Jl, Grabherr, M, Greally, Jm, Gu, W, Hore, Ta, Huttley,
Ga, Kleber, M, Jirtle, Rl, Koina, E, Lee, Jt, Mahony, S, Marra, Ma, Miller, Rd,
Nicholls, Rd, Oda, M, Papenfuss, At, Parra, Ze, Pollock, Dd, Ray, Da, Schein,
Je, Speed, Tp, Thompson, K, Vandeberg, Jl, Wade, Cm, Walker, Ja, Waters, Pd,
Webber, C, Weidman, Jr, Xie, X, Zody, Mc; Broad Institute Genome Sequencing
Platform; Broad Institute Whole Genome Assembly Team, Graves JA, Ponting CP,
Breen M, Samollow PB, Lander ES, Lindblad-Toh K, 2007. Genome of the marsupial
Monodelphis domestica reveals innovation in noncoding sequences. Nature 447:
167–177.
Morescalchi, MA, Schempp,
W, Consigliere, S, Bigoni, F, Wienberg, J and Stanyon, R, 1997, Mapping
chromosomal homology between humans and the black-handed spider monkey by
fluorescence in situ hybridization. Chromosome Research, 5: 527-
536.
Moulin, S,
Gerbault-Seureau, M, Dutrilluaux, B and Richard, FA, 2008 Phylogenomics
of African guenons. Chromosome Research, 16 (5): 783-99.
Müller, S and Wienberg, J,
2001b, “Bar-coding” primate
chromosomes: molecular cytogenetic screening for the ancestral hominoid
karyotype. Human Genetics, 109 (1): 85-94.
Müller, S, Hollaz, M and
Wienberg J, 2003 Chromosomal phylogeny
and evolution of gibbons (Hylobatidae). Human Genetics, 113: 493-501.
Müller, S, Neusser, M,
O’Brien, PCM and Wienberg J, 2001a, Molecular
cytogenetic characterization of the
EBV-producing cell line B 95-8 (S. oedipus, Platyhrrini) by chromosome sorting
and painting. Chromosome Research, 9: 689-693.
Muller, S, O’Brien, PCM,
Ferguson-Smith, MA, Wienberg, J, 1997, Reciprocal chromosome painting between
human and prosimians ( Eulemur macaco macaco and E. fulvus mayottensis).
Cytogenet. Cell Genet. 78: 260-271.
Müller, S, Stanyon, R,
O’Brien, PCM, Ferguson-Smith, MA, Plesker, R and Wienberg, J, 1999, Defining the ancestral karyotype of all
primates by multidirectional chromosome painting between tree shrews, lemurs
and humans. Chromosoma,108: 393-400.
Murphy, W., Larkin, DM,
Everts-Van Der Wind, A, Bourque, G, Tesler, G, Auvil, L, Beever, JE, Chowdhary,
BP, Galibert, F and Gatze, L, et al. 2005, Dynamics of mammalian chromosome evolution inferred from multispecies
comparative maps. Science, 309: 613-617.
Murphy, WJ, Eizirik, E,
Johnson, WE, Zhang, YP, Ryder, OA and O’Brien, SJ, 2001a,, Molecular
phylogenetics and the origins of placental mammals. Nature 409: 614–618.
Nash, WG, Wienberg ,J,
Ferguson-Smith, MA, Menniner, JC And O’Brien, SJ, 1998, Comparative genomics: tracking chromosome evolution in the family
Ursidae using reciprocal chromosome painting. Cytogenetics and Cell
Genetics, 83: 182- 192.
Neusser, M, Stanyon, R,
Bigoni, F, Wienberg, J and Muller, S, 2001, Molecular cytotaxonomy of New World monkeys (Platyrrhini) a comparative
analysis of five species by multi-color chromosome painting gives evidence for
a classification of Callimico goeldii within the family of Callitrichidae.
Cytogenetics and Cell Genetics, 94: 206-215.
Nie, W, Wang, J, Weiting, Su, Wang, Y, Yang, F,
2009, Chromosomal rearrangements underlying karyotype differences between
Chinese pangolin (Manis pentadactyla) and Malayan pangolin (Manis javanica)
revealed by chromosome painting.
Chromosome Research 17:321–329
Nie, W, Fu, B, O’Brien PCM,
Wang, J, Su, W, Tanomtong, A, Volobouev, V, Ferguson-Smith, MA and Yang, F,
2008, Flying lemurs – The
‘flying tree shrews’? Molecular cytogenetic evidence for a
Scandentia-Dermoptera sister clade. BMC Biology, 6: 18-28.
Nie, W, Liu, R, Chen, Y,
Wang, J and Yang, F, 1998, Mapping
chromosomal homologies between humans and two langurs (Semnopithecus francoisi
and S. phayrei) by chromosome painting. Chromosome Research, 6
(6): 447-453.
Nie, W, O’Brien, PCM, Fu,
B, Wang ,J, Su, W, Ferguson-Smith, MA, Robinson, TJ and Yang, F, 2006, Chromosome painting between human and
lorisiform prosimians: evidence for the HSA 7/16 synteny in the primate
ancestral karyotype. American Journal of Physical Anthropology, 129 (2):
250-259.
Nie, W, Rens, W, Wang, J
and Yang, F, 2001, Conserved
chromosome segments in Hylobates hoolock revealed by human and H. leucogenys
paint probes. Cytogenetics and Cell Genetics, 92(3-4):248-53.
Nie, W, Wang, J, O_Brien,
PCM et al, 2002, The genome phylogeny of domestic cat, red panda and five
mustelid species revealed by comparative chromosome painting and G-banding.
Chromosome Res 10: 209-222.
Pardini, AT, O'Brien, PCM, Fu, B, Bonde, RK, Elder
FFB, Ferguson-Smith MA, Yang F and Robinson T.J, 2007, Chromosome
painting among Proboscidea, Hyracoidea and Sirenia: support for Paenungulata
(Afrotheria, Mammalia) but not Tethytheria. Proc. R. Soc. B 274,
1333–1340.
Perelman, PL, Graphodatsky,
AS, Dragoo, JW,. Serdyukova,
NA,. Stone, G, Cavagna, P.,. Menotti, A, W. Nie,. O_Brien P. C. M , Wan, J,
Burkett, GS, Yuki, K, ,Roelke, ME, O_Brien, SJ, Yang, F. and Stanyon R, 2008,
Chromosome painting shows that skunks (Mephitidae, Carnivora) have highly
rearranged karyotypes. Chromosome Research 16:1215–1231
Picone, B and Sineo, L, 2010, Reconstructing the
Phylogeny of the Human Chromosome 4 Synteny using Comparative Karyology and
Genomic Data Analysis. Caryologia Vol. 63, no. 3: 314-334.
Richard, F, Lombard, M and
Dutrillaux B, 2000, Phylogenetic
origin of human chromosomes 7, 16, and 19 and their homologs in placental
mammals. Genome Research, 10(5): 644-651.
Richard, F, Lombard, M and
Dutrillaux, B, 1996, ZOO-FISH suggests
a complete homology between human and capuchin monkey (Platyrrhini) euchromatin.
Genomics, 36: 417-423.
Richard, F, Lombard, M and
Dutrillaux, B, 2003a, Reconstruction
of the ancestral karyotype of eutherian mammals. Chromosome Research,
11: 605-618.
Richard, F, Messaoudi, C,
Bonnet-Garnier, A, Lombard, M and Dutrillaux B, 2003b, Highly
conserved chromosomes in an Asian squirrel (Menetes berdmorei, Rodentia:
Sciuridae) as demonstrated by ZOO-FISH with human probes. Chromosome
Research, 11: 597-603.
Richard, F, Messaoudi, C,
Lombard, M and Dutrillaux, B, 2001. Chromosome
homologies between man and mountain zebra (Equus zebra hartmannae) and
description of a new ancestral synteny involving sequences homologous to human
chromosome 4 and 8. Cytogenetics and Cell Genetics, 93: 291-296.
Robinson, TJ and
Ruiz-Herrera, A, 2008, Defining the ancestral eutherian karyotype:
A cladistic interpretation of chromosome painting and genome sequence assembly
data. Chromosome Research, 16: 1133-1141.
Robinson, TJ, Fu, B,
Ferguson-Smith, MA and Yang, F, 2004, Cross-species chromosome painting in the
golden mole and elephant shrew: support for the mammalian clades Afrotheria and
Afroinsectiphillia but not Afroinsectivora. Proceedings of the Royal
Society of London B, Biological Sciences, 271 (1547): 1477-1484.
Romanenko, Sa, Volobouev,
Vt, Perelman, PL, Lebedev, V, Serdukova N, Trifonov, V, Biltueva L, Nie, W,
O_Brien, PCM., Bulatova, N, Ferguson-Smith, MA, Yang, F, Graphodatsky, AS, 2007, Karyotype evolution
and phylogenetic relationships of hamsters (Cricetidae, Muroidea, Rodentia)
inferred from chromosomal painting and banding comparative genomics. Chromosome
Research (2007) 15:283-297.
Ruiz-Herrera, A,
Castresana, J and Robinson TJ, 2006, Is
mammalian chromosomal evolution driven by regions of genome fragility? Genome
Biology, 7 (12): 115-131.
Ruiz-Herrera, A, Garcia, F,
Aguilera, M, Garcia, M and Ponsa’, FM, 2005,
Comparative chromosome painting
in Aotus reveals a highly derived evolution. American Journal of
Primatology, 65: 173-185.
Ruiz-Herrera, A, Ponsà, M,
Garcia, F, Egozcue, J, Garcia, M, 2002a, Fragile sites in human and Macaca
fascicularis chromosomes are breakpoints in chromosome evolution. Chromosome
Res, 10: 33-44.
Rumpler, Y, Warter, S,
Hauwy, M, Fausser, JL, Roos, C and Zinner, D, 2008, Comparing
chromosomal and mitochondrial phylogenies of sportive lemurs (genus Lepilemur,
Primates). Chromosome Research, 16: 1143-1158.
Scherthan H, Cremer T,
Arnasson U, Weier HU, Lima-De-Faria A and Fronicke L, 1994, Comparative chromosome painting studies
discloses homologous segments in distantly related mammals. Nature
Genetics, 6: 342-347.
Serreau, M. Bonnet-Garnier,
A, Richard, F and Dutrillaux, B, 2004, Chromosome
painting comparison of Leontopithecus chrysomelas (Callitrichinae, Platyrrhini)
with man and its phylogenetic position. Chromosome Research, 12(7): 691-
701.
Stanyon R, Garofalo F,
Steinberg ER, Capozzi O, Di Marco S,
Nieves M, Archidiacono N, Mudry MD, 2011, Chromosome Painting in Two Genera of
South American Monkeys: Species Identification, Conservation, and Management.
Cytogenet Genome Res, DOI: 10.1159/000324415
Stanyon R, Bigoni F, Slaby
T, Muller S, Stone G, Bonvicino CR, Neusser M and Seuanez HN, 2004, Multi-directional chromosome painting maps
homologies between species belonging to three genera of New World monkeys and
humans. Chromosoma, 113: 305-315.
Stanyon R, Consigliere S,
Muller S, Morescalchi A, Neusser M and Wienberg J, 2000, Fluorescence in situ hybridization (FISH)
maps chromosomal homologies between the dusky titi and squirrel monkey.
American Journal of Primatololgy, 50: 95-107.
Stanyon R, Dumas F, Stone G
and Bigoni F, 2006, Multidirectional chromosome painting reveals
a remarkable syntenic homology between the greater galagos and the slow loris.
American Journal of Primatology, 68: 349-359.
Stanyon R, Yang F,
Morescalchi Am, Galleni L, 2004, Chromosome painting in the long-tailed field
mouse provides insights into the ancestral murid karyotype. Cytogenet Genome
Res 105: 406-411.
Stanyon, R, Bonvicino, CR,
Svartman, M and Seuanez, HN, 2003, Chromosome
painting in Callicebus lugens, the species with the lowest diploid number
(2n=16) known in primates. Chromosoma, 112: 201-206.
Stanyon, R, Bruening, R,
Stone, G, Shearin, A and Bigoni, F, 2005, Reciprocal painting between humans, De Brazza’s and patas monkeys
reveals a major bifurcation in the Cercopithecini phylogenetic tree.
Cytogenetic and Genome Research, 108: 175-182.
Stanyon, R, Koelher, U and
Consigliere, S, 2002, Chromosome
painting reveals that galagos have highly derived karyotypes. American
Journal of Physical Anthropology, 117: 319-326.
Stanyon, R, Rocchi, M,
Capozzi, O, Roberto, R, Misceo, D, Ventur,a M, Cardone, MF, Bigoni, F and
Archidiacono, N, 2008, Primate
chromosome evolution: Ancestral karyotypes, marker order and neocentromeres.
Chromosome Research, 16:17-39.
Stanyon, R, Stone, G,
Garcia, M, Froenicke, L, 2003. Reciprocal chromosome painting shows that squirrels,
unlike murid rodents, have a highly conserved genome organization. Genomics 82:
245–249
Stanyon, R. Consigliere, S,
Bigoni, F, Ferguson-smith, M, O’Brien, PCM and Wienberg, J, 2001, Reciprocal chromosome painting between a New
World primate, the woolly monkey, and humans. Chromosome Research, 9:
97-106.
Svartman, M, Stone, G, and
Stanyon, R, 2006, The ancestral eutherian karyotype is present in Xenarthra.
PLoS Genet 2(7):e109.
Svartman, M, Stone, G,
Page, JE and Stanyon, R, 2004, A
chromosome painting test of the basal eutherian karyotype. Chromosome
Research, 12: 45-53.
TianY, Nie W, Wang J,
Ferguson-Smith MA and Yang F, 2004, Chromosome
evolution in bears: reconstructing phylogenetic relationships by cross species
chromosome painting. Chromosome Research, 12:55-63.
Trifonov, VA, Stanyon, R,
Nesterenko, AI, Fu, B, Perelman, PL, O’Brien, PCM, Stone, G, Runtsova NV, Houck
ML and Robinson, TJ, et al. 2008, Multidirectional
cross-species painting illuminates the history of karyotypic evolution in
Perissodactyla. Chromosome Research, 16: 89- 107.
Volleth M And Müller S,
2006, Zoo-FISH in the European mole
(Talpa europaea) detects all ancestral Boreo-Eutherian human homologous
chromosome associations. Cytogenetic and Genome Research, 115: 154-157.
Volleth M, Klett C, Kollak
A, Dixkens C, Wintery, Just W, Vogel W, and Hamaister, H,, 1999, ZOO-FISH
analysis in a species of the order Chiroptera: Glossophaga soricina
(Phyllostomidae). Chromosome Research, 7: 57-64.
Volleth, M, Heller, KG,
Pfeiffer, RA and Hamasteir, H, 2002, A
comparative ZOO-FISH analysis in bats elucidates the phylogenetic relationships
between Megachiroptera and five microchiroptera families. Chromosome
Research, 10: 477-497.
Volleth, M, Klett, C,
Kollak, A, Dixkens, C, Winter, Y, Just, W, Vogel, W and Hamaister H, 1999. ZOO-FISH
analysis in a species of the order Chiroptera: Glossophaga soricina
(Phyllostomidae). Chromosome Research, 7: 57-64.
Walker, FO, 2007,
“Huntington’s disease”. Lancet,369 (9557): 218-228.
Warter, S, Hauwy, M,
Dutrillaux, B and Rumpler, Y, 2005, Application
of molecular cytogenetics for chromosomal evolution of the Lemuriformes (Prosimians). Cytogenetic and
Genome Research, 108: 197-203.
Wienberg, J, 2004, The evolution of eutherian chromosomes.
Current Opinion in Genetics and Development, 14: 657-666.
Wienberg, J, 2005, Fluorescence in situ hybridization to
chromosomes as a tool to understand human and primate genome evolution. Cytogenetic
and Genome Research, 108: 139-160.
Wienberg, J, and Stanyon,
R, 1995, "Chromosome painting in mammals as an approach to
Wienberg, J, Stanyon, R,
Jauch, A and Cremer, T, 1992, Homologies
in human and Macaca fuscata chromosomes revealed by in situ suppression
hybridization with human chromosome-specifi c DNA libraries. Chromosoma,
101: 265-270.
Wienberg, J, Stanyon, R,
Nash, WG, O’Brien, PCM, Yang, F, O’Brien, SJ and Ferguson-smith, MA, 1997, Conservation
of human vs. feline genome organization revealed by reciprocal chromosome
painting. Cytogenetics and Cell Genetics, 77: 211-217.
Wright, TJ et al. (1997) A
transcript map of the newly defined 165 kb Wolf–Hirschhorn
syndromecritical region.Hum.Mol.Genet. 6, 317–324
Yang, F, Alkalaeva, EZ,
Perelman, PL, Pardini, AT, Harrison, WR, O’Brien, PCM, Fu, B, Graphodatsky, AS,
Ferguson-Smith, M.A. and Robinson, TJ, 2003, Reciprocal chromosome painting among human, aardvark, and elephant
(superorder Afrotheria) reveals the likely eutherian ancestral karyotype. Proceedings
of the National Academy
of Sciences of the United States of America, 100: 1062-1066.
Yang, F, Fu, B, O’Brien,
PCM, Nie, W, Ryder, OA and Ferguson-Smith, MA, 2004. Refined genome- wide comparative map of the domestic horse, donkey and
human based on cross-species chromosome painting: insight into the occasional
fertility of mules. Chromosome Research, 12: 65-76.
Yang, F, Graphodatsky, AS,
Li, T, Fu, B, Dobigny, G, Wang, J, Perelman, PL, Serdukova, NA, Su, W and
O’Brien, PCM, et al. 2006, Comparative
genome maps of the pangolin, hedgehog, sloth, anteater and human revealed by
cross-species chromosome painting: further insight into the ancestral karyotype
and genome evolution of eutherian mammals. Chromosome Research, 14:
283-296.
Yang, F, Graphodatsky, AS,
O’Brien, PCM, Colabella, A, Solanky, N, Squire, M, Sargan, DR and Ferguson-Smith,
MA, 2000, Reciprocal chromosome painting illuminates the history of genome
evolution of the domestic cat, dog and human. Chromosome Research, 8:
393-404.
Yang, F, Muller, S, Just,
R, Ferguson-Smith, MA and Wienberg J, 1997a, Comparative chromosome painting in mammals: Human and the Indian
muntjac (Muntiacus muntjak vaginalis). Genomics, 39 (3): 396-401.
Yang, F, O’Brien, PCM,
Graphodatsky, AS, Solanky, N, Trifonov,
VA, Rens, W, Sargan, D, Ferguson-Smith,
MA.1999. A Complete Comparative Chromosome Map for the Dog, Red Fox, and Human
and Its Integration with Canine Genetic Maps. Genomics 62, 189–202.
Ye, J, Biltueva, L, Huang,
L, Nie, W, Wang, J, Jing, M, Su, W, Vorobieva, NV, Jiang, X and Graphodatsky,
AS, et al. 2006, Cross-species
chromosome painting unveils cytogenetic signatures for the Eulipotyphla
and evidence for the polyphyly of Insectivora. Chromosome Research, 14:
151-15.
Zollino M, Lecce R,
Selicorni A, Murdolo M, Mancuso I, Marangi G, Zampino G, Garavelli L, Ferrarini
A, and Rocchi M,., et al. 2004, A
double cryptic chromosome imbalance is an important factor to explain
phenotypic variability in Wolf-Hirschhorn syndrome European. Journal of
Human Genetics, 12: 797-804.