
Dumas, F., Bigoni, F.,
2010, Filogenomica dei Primati: evidenze dalla citogenetica molecolare.
Antropo, 23, 1-10. www.didac.ehu.es/antropo
Filogenomica dei Primati: evidenze dalla citogenetica molecolare
Phylogenomics in Primates: evidence from Molecular cytogenetics
F.
Dumas1, F. Bigoni2
1Dipartimento
di Biologia Animale “G. Reverberi”, via
Archirafi 18, 90123 Palermo, Italy. fdumas@unipa.it
2 Dipartimento di Biologia
Evoluzionistica “Leo Pardi”, via del Proconsolo 12, 50122 Firenze, Italy. francesca.bigoni@unifi.it
Parole chiave: filogenesi,
evoluzione, genomi, cromosomi, mammiferi
Keywords: phylogeny, evolution, genomes, chromosomes, mammals
Riassunto
Il cariotipo
ancestrale dei Primati (Proscimmie, scimmie del Nuovo Mondo, scimmie del Vecchio
mondo, scimmie antropomorfe e Homo) e più di recente anche di tutti i Mammiferi
euteri è stato ricostruito mediante la citogenetica molecolare. L’ibridazione
fluorescente in situ di sonde cromosomiche “painting” permette di individuare
le omologie cromosomiche tra specie a confronto e i riarrangiamenti
intercromosomici (traslocazioni) che si verificano nel corso dell’evoluzione
genomica. E’ stato dimostrato con
la tecnica “painting” che i genomi generalmente sono altamente conservati e
costituiti da pochi segmenti cromosomici di grandi dimensioni. I
riarrangiamenti di questi segmenti in diverse combinazioni spiegano la
diversità riscontrata nei diversi cariotipi. L’analisi cladistica e
parsimoniosa delle associazioni sinteniche conservate e derivate condivise permette lo studio delle
relazioni di parentela tra le specie. L’approccio della citogenetica consente
inoltre lo studio dell’evoluzione dei cromosomi umani negli ultimi 90 milioni
di anni.
Abstract
The ancestral karyotype of Primates (Prosimians, New Word Monkeys, Old
Word Monkeys, apes and humans), and later, that of all eutherian mammals has
been reconstructed using molecular cytogenetics. Fluorescence In Situ Hybridization (FISH) using probes specific to whole single chromosomes
(chromosome paints) allows the mapping of chromosomal homology between species
and reveals the interchromosomal rearrangements (translocations) that occurred
during genome evolution. Chromosome painting shows that genomes are generally
conserved in a limited number of large chromosomal segments. Rearrangements of
these segments explain much of the diversity that exists between the karyotypes
of various species. A Cladistic analysis using parsimony of chromosomal
syntenies and associations can provide valuable information for determining the
evolutionary, phylogenetic relationships of species. Furthermore, the
cytogenetic approach allows us to trace the evolutionary history of each human
chromosome over the last 90 million years.
Evoluzione genomica e filogenesi
La Teoria
dell’Evoluzione di Charles Darwin
afferma che le forme di vita presenti su questo pianeta sono imparentate
tra loro, in quanto tutte discendono da antenati comuni. Per esempio, gli
esseri umani e gli scimpanzé, derivano dalla divergenza da un comune antenato
avvenuta circa 6 milioni di anni fa, invece cani ed esseri umani hanno avuto un
antenato comune probabilmente oltre i 90 milioni di anni fa. Allontanandoci
ancor più nel tempo, l’uomo e l’opossum condividono un antenato comune che
risale a più di 150 milioni di anni fa.
In passato, lo studio dell’evoluzione si è basato principalmente
sull’anatomia comparata e sulle scoperte della paleontologia, ma durante gli
ultimi cinquanta anni sono stati raggiunti grandi risultati, in questo ambito,
grazie alla genomica comparata. Questo tipo di indagine ha fornito dati nuovi
essenziali per ricostruire i processi evolutivi che hanno dato origine ai
nostri parenti più prossimi e alla nostra stessa specie, Homo sapiens.
La genomica
comparata permette infatti la ricostruzione del genoma ancestrale di gruppi di
specie imparentate per ciascun nodo principale di ramificazione dell’albero
della vita. Tale ricostruzione consente da un lato l’analisi della relazione di
parentela tra le specie (filogenesi) e dall’altro la valutazione dei passaggi
che hanno determinato la forma attuale dei cromosomi umani a partire dal
cariotipo ancestrale di tutti i Mammiferi (Ferguson-Smith and Trifonov, 2007,
Stanyon et al., 2008)
La Citogenetica Comparata
Tra gli approcci
della genomica comparata quello della citogenetica è il primo ad essere stato
applicato nello studio del genoma dei Primati. I primi studi di Citogenetica
comparata utilizzarono la tecnica della colorazione di interi cromosomi che
permise una valutazione delle differenze cromosomiche a livello morfologico
(Chiarelli, 1963). Le ipotesi relative ai genomi ancestrali venivano formulate
in termini di numero diploide (2n) e di numero di braccia cromosomiche (Numero
Fondamentale o FN). Successivamente con l’introduzione dei metodi di
colorazione differenziata (bandeggio cromosomico) è stato possibile distinguere
ed appaiare i cromosomi omologhi. Mediante l’analisi dei cromosomi bandeggiati
è stato possibile dimostrare che i cromosomi dei Primati e dei Mammiferi si
sono in larga parte conservati durante il processo evolutivo (Dutrillaux 1979,
Yunis and Prakash 1982, Nash and O’Brien 1982).
Citogenetica Molecolare
A partire dagli
anni ‘90 (Wienberg et al., 1990) i cariotipi appartenenti a specie diverse di
Mammiferi sono stati comparati a livello molecolare e messi in relazione tra di
loro mediante la tecnica della Citogenetica Molecolare Comparativa nota come
“chromosome painting” che prevede l’ibridazione in situ Fluorescente
(FISH-Fluorescence In Situ Hybridization) di sonde cromosomiche umane
(Fig.1).
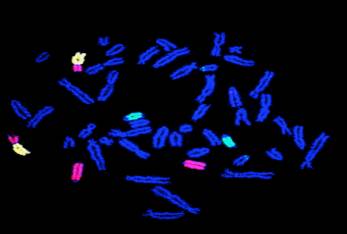
Figura
1. Esempio di FISH con sonde cromosomiche
“painting” umane su una metafase
di scimmia Platyrrhina (Callicebus cupreus): sonda “paint” 17 in verde , sonda 8 in rosso e 18 in giallo.
Figure 1.
Example of FISH with Human chromosome painting probes on a Platyrrhini
metaphase (Callicebus cupreus): painting Probe 17-green, probe 8-red, probe 18
yellow.
La tecnica FISH
consiste nell’ibridare sonde di DNA marcate con una sostanza fluorescente su
DNA di una metafase “target” e sfrutta la complementarietà delle basi azotate
della sonda e del preparato cromosomico, permettendo di stabilire le omologie
cromosomiche tra specie diverse. Il “chromosome painting” consente di
individuare le omologie a livello di interi cromosomi o parti di essi e i
riarrangiamenti intercromosomici (traslocazioni, fissioni e fusioni) che
intervengono durante il processo evolutivo. Con il painting cromosomico è
possibile, quindi, determinare quali cromosomi, segmenti di cromosoma o
sintenie (e quindi anche la localizzazione di due o più geni su uno stesso
cromosoma) siano stati conservati tra i genomi di due o più specie messe a
confronto. Permette inoltre di determinare quanti riarrangiamenti cromosomici
sono stati necessari per trasformare il genoma di una specie in quello di
un’altra. Negli ultimi anni sono state utilizzate anche sonde sub regionali o
locus specifiche (Fig.2) prodotte mediante microdissezione o per clonaggio di
DNA all’interno di vettori.
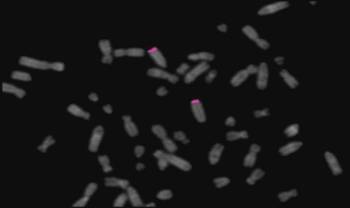
Figura
2. Esempio di FISH con sonda locus specifica WolfHirshorn (4p.11) su una metafase di
una Platyrrhina (Lagothrix lagotricha).
Figure 2.
Example of FISH with the single
locus probe WolfHirshorn (4p.11) on a Platyrrhini metaphase (Lagothrix
lagotricha).
Queste sonde,
caratterizzate da elevato potere risolutivo, permettono di individuare i
riarrangiamenti intracromosomici che non sono rivelabili mediante painting. La
tecnica FISH con sonde di DNA clonate in BACs (Bacterial Artificial
Chromosomes) o in YACs (Artificial Yeast Chromosomes) permette di identificare,
infatti, i riarrangiamenti intracromosomici (inversioni e lo spostamento del
centromero) e i punti di rottura cromosomici con un livello di risoluzione
elevato (100 kb), inferiore solo alla caratterizzazione mediante
sequenziamento.
Analisi Cladistica dei dati sui cromosomi
Nelle
ricostruzioni filogenetiche ottenute mediante la citogenetica viene adottato un
approccio cladistico e parsimonioso al fine di individuare le sintenie
cromosomiche ancestrali e le nuove associazioni cromosomiche formatesi a
seguito dei riarrangiamenti
(Wienberg and Stanyon, 1995). Per distinguere i caratteri conservati da quelli
derivati si ricorre al confronto con una specie affine, ma esterna al gruppo
considerato (outgroup). Secondo il principio di parsimonia tra le varie
interpretazioni possibili di un fenomeno (organizzazione cromosomica) si
preferisce quella che comporta il minor numero di passaggi ipotizzabili. I
riarrangiamenti cromosomici determinano una riorganizzazione genomica formando
nuove associazioni sinteniche costituite dalla diversa combinazione degli
omologhi dei cromosomi umani. Quando una sintenia cromosomica viene ritrovata
intatta in diverse specie, questa può essere interpretata come condizione
ancestrale. Per esempio la presenza di una sintenia cromosomica uguale in
ordini di Mammiferi molto divergenti tra loro, suggerisce la sua presenza nel
cariotipo ancestrale di tutti i Mammiferi. I riarrangiamenti cromosomici che si
fissano nel cariotipo di una specie rappresentano degli eventi rari e
strettamente legati al processo di speciazione. Il basso tasso di
riarrangiamenti intercromosomici (circa due per ogni 10 milioni di anni)
permette una rapida distinzione delle omologie tra le varie specie. Poiché i
riarrangiamenti sono eventi rari, i tratti derivati comuni tra due specie
(nuove associazioni sinteniche) sono utili per le ricostruzioni filogenetiche.
Gli studi di “painting” cromosomico sono stati effettuati inizialmente su
specie di Primati molto vicine tra loro, utilizzando sonde cromosomiche umane
per analizzare i cromosomi di scimmie antropomorfe, scimmie del Vecchio Mondo
(Catarrhini), scimmie del Nuovo Mondo (Platyrrhini) e Proscimmie
(Strepsirrhini) ed in seguito specie di Mammiferi più distanti da un punto di
vista filogenetico (Wienberg et al.
1990, 1992; Stanyon et al., 1992; Jauch et al. 1992;
Scherthan et al., 1994; Wienberg e Stanyon 1995, 1997, 1998).
Uso del Flow sorting per generare “chromosome paints”
Mediante la
tecnica del “flow sorting” che
permette di discriminare i cromosomi in base alle loro dimensioni e contenuto
di basi azotate è possibile ottenere oggi oltre che sonde cromosomiche umane,
anche sonde “painting” provenienti da altre specie di Mammiferi utili in
esperimenti di painting cromosomico reciproco (Stanyon and Stone, 2008).
Mediante tale tecnica i cromosomi mitotici in sospensione colorati con due
fluorocromi che hanno una diversa specificità per le coppie di basi azotate,
vengono introdotti all’interno di un tubicino che permette il passaggio di un
cromosoma alla volta. L’intensità della fluorescenza di ciascuno delle
centinaia di cromosomi viene misurata quando essi sono colpiti da due raggi
laser. L’intensità viene rivelata in “peaks” distinti in un plotter ed è così
possibile discriminare e deflettere i diversi cromosomi in differenti
sospensioni. Successivamente con un primo giro di PCR si amplificano
direttamente i cromosomi “sorteggiati”, e con una reazione secondaria di PCR si
marcano i prodotti primari.
Il “painting”
cromosomico reciproco (Zoo-FISH) consiste nell’ibridare sonde umane sulle
metafasi di una specie e le sonde della stessa specie su metafasi umane. Questa
tecnica permette di analizzare le omologie cromosomiche in due esperimenti
indipendenti di FISH e di individuare inoltre i punti di rottura coinvolti
durante l’evoluzione cromosomica (Dumas et al., 2005, 2007). Questa tecnica
reciproca di Zoo-FISH è stata utilizzata per la comparazione di sintenie
cromosomiche nei Mammiferi
placentati (Wienberg and Stanyon,
1997; O’Brien et al., 1999; Murphy et al., 2001).
Partendo dalla
ricostruzione del cariotipo ancestrale di Mammiferi placentati, è possibile,
inoltre, determinare i passi più importanti che negli ultimi 90 milioni di anni
hanno portato alla formazione dei cromosomi umani (Stanyon et al., 2008).
L’utilizzo di
sonde del tipo “chromosome paint” presenta tuttavia alcuni limiti. Le sonde
“paint” sono eccellenti nell’identificare i riarrangiamenti intercromosomici
(traslocazioni, fissioni e fusioni), ma non lo sono altrettanto nell’individuare
i cambiamenti intracromosomici come le inversioni e le duplicazioni. Inoltre il
livello di risoluzione del “painting” è compreso tra 5 e 10 Mb per cui mediante
questo approccio sfuggono i cambiamenti intercromosomici molto piccoli. Tale
limite viene superato, come già detto, mediante il mappaggio di sonde piccole
di DNA clonate all’interno di vettori (“Bacterial Artificial Chromosome”-BACs)
e di sonde locus specifiche (Stanyon et al., 2008).
In ultimo è
dimostrato che il “painting” cromosomico tra Marsupiali e Mammiferi placentati,
non ha avuto mai successo, e come conseguenza la mancanza di un “outgroup”
appropriato per i Mammiferi Euteri ha precluso il raggiungimento di conclusioni
stabili riguardo il contenuto del genoma ancestrale ricostruito fin oggi
(Svartman et al., 2004). Tuttavia, recentemente si è ottenuta la sequenza del
genoma del marsupiale Monodelphis domestica, l’opossum, (Mikkelsen
et al., 2007) attraverso cui è stato possibile convalidare le ipotesi
proposte mediante painting.
Evoluzione cromosomica nei Mammiferi
L’evoluzione del
cariotipo nei Mammiferi è stato un processo continuo nel corso degli ultimi 100
milioni di anni, durante i quali si sono verificate divergenze rispetto al
comune cariotipo ancestrale. Successivi eventi mutazionali hanno causato la
riorganizzazione della struttura dei cromosomi e del loro numero all’interno
dei cariotipi.
Nelle
fasi iniziali della citogenetica classica (colorazione uniforme o bandeggio dei
cromosomi) alcuni ricercatori hanno proposto che il cariotipo ancestrale dei
Mammiferi avesse un numero diploide alto, e che l’evoluzione abbia fatto il suo
corso attraverso fusioni cromosomiche. Altri studiosi hanno proposto che le
fissioni fossero invece il principale meccanismo evolutivo, e quindi hanno prospettato
numeri diploidi bassi; altri ancora hanno mantenuto una posizione intermedia
proponendo che entrambi i meccanismi fossero importanti (cf. Stanyon et al.,
2002). Ora sappiamo che ciò è corretto e che il numero diploide ancestrale dei
mammiferi placentati è intermedio.
Conservazione del genoma
I risultati del
“painting” cromosomico dimostrano un alto livello di conservazione dei
cromosomi in tutti gli ordini di Mammiferi placentati, anche se alcuni cladi
sono caratterizzati da una rapida evoluzione cromosomica e presentano quindi
dei genomi altamente riarrangiati. Per esempio l’evoluzione del cariotipo dei
roditori, degli equidi, dei gibboni (Muller et al., 2003) è avvenuto con un
alto tasso evolutivo mediante riarrangiamenti sia intracromosomici che intercromosomici
(Romanenko, S.2007, Yang, F. et al., 2004). Al contrario il processo evolutivo
è stato relativamente conservativo in gatti, foche (Froenicke et al., 1997) e cetacei (Bielec et al., 1998).
Sintenie ed associazioni cromosomiche
Nei Mammiferi
durante l’evoluzione alcune sintenie del cariotipo ancestrale si sono
conservate mentre altre nuove associazioni sinteniche sono state formate a
causa dei riarrangiamenti (cf. Ferguson-Smith e Trifonov, 2007). Attraverso il
painting cromosomico è stato dimostrato che alcuni riarrangiamenti e nuove
associazioni sinteniche sono marker filogenetici per alcuni cladi e che gli
omologhi di molti cromosomi umani sono intatti nei Mammiferi placentati. I
cromosomi 13 e 17 per esempio sono conservati e da considerarsi sintenie
presenti nel cariotipo ancestrale dei Mammiferi, altri omologhi dei cromosomi
umani sono conservati nella maggior parte dei Mammiferi studiati e presenti su
un cromosoma, ma in associazione con altre sintenie: 3/21, 14/15, 7/16, 16/19,
due cromosomi con omologia 12/22 e l’associazione 4/8. Quest’ultima
associazione è presente in tutti gli ordini dei Mammiferi euteri eccetto che
nei Primati e certi specie di Afrotheria (Kellogg et al., 2007). Tuttavia è
stato dimostrato mediante sequenziamento che l’associazione 4/8 è presente
nell’outgroup dei mammiferi placentati, l’opossum, per cui questa associazione
è inserita nel cariotipo ancestrale dei Mammiferi. Una delle due associazioni
sinteniche ancestrali degli omologhi dei cromosomi umani 12/22, e ulteriormente
associata con un segmento dell’omologo del cromosoma umano 10, è stata
osservata nei carnivori, nei cetartiodattili, nel cavallo, nel pipistrello ed
anche nell’opossum a conferma che essa è presente nel cariotipo ancestrale di
tutti i Mammiferi. L’associazione 1/19 è presente nei Mammiferi (Yang et al,
2003; Svartman et al, 2004, 2006) ma non è omologa a quella presente
nell’opossum. Pertanto tale associazione non è inserita nel cariotipo
ancestrale dei Mammiferi (Stanyon et al., 2008).
Evoluzione cromosomica nei Primati
Negli ultimi
anni approssimativamente 50 specie di Primati sono state analizzate attraverso
le tecniche di citogenetica molecolare, fornendo una descrizione sulle
dinamiche dei cambiamenti genomici avvenuti durante l’evoluzione umana e quella
dei nostri parenti più vicini. Le
specie di Primati studiate appartengono alle Proscimmie, alle scimmie del Nuovo
Mondo e del Vecchio Mondo e Homo sapiens. Il genoma del comune
antenato di tutti i Primati, definito facendo riferimento all’omologia con il
cariotipo umano, ha numero diploide di 2n=50 (Stanyon et al., 2008) con i
seguenti cromosomi 1, 2a, 2b, 3/21, 4, 5, 6, 7a, 7b/16b, 8, 9, 10a, 10b, 11,
12a/22a, 12b/22b, 13, 14/15, 16a, 17, 18, 19a, 19b, 20, X ed Y (Fig.3).
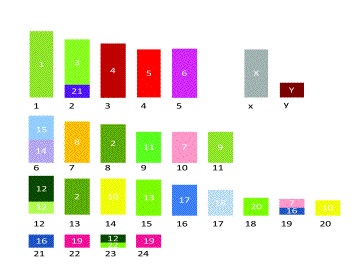
Figura
3.
Ipotetico Cariotipo ancestrale di tutti i Primati con numero diploide 2n=50
costruito mediante “painting” cromosomico comparativo. Ogni cromosoma è
rappresentato da un rettangolo, Il
numero all’interno indica l’omologia con i cromosomi umani. Ogni sintenia
cromosomica umana è indicata con un colore diverso.
Figure 3. Hypothetical ancestral primate karyotype with diploid number 2n=50 obtained through comparative chromosome painting. Every chromosomes is represented by a rectangle, the inside number represents the homology with human chromosomes. Each human chromosomal synteny is indicate by a different color.
Strepsirrhini
Un gruppo di
Primati che è stato analizzato attraverso la tecnica FISH è quello delle
proscimmie. Sia i caratteri morfologici che i cariotipi delle proscimmie
ottenuti mediante bandeggio cromosomico, erano in passato considerati primitivi
e quindi più simili all’antenato di tutti Primati (Dutrillaux, 1979). I dati
ottenuti attraverso il painting cromosomico, tuttavia, indicano la situazione
opposta, in quanto sono stati dimostrati molti riarrangiamenti e sintenie
cromosomiche derivate. Queste specie non possono quindi essere considerate
vicine al cariotipo ancestrale dei Primati (Stanyon et al., 2006).
Anthropoidea
Il cariotipo
ancestrale delle Anthropoidea (scimmie del Vecchio Mondo e scimmie del Nuovo
Mondo) ha un numero diploide di 2n=50. Il cariotipo di tutte le specie sono
marcate da un punto di vista filogenetico dalla fissione della sintenia
ancestrale 7/16, dalla traslocazione reciproca che diede origine ai cromosomi
12 e 22 e dalla fusione dei segmenti 19a e 19b.
Le Scimmie del Nuovo Mondo o Platyrrhini
Le Scimmie del
Nuovo Mondo sono state ampiamente studiate attraverso la tecnica FISH che ha
permesso di dimostrare che i riarrangiamenti cromosomici sono markers utili
nelle ricostruzioni filogenetiche e
nella comprensione della tassonomia delle scimmie del Nuovo Mondo
(Neusser et al., 2001). Le scimmie del Nuovo Mondo presentano un’ampia gamma di
modificazioni morfologiche ed etologiche, per cui risulta difficile
identificare omologie a questo livello di studio. E’ stato dimostrato mediante
painting che il numero di specie riconosciute è sottostimato in quanto esistono
specie identiche da un punto di vista morfologico, ma diverse dal punto di
vista genomico. Per esempio, la scimmia gufo (Aotus trivirgatus), noto modello
animale per diverse malattie umane, mostra almeno 12 differenti cariotipi
all’interno della sua area di distribuzione geografica; la stessa situazione si
verifica per le scimmie urlatrici del genere Alouatta (Consigliere et
al. 1998) e per il genere Callicebus (Dumas et al., 2005).
Il painting cromosomico supporta la monofilia dei Primati del Nuovo Mondo:
tutti condividono, infatti, le associazioni sinteniche 8/18, 10/16, 2/16, 7/5
(Neusser et al., 2001, Dumas et al., 2007). Inoltre i dati FISH risolvono il
dibattito sulla collocazione filogenetica di Callimico goeldii. Il painting
cromosomico dimostra la presenza di riarrangiamenti cromosomici e di
associazioni sinteniche (5/7 , 8/18 e 10/16) che legano filogeneticamente Callimico ai marmosets
(Neusser et al., 2001).
Le Scimmie del Vecchio Mondo o Catarrhini
Il cariotipo
ancestrale delle scimmie del vecchio Mondo ha un numero diploide di 2n=46. Il
loro cariotipo è caratterizzato dalla fissione dell’associazione ancestrale
3/21 e dalle fusioni che hanno dato origine ai cromosomi 7, 10 e 16. Le scimmie
del Vecchio Mondo, famiglia Cercopithecidae, vengono suddivise in
Cercopithecini (babbuini, macachi, guenoni) e Colobini (scimmie asiatiche ed
africane). Sebbene da un punto di vista filogenetico queste specie siano,
rispetto all’uomo, più distanti dei Gibboni, esse presentano un cariotipo molto
più simile all’uomo. Fatta eccezione per i guenoni (cercopithecini), che si differenziano
dagli altri Primati del Vecchio Mondo per numerosi eventi di fusione e
fissione, in questo gruppo è stato osservato solo un piccolo numero di
riarrangiamenti intercromosomici. In Macaca e Papio tutte le sintenie umane
sono intatte tranne quella omologa al cromosoma umano 2 che è divisa (Wienberg et al., 1992). Poche sono le
associazioni sinteniche presenti in Macaca e Papio: 7/21, 14/15 e 20/22.
Il painting
cromosomico supporta inoltre la monofilia dei Colobini che condividono
l’associazione 21/22 e la suddivisione in un clade africano ed uno asiatico
(Bigoni et al., 1997, 2003, 2004).
Il cariotipo
ancestrale dei Cercopithecini ha numero diploide di 2n=48. Citogeneticamente il
cariotipo dei cercopitheci è caratterizzato dalla fissione dei cromosomi 3 e 5
e dalla fusione che forma l’associazione 20/21 (Finelli et al., 1999, Stanyon
et al., 2005, Moulin et al., 2008). Nei Cercopithecini, il painting cromosomico
ha dimostrato che le fissioni sono responsabili dell’aumento del numero
diploide di cromosomi. Inoltre i dati suggeriscono una biforcazione
filogenetica costituita da un ramo che porta ai Cercopithecini terrestri (Erythrocebus
patas/Chlorocebus aethiops) e dall’altro ai Cercopithecini arboricoli (Cercopithecus
neglectus, C. wolfi) (Stanyon et al., 2005), anche se questa interpretazione
non è sempre condivisa (Dutrillaux et al., 1979, Disotell and Raumer 2002,
Moulin et al., 2008).
Ominoidi
Gli ominoidi
(Hylobatidae, Pongidae e Hominidae) sono caratterizzati da un cariotipo
ancestrale con numero diploide 2n=48. I cariotipi degli ominoidi sono
caratterizzati dalla fissione dell’associazione ancestrale 14/15.
Nei Gibboni, il
“painting” cromosomico ha messo in evidenza un altissimo numero di
riarrangiamenti, soprattutto traslocazioni, alcune comuni a tutte le specie,
altre caratterizzanti alcuni raggruppamenti (Jauch et al., 1992; Koehler et al
1995a, 1995b).
Grande Scimmie Antropomorfe e l’uomo
La nostra specie
e le scimmie Antropomorfe presentano un cariotipo notevolmente simile; Homo
sapiens
(2n=46) tuttavia differisce dalle grandi scimmie (2n=48) per un corredo
cromosomico ridotto di un paio di cromosomi. Quando si ibrida la sonda
cromosomica umana 2 sulle metafasi dello scimpanzè si ottengono due segnali su
due cromosomi acrocentrici. Lo stesso risultato si riscontra nella maggior
parte dei Primati e negli altri Mammiferi dimostrando che il cromosoma umano 2
ha avuto origine dalla fusione di due cromosomi. Nelle grandi scimmie non sono
presenti altri riarrangiamenti intercromosomici, fatta eccezione per una
traslocazione reciproca tra gli omologhi dei cromosomi umani 5 e 17, avvenuta
nel Gorilla (Wienberg et al., 1990, Stanyon et al., 1992).
Gli studi
effettuati mediante bandeggiamento dei cromosomi, tuttavia, hanno suggerito che
l’evoluzione del cariotipo umano e delle grandi scimmie sia caratterizzato
soprattutto da riarrangiamenti intracromosomici (Yunish and Prakash, 1982).
Molti di questi riarrangiamenti sono stati confermati recentemente attraverso
la tecnica FISH con sonde molecolari BAC o locus specifiche (Stanyon et al.,
2008) e dagli assemblaggi delle sequenze del genoma.
Conclusione
I dati forniti
dalla citogenetica molecolare comparativa permettono l’indagine di tutto
l’assetto genomico a livello cromosomico delle specie a confronto. L’utilizzo
di pannelli di BAC con frammenti di DNA clonati permettono di approfondire lo
studio a livello di ordine dei marker genici all’interno di ciascun cromosoma.
Le prospettive future prevedono l’utilizzo parallelo e complementare degli approcci
descritti agli studi di sequenziamento. Questa raccolta di dati a livelli
risolutivi diversi permetterà una indagine genomica ancora più precisa. Un
approccio integrato delle diverse metodologie afferenti alla genomica comparata
fornirà un’analisi sempre più dettagliata sull’origine e sull’evoluzione dei
Mammiferi, dei Primati, incluso Homo sapiens.
Bibliografia
Bielec, PE, Gallagher, DS, Womack, JE, Busbee, DL, 1998, Homologies between human and dolphin chromosomes detected by heterologous chromosome painting. Cytogenet Cell Genet 81:18–25
Bigoni, F, Houck, ML, Ryder, OA, Wienberg, J, Stanyon, R, 2004, Chromosome painting shows that Pygathrix nemaeus has the most basal karyotype among Asian Colobinae. Int J Primatol 25:679–688
Bigoni, F, Stanyon, R, Koehler, U, Morescalchi, AM, Wienberg, J, 1997b, Mapping homology between human and black and white colobine monkey chromosomes by fluorescent in situ hybridization. Am J Primatol 42:289–298.
Bigoni, F, Stanyon, R, Wimmer, R, Schempp, W, 2003, Chromosome painting shows that the proboscis monkey (Nasalis larvatus) has a derived karyotype and is phylogenetically nested within Asian Colobines. Am J Primatol 60:85–93
Consigliere, S, Stanyon, R, Koehler, U, Arnold, N, and Wienberg, J, 1998, In situ hybridization (FISH) maps chromosomal homologies between Alouatta belzebul (Platyrrhini, Cebidae) and other primates and reveals extensive interchromosomal rearrangements between howler monkey genomes. Am J Primatol 46(2):119-133.
Chiarelli, B, 1963, Comparative morphometric analysis of primate chromosomes. III. The chromosomes of the geneta Hylobates, Colobus and Presbytis. Caryologia 16: 637-648.
Disotell, T,R, Raaum RL, 2002,
Molecular timescale and gene tree incongruence in the guenons. In: Glenn
ME, Cords M, eds. The Guenons: Diversity and Adaptation in African Monkeys. New York: Kluwer Academic/Plenum
Publishers, pp. 27-36.
Dumas, F, Bigoni, F, Stone, G, Sineo L, & Stanyon, R, 2005, Mapping Genomic rearrangements in titi monkeys by chromosome flow sorting and Multidirectional in situ hybridization. Chromosome Research 13: 85-96.
Dumas, F, Stanyon, R, Sineo, L, Stone, G, Bigoni, F, 2007, Phylogenomics of species from four Genera of New World monkeys by flow sorting and reciprocal chromosome painting. BMC Evolutionary Biology 2007, 7 (Suppl 2): S11
Dutrillaux, B, 1979, Chromosomal evolution in primates: tentative phylogeny from Microcebus murinus (Prosimian) to man. Hum Genet 48:251–314.
Ferguson-Smith, MA, Trifonov, V, 2007, Mammalian karyotype evolution. Nat Rev Genet. Dec;8(12):950-62.
Froenicke, L, Müller-Navia, J, Romanakis, K, Scherthan, H, 1997, Chromosomal homeologies between human, harbor seal (Phoca vitulina) and the putative ancestral carnivore karyotype revealed by Zoo-FISH. Chromosoma 106:108–113.
Finelli P, Stanyon R, Plesker R, Ferguson-Smith M.A, O’Brien PCM, Wienberg J, 1999, Reciprocal chromosome painting shows that the great difference in diploid number between human and African green monkey is mostly due to non-robertsonian fissions. Mammalian Genome 10: 713-718.
Jauch A, Wienberg J, Stanyon R, Arnold N, Tofanelli S, Ishida T,Cremer T (1992) Reconstruction of genomic rearrangements in great apes and gibbons by chromosome painting. Proc Natl Acad Sci USA 89:8611–8615.
Kellogg, ME, Burkett, S, Dennis, TR, Stone, G, Gray, BA, McGuire, PM, Zori, RT, and Stanyon, R, 2007, Chromosome painting in the manatee supports Afrotheria and Paenungulata. BMC Evol Biol 7:6.
Koehler, U, Arnold, N, Wienberg, J, Tofanelli, S, and Stanyon, R, 1995, Genomic reorganization and disrupted chromosomal synteny in the siamang (Hylobates syndactylus) revealed by fluorescence in situ hybridization. Am J Phys Anthropol 97(1):37-47.
Koehler, U, Bigoni, F, Wienberg, J, and Stanyon R, 1995, Genomic reorganization in the concolor gibbon (Hylobates concolor) revealed by chromosome painting. Genomics 30(2):287-292.
Mikkelsen, TS, Wakefield, MJ, Aken, B, Amemiya,CT, Chang, JL, Duke, S, Garber, M, Gentles, AJ, Goodstadt, L, Heger, A, Jurka, J, Kamal, M, Mauceli, E, Searle, SM, Sharpe T, Baker ML, Batzer MA, Benos PV, Belov K, Clamp M, Cook A, Cuff, J, Das, R, Davidow, L, Deakin, JE, Fazzari, MJ, Glass, JL, Grabherr, M, Greally, JM, Gu, W, Hore, TA, Huttley, GA, Kleber, M, Jirtle, RL, Koina, E, Lee, JT, Mahony, S, Marra, MA, Miller, RD, Nicholls, RD, Oda, M, Papenfuss, AT, Parra, ZE, Pollock, DD, Ray, DA, Schein, JE, Speed, TP, Thompson, K, VandeBerg, JL, Wade, CM, Walker, JA, Waters, PD, Webber, C, Weidman, JR, Xie, X, Zody, MC; Broad Institute Genome Sequencing Platform; Broad Institute Whole Genome Assembly Team, Graves JA, Ponting CP, Breen M, Samollow PB, Lander ES, Lindblad-Toh K, 2007. Genome of the marsupial Monodelphis domestica reveals innovation in noncoding sequences. Nature 447: 167–177.
Moulin, S, Gerbault-Seureau, M, Dutrillaux, B, & Richard F, 2008, Phylogenomics of African guenons. Chromosome Research 16:783–799.
Romanenko, SA, Volobouev VT,. Perelman P,L, Lebedev, VS, Serdukova NA,. Trifonov, VA, Biltueva LS, Nie, W, O_Brien, PC. M., Bulatova, NSh, Ferguson-Smith, MA, Yang & Graphodatsky AS, 2007 Karyotype evolution and phylogenetic relationships of hamsters (Cricetidae, Muroidea, Rodentia) inferred from chromosomal painting and banding comparison. 15, 293–298
Muller, S, Hollatz, M, Wienberg, J, 2003, Chromosomal phylogenyand evolution of gibbons (Hylobatidae). Hum Genet 113: 493.
Murphy, WJ, Stanyon, R, O’Brien, SJ, 2001b, Evolution of mammalian genome organization inferred from comparative gene mapping. Genome Biol
Nash, WG, O’Brien, SJ, 1982, Conserved regions of homologous G-banded chromosomes between orders in mammalian evolution: carnivores and primates. Proc Natl Acad Sci USA 79:6631–6635
Neusser, M, Stanyon, R, Bigoni, F, Wienberg, J, and Muller, S, 2001, Molecular cytotaxonomy of New World monkeys (Platyrrhini) – comparative analysis of five species by multi-color chromosome
O'Brien, SJ, Menotti-Raymond, M, Murphy, WJ, Nash, WG, Wienberg, J, Stanyon, R, Copeland, NG, Jenkins, NA, Womack, JE, and Marshall Graves, JA, 1999, The promise of comparative genomics in mammals. Science 286(5439):458-462, 479-481.
Scherthan, H, Cremer, T, Arnason, U, Weier, HU, Limade- Faria, A, Froenicke, L, 1994, Comparative chromosome painting discloses homologous segments in distantly related mammals. Nat Genet 6:342–347
Stanyon, R, Wienberg J, Romagno, D, Bigoni, F, Jauch, A, Cremer, T, 1992, Molecular and classical cytogenetic analyses demonstrate an apomorphic reciprocal chromosomal translocation in Gorilla gorilla. Am J Phys Anthropol 88:245–250
Stanyon, R, O’Brien, PC, Ferguson-Smith, MA, Plesker, R, Wienberg,
J, 1999, Defining the ancestral karyotype for all primates by
multidirectional chromosome painting between tree shrews, lemurs and humans. Chromosoma 108:393–400
Stanyon, R, Koehler U, and Consigliere, S, 2002, Chromosome painting reveals that galagos have highly derived karyotypes. Am J Phys Anthropol 117(4):319-326.
Stanyon R, Bruening R, Stone G, Shearin A, Bigoni F, 2005, Reciprocal painting between humans, De Brezza’s and patas monkeys reveals a major bifurcation in the Cercopithecini phylogenetic tree. Cytogenetic genome Research 108: 175-182.
Stanyon, R, Rocchi, M, Capozzi, O, Roberto, R, Misceo, D, Ventura,
M, Cardone, MF, Bigoni, F, and Archidiacono, N, 2008, Primate chromosome
evolution: ancestral karyotypes, marker order and neocentromeres. Chromosome Res 16(1):17-39.
Stanyon, R. and Stone, G, 2008, "Phylogenomic analysis by chromosome sorting and painting." Methods Mol Biol 422: 13-29.
Svartman M, Stone G, Page JE, and Stanyon R, 2004, A chromosome painting test of the basal eutherian karyotype. Chromosome Res 12(1):45-53.
Svartman, M, Stone, G, and Stanyon, R, 2006, The ancestral eutherian karyotype is present in Xenarthra. PLoS Genet 2(7):e109.
Wienberg, J, Jauch A, Stanyon, R, Cremer, 1990, Molecular cytotaxonomy of primates by chromosomal in situ suppression hybridization, Genomics 8:347–350
Wienberg J, Stanyon R, Jauch A, Cremer T, 1992, Homologies in human and Macaca fuscata chromosomes revealed by in situ suppression hybridization with human chromosome specific DNA libraries. Chromosoma 101:265–270.
Wienberg, J, and Stanyon, R, 1995, "Chromosome painting in mammals as an approach to comparative genomics." Curr Opin Genet Dev 5(6): 792-7.
Wienberg, J, and Stanyon, R, 1997, Comparative painting of mammalian chromosomes. Current Opinion in Genetic Development 7: 784–791.
Wienberg, J., and Stanyon R, 1998, "Comparative Chromosome Painting of Primate Genomes." ILAR J 39(2-3): 77-91.
Yang, F, Alkalaeva EZ, Perelman, PL, Pardini AT, Harrison, WR, O'Brien, PC, Fu, B, Graphodatsky, AS, Ferguson-Smith, MA, Robinson, TJ, 2003, Reciprocal chromosome painting among human, aardvark, and elephant (superorder Afrotheria) reveals the likely eutherian ancestral karyotype. Proc Natl Acad Sci U S A, 100(3):1062-1066.
Yang, F, Fu B, O’Brien, PCM, Nie, W, Ryder, OA, Ferguson-Smith, MA, 2004, Refined genome-wide comparative map of the domestic horse, donkey and human based on cross-species chromosome painting: insight into the occasional fertility of mules. Chromosome Res 12:65–76
Yunis, JJ, Prakash O, 1982, The origin of man: a chromosomal pictorial legacy. Science 215:1525–1530