
Peña, J.A., Alfonso-Sánchez, M.A., Pérez-Miranda, A.M., García-Obregón, S., Gómez-Pérez, L., 2009, GeDis: Un programa para análisis de datos en Antropogenética. Antropo, 20, 49-56. www.didac.ehu.es/antropo
GeDis: Un programa para análisis de datos en Antropogenética
GeDis: Software for data analysis in Anthropogenetics
Jose A. Peña, Miguel A. Alfonso-Sánchez, Ana M. Pérez-Miranda, Susana García-Obregón, Luis Gómez-Pérez
Departamento de Genética, Antropología Física y Fisiología Animal. Facultad de Ciencia y Tecnología. Universidad del País Vasco.
Dirección para correspondencia: Jose A. Peña. Departamento de Genética, Antropología Física y Fisiología Animal. Facultad de Ciencia y Tecnología. Universidad del País Vasco. Aptdo. 644. 48080 Bilbao (SPAIN). E-mail: joseangel.pena@ehu.es
Palabras clave: Programa informático, Antropogenética, Distancia genética.
Keywords: Software, Anthropogenetics, Genetic distance.
Resumen
Existen diferentes programas informáticos para realizar los tratamiento de datos que se realizan habitualmente a partir de frecuencias génicas en Antropogenética. La elaboración de matrices de distancia genética, la aplicación de métodos de estadística multivariante como el escalamiento multidimensional, la autocorrelación espacial o el análisis de clinas, pueden hacerse con diferentes programas, pero a menudo los métodos se encuentran dispersos entre ellos y la entrada de datos puede ser poco amigable. Con el diseño de GeDis se ha desarrollado conjuntamente una entrada de datos sencilla, una colección representativa de métodos y la posibilidad de exportar datos a otros programas de uso frecuente.
Abstract
Several computer programs exist to carry out the processing of gene frequency data in Anthropogenetics. The construction of genetic distance matrices for applying statistical multivariate methods such as multidimensional scaling analysis, spatial autocorrelation analysis or analysis of the spatial distribution of the allele frequencies (gene frequency clines) can be performed with a variety of computer programs. Yet, these methods are often dispersed in different softwares and the data entry may be hard. With the design of GeDis we seek to develop a computer program characterized by an easy data entry, by a representative collection of genetic and statistical methods and by the possibility of exporting data to other programs of frequent usage.
Introducción
El programa GeDis ha sido diseñado para cubrir las necesidades básicas de tratamiento de datos relativos a frecuencias génicas en Antropogenética. Puesto que los programas disponibles suelen proponer formatos de entrada de datos muy heterogéneos y a veces extraordinariamente tediosos de cumplimentar, se ha diseñado un programa con una entrada de datos sencilla y relativamente flexible, se han implementado algunos de los métodos más comunes para el tratamiento de los datos y se ha facilitado la exportación de datos a otros programas.
En su última versión disponible (v 1.7), GeDis permite leer datos desde una hoja de cálculo, aplicar dos coeficientes de distancia genética (R de Harpending y Jenkins y FST de Reynolds), realizar un Análisis de Escalamiento Multidimensional (MDS) sobre las matrices obtenidas con dichas distancias, utilizar la distancia de Harpending y Jenkins sobre una base de datos con valores perdidos, realizar un Análisis de Clinas sobre las frecuencias de los diferentes alelos o haplotipos, analizar la Autocorrelación Espacial para un grupo de 7 clases de distancia y exportar los datos con el formato adecuado para Arlequin y PHYLIP. El lenguaje de programación utilizado ha sido Java, de modo que GeDis puede ejecutarse en ordenadores con sistemas operativos Linux, Unix, Mac OSX y Windows.
Ejecución del programa
Como condición imprescindible para poder utilizar GeDis, es preciso tener instalado Java previamente.
Para iniciar GeDis en cualquier sistema operativo, desde un terminal en el que se ha accedido al directorio donde se encuentra el programa, se escribirá
java -jar GeDis.jar
No obstante, en algunos sistemas operativos puede utilizarse una ejecución directa a partir de un icono del programa. Es el caso de Mac OSX, donde arranca haciendo doble clic sobre GeDis.app y de Windows, donde arranca haciendo doble clic sobre GeDis.exe.
Formato de datos
Los datos en GeDis se leen desde un fichero con formato XLS denominado Data.xls, que consta de tres hojas. En la primera se anotan las frecuencias génicas, en la segunda las coordenadas geográficas y en la tercera los rangos para el análisis de Autocorrelación espacial. En todo caso, es preciso como mínimo disponer de los datos correspondientes a la primera hoja. En las casillas que deba leer el programa no debe haber fórmulas, ya que darán error de lectura. En el resto de casillas pueden incluirse otros datos, fórmulas o gráficos, ya que el programa ignorará su contenido. De este modo, pueden conservarse listados de frecuencias de otras poblaciones que pueden alternativamente incluirse o eliminarse en sucesivas iteraciones. No es preciso cerrar la hoja de cálculo mientras se ejecuta GeDis y no es preciso reiniciar GeDis para leer nuevos datos. Pero es fundamental que el fichero Data.xls se encuentre en el mismo directorio que el programa.
Primera hoja
El formato de datos de la primera hoja del libro Data.xls se estructura de la forma que se describe a continuación (figura 1).
Casilla A1: Número de poblaciones.
Casilla B1: Número de marcadores genéticos utilizados.
Fila 2, desde la casilla A2 en adelante: Número de alelos de cada marcador, hasta el total de marcadores especificados en la casilla B1.
Fila 3, desde la casilla B3 en adelante: Etiquetas de las poblaciones. Pueden contener espacios, símbolos y no hay límite para su tamaño, pero unas etiquetas muy largas harán confusas las representaciones y la lectura del fichero de resultados. No se lee la casilla A3.
Fila 4, en la casilla A4, etiqueta del primer alelo y desde la casilla B4 en adelante, frecuencias del alelo en todas las poblaciones.
Fila 5 y sucesivas, hasta el número total de alelos indicados en la fila 2, el mismo formato que la fila 4.
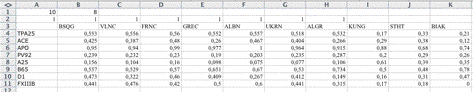
Figura 1. Formato de los datos de la primera hoja de Data.xls.
Figure 1. Format of the first sheet.
Segunda hoja
Si se desea que el programa ubique sobre un mapamundi la localización de las diferentes poblaciones, realizar un Análisis de Clinas o un análisis de Autocorrelación Espacial, será preciso anotar en la segunda hoja las coordenadas geográficas de las poblaciones, con el siguiente formato (figura 2):
Casilla A1: Número de poblaciones, como en la primera hoja.
Fila 2, desde la casilla A2 en adelante: Etiquetas de las poblaciones. Deberán ser las mismas etiquetas de la hoja 1 y en el mismo orden.
Fila 3, desde la casilla A3 en adelante: Grados de latitud para cada población.
Fila 4, desde la casilla A4 en adelante: Minutos de latitud para cada población.
Fila 5, desde la casilla A5 en adelante: 1 para latitud Norte y -1 para latitud Sur.
Fila 6, desde la casilla A6 en adelante: Grados de longitud para cada población.
Fila 7, desde la casilla A7 en adelante: Minutos de longitud para cada población.
Fila 8, desde la casilla A8 en adelante: 1 para longitud Este y -1 para longitud Oeste.
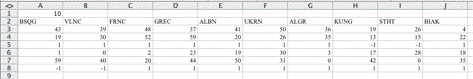
Figura 2. Formato de la segunda hoja.
Figure 2. Format of the second sheet.
Tercera hoja
Columna A, desde A1 hasta A6: 6 valores de distancia geográfica en Kilómetros en orden creciente para establecer los rangos correspondientes al Análisis de Autocorrelación Espacial (figura 3).
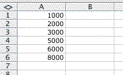
Figura 3. Formato de la tercera hoja.
Figure 3. Format of the third sheet.
Opciones del menú
Input / Read Data from Data.xls
Mediante esta opción, previa a todas las demás, el programa lee los datos de acuerdo al formato especificado en la sección anterior.
Si no hay errores fatales en el formato, en la parte superior de la pantalla se detallan el número de poblaciones, de loci y de alelos leídos. Además, se especifica si se han leído sólo las frecuencias o también las coordenadas y los rangos de distancia. En el caso de que se hayan introducido las coordenadas geográficas de las poblaciones (hoja 2 de Data.xls), las etiquetas correspondientes a las mismas aparecerán ubicadas aproximadamente sobre un mapa mundi (figura 4). Simultaneamente, este mapa se salva en un fichero EPS denominado OutPops.eps.
Además, aparecerán hasta tres hojas con los datos introducidos, con el objeto de poder verificarlos. Estas ventanas son meramente informativas, de modo que no es posible corregir los datos en ellas. Para ello es preciso modificar los datos en el fichero original.
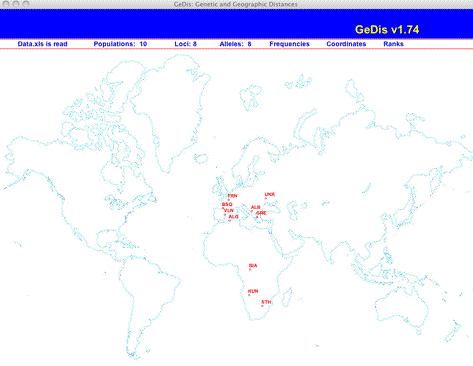
Figura 4. Ventana de GeDis tras la lectura de datos.
Figure 4. Window of GeDis after reading the data.
Distances / Harpending and Jenkins'R
El programa calcula la distancia R de Harpending y Jenkins (1973) entre cada par de poblaciones y a continuación realiza un Análisis de Escalamiento Multidimensional mediante el paquete de rutinas MDSJ, con la opción de maximización del estrés (University of Konstanz, Department of Computer & Information Science, Algorithmics Group, 2008). La matriz de distancias aparece en una nueva ventana, en tanto que el Análisis de Escalamiento Multidimensional aparece representado en la ventana principal de GeDis. Por otra parte, el gráfico resultante se salva en un fichero EPS denominado OutRDist.eps (figura 5).
Distances / Reynolds'Fst
El programa calcula una matriz de distancias mediante el coeficiente FST de Reynolds et al. (1983). A partir de esta matriz realiza un Análisis de Escalamiento Multidimensional, con la misma rutina que en la opción anterior. Del mismo modo, la matriz de distancias aparece en una nueva ventana, en tanto que el Análisis de Escalamiento Multidimensional aparece representado en la ventana principal de GeDis. El gráfico resultante se salva en un fichero EPS denominado OutFstDist.eps
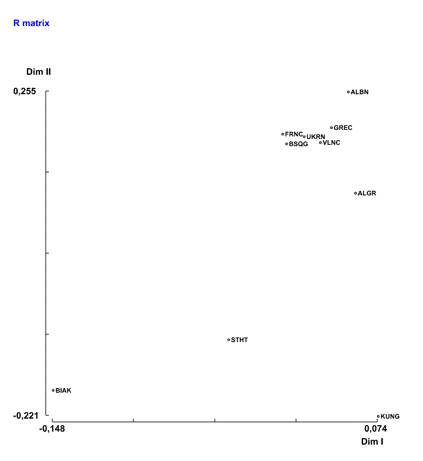
Figura 5. Análisis de Escalamiento Multidimensional obtenido a partir de la distancia R.
Figure 5. MDS obtained from the R distance.
Missing data / Harpending and Jenkins'R with missing values
Es posible realizar, mediante esta opción del menú, un Análisis de Escalamiento Multidimensional sobre una matriz de distancias R de Harpending y Jenkins (1973), aún cuanda se carezca de algunas frecuencias poblacionales en algunos alelos. El valor que debe introducirse en las casillas de las frecuencias ausentes es 1,1.
Cuando existan valores ausentes, el programa lo notificará en la ventana principal después de leer los datos, no podrá hacerse ningún otro cálculo y aquellas poblaciones con valores ausentes aparecerán en el Análisis de Escalamiento Multidimensional marcadas en color rojo.
No es preciso insistir en el hecho de que la fiabilidad estadística de los resultados obtenidos en estas condiciones es escasa y obviamente disminuye cuanto mayor sea el número de valores desaparecidos. El gráfico resultante se salva en un fichero EPS denominado OutRDistMissing.eps
Graphics / Genetic clines
Si se han introducido las coordenadas geográficas de las poblaciones, puede realizarse un análisis de clinas de las frecuencias génicas. El programa calculará el coeficiente de correlación de las frecuencias de cada alelo en relación a las coordenadas geográficas de las poblaciones, respecto a un sistema de coordenadas móviles que rotan 360 grados. Cuando existan una o varias correlaciones significativas para un alelo, el programa mostrará la clina para la orientación en la que la significación sea máxima.
El resultado final es un gráfico en el que una línea representa el grado de orientación de cada clina. Asimismo, aparecerán representadas la media y la varianza de Wahlund relativas (respecto del total de alelos considerados) de cada alelo con clina; en el primer caso la línea es de color azul y en el segundo de color verde (figura 6). El correspondiente gráfico se salva como OuCline.eps.
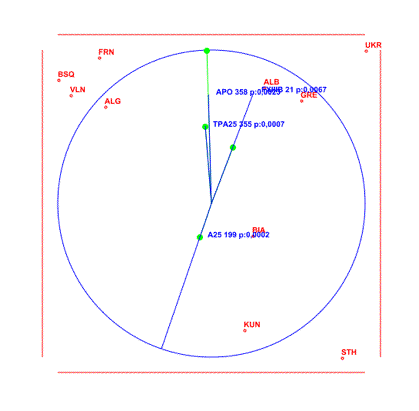
Figura 6. Análisis de clinas. La orientación de la clina se representa por el grado de inclinación la línea. La media relativa se representa por la línea azul. La varianza de Wahlund relativa se representa por la línea y el círculo verdes. La circunferencia azul representa el valor máximo de media y varianza. Para cada clina se especifica en azul el alelo correspondiente, los grados de orientación y la significación. Las etiquetas rojas representan la posición relativa de las poblaciones.
Figure 6. Cline analysis. The orientation is represented by the angle of the line. The mean frequency is represented by the blue line. The Wahlund’s variance is represented by the green line. The blue circumference represents the maximum value of mean and variance. For every clina there are specified in blue the allele label, the degrees and the signification. The red labels represent the relative position of the populations.
Graphics / Spatial autocorrelation
Para poder realizar un análisis de Autocorrelación Espacial es preciso haber incluído las coordenadas geográficas de las poblaciones y una serie de 6 valores en Kilómetros que determinan los rangos de distancia que serán considerados.
GeDis calcula el índice de Moran (1950) para cada clase de distancia, ofreciendo además el valor asociado de significación estadística (figura 7). El gráfico resultante se guarda como OutMoran.eps.
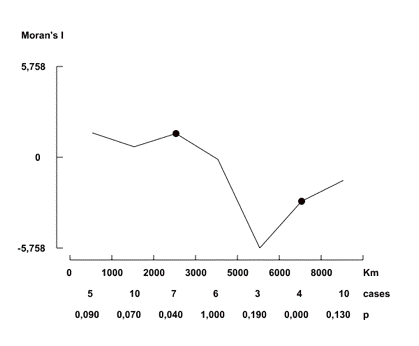
Figura 7. Análisis de Autocorrelación Espacial.
Figure 7. Spatial Autocorrelation Analysis.
Output / Save results
Mediante esta opción el programa salva en el fichero denominado Output.txt las frecuencias génicas introducidas, las coordenadas geográficas de las poblaciones, las medias de las frecuencias y sus varianzas estandarizadas, las clinas existentes, incluyendo el alelo, ángulo, coeficiente de correlación de Pearson, grados de libertad y probabilidad asociada, la I de Moran para cada clase de distancia y su correspondiente probabilidad y las coordenadas del Análisis de Escalamiento Multidimensional de las poblaciones en los análisis realizados.
Output / Save to Arlequin
Las frecuencias alélicas o haplotípicas leídas por GeDis del fichero Data.xls pueden exportarse al programa Arlequin. Mediante esta opción del menú se crearán tatos ficheros de datos con formato Arlequín como loci se incluyan en la base de datos, denominándose infile1.arp, infile2.arp, etc.
Output / Save to Phylip
También pueden exportarse los datos a Phylip. En este caso se creará un único fichero con las frecuencias de todos los marcadores, denominado infile.
Descargar GeDis
El programa se encuentra en la dirección http://www.ehu.es/~ggppegaj/javaes.html
También puede accederse desde la dirección http://www.didac.ehu.es/antropogenetica, en la sección software.
El fichero disponible en estas direcciones se denomina GeDis.zip e incluye, comprimidos, los siguientes archivos :
GeDis.app. Versión ejecutable para Mac OSX.
GeDis.exe. Versión ejecutable para Windows.
GeDis.jar. Versión multiplataforma.
Data.xls. El fichero de datos.
map.jpg. Imagen para la carátula del programa.
world.jpg. Imagen para la representación de la ubicación de las poblaciones.
Agradecimientos. Este trabajo ha sido financiado parcialmente por una beca predoctoral de la Universidad del País Vasco (Luis Gómez-Pérez), un proyecto Saiotek (S -PE08UN63) del Departamento de Industria, Comercio y Turismo del Gobierno Vasco y una subvención para actividades de grupos de investigación consolidados (IT-424-07) del Gobierno Vasco.
Bibliografía
Harpending, H.C., Jenkins, T., 1973. Genetic distance among South African populations. En Methods and Theories of Anthropological Genetics, editado por M.H. Crawford y P.L. Workman. Albuquerque: University of Mexico press pp. 177-199.
Moran, P.A.P.. 1950. Notes on continuous stochastic phenomena. Biometrika 37, 17–23.
Reynolds, J., Weir, B.S., Cockerman, C.C., 1983. Estimation of the coancestry coefficient: bases for a short term genetic distance. Genetics 105, 767–779
University of Konstanz, Department of Computer & Information Science, Algorithmics Group., 2008, MDSJ – Multidimensional Scaling for Java. http://www.inf.uni-konstanz.de/algo/software/mdsj/