
Peña, J. A., Alfonso-Sánchez, M. A.,
García-Obregón, S., Pérez-Miranda, A., 2002. Aislamiento
por distancia en poblaciones europeas sobre la base de polimorfismos
genéticos séricos. Antropo, 2, 31-39. www.didac.ehu.es/antropo
Aislamiento por
distancia en poblaciones europeas sobre la base de polimorfismos
genéticos séricos
Isolation by distance in European
populations from blood polymorphisms
Jose A. Peña,
Miguel A. Alfonso-Sánchez, Susana García-Obregón, Ana
Pérez-Miranda
Dpto. Biología
Animal y Genética. Universidad del País Vasco. Aptdo. 644. 48080
Bilbao (SPAIN). Tel. 946 012 600. E-mail: ggppegaj@lg.ehu.es
Palabras clave: Clinas, Aislamiento por la
distancia, Flujo génico, Deriva genética, Europa
Key words: Clines, Isolation by distance, Gene
flow, Genetic drift, Europe
Resumen
El objetivo de este trabajo se
centra en el análisis de la diversidad de los patrones espaciales
observados en Europa y en su interpretación como evidencias de procesos
poblacionales. Se ha analizado la variación espacial de 62 alelos y
haplotipos de 16 sistemas de polimorfismos séricos para un promedio de
37 poblaciones.
Se han detectado gradientes
significativos en 41 alelos. Entre ellos, predominan las clinas Norte-Sur y
Nordeste-Sudoeste, para las que se ha desechado la hipótesis de
selección latitudinal. La distribución de frecuencias del
parámetro a
de Malécot es normal, como cabría esperar si el origen de las
clinas fuese común. Estos resultados apoyan la hipótesis de una
gran expansión poblacional al final del Paleolítico desde el
Sudoeste hacia el Nordeste de Europa.
Abstract
This study seeks to report the
diversity of gene frequency spatial distribution patterns observed in Europe
and their interpretation as evidence of population processes. Spatial variation
in 62 allele and haplotype frequencies for blood polymorphisms in 16 genetic
systems was analyzed for 37 populations on average.
There were found significant
gradients for 41 allele frequencies, predominating among them North-south and
Northeast-southwest clinal trends.
In explaining these gradients, latitudinal selection hypothesis was rejected.
Frequencies of Malécot's a parameter fitted a normal distribution, as expected under
the assumption of common origin of clines. These results support the hypothesis
of a major late Paleolithic population expansion from Southwestern to
Northeastern Europe.
Introducción
Dentro del campo de la
Antropología física, actualmente no abundan los trabajos que se
centran en el análisis de polimorfismos séricos tradicionales, es
decir, aquellos polimorfismos que se identifican a partir de los productos
originados directa o indirectamente por los genes. Dentro de este grupo pueden
incluirse grupos sanguíneos, proteinas plasmáticas, enzimas
eritrocitarios, antígenos leucocitarios o alotipos de las
inmunoglobulinas. Por el contrario, son cada vez más frecuentes los
trabajos realizados sobre polimorfismos analizados sobre la molécula de
ADN, incluyendo minisatélites, microsatélites, inserciones ALU,
mutaciones puntuales (SNPs), alelos de diferentes genes (HLA entre otros), ADN
mitocondrial, etc. Ello no implica, sin embargo, que el primer grupo carezca de
interés. De hecho, existe una enorme base de datos reunida a lo largo de
décadas sobre frecuencias alélicas y haplotípicas
obtenidas a partir de polimorfismos séricos en diferentes poblaciones de
todo el mundo, pudiendo accederse a diferentes recopilaciones con una gran
cantidad de datos (Mourant et al., 1976; Steinberg y Cook, 1981; Tills et al., 1983; Roychoudhury y Nei,
1988; Tsuji et al.,
1992; Cavalli-Sforza et al.,
1994; Charron, 1997) que resultan muy interesantes para el estudio de la
diversidad del genoma humano.
El análisis de la
información acumulada sobre diferentes polimorfismos ha permitido
abordar el estudio de los patrones de variación geográfica del
patrimonio genético de las poblaciones humanas. Han sido varios los
métodos que se han utilizado para desvelar las clinas existentes y sus
posibles causas. Entre ellos, son bien conocidos los mapas sintéticos,
obtenidos mediante una metodología basada en el análisis de
componentes principales con interpolaciones y suavizados a posteriori, diseñada por Menozzi et
al. (1978).
Posteriormente, este método fue elaborado con más detalle por
Piazza et al.
(1981). Sin embargo, algunos aspectos metodológicos de los mapas
sintéticos han sido abiertamente criticados (Sokal et al., 1999a, 1999b; Rendine et
al., 1999).
También han sido frecuentemente utilizados otros métodos, como la
autocorrelación espacial (Sokal y Oden, 1978) y las técnicas de
detección de zonas de cambios genéticos abruptos (Barbujani et
al., 1989).
Sobre la interpretación de
los resultados obtenidos por estos métodos a partir de diferentes
polimorfismos, ha existido en los últimos años una notable
controversia.
La expansión por Europa de
los primeros agricultores y ganaderos desde el Este, conocida como la
difusión démica del Neolítico, podría para algunos
autores (Menozzi et al,
1978; Ammerman y Cavalli-Sforza, 1984) haber determinado un reemplazamiento de
buena parte de las poblaciones preexistentes, de acuerdo a los resultados
obtenidos mediante mapas sintéticos a partir de un grupo de loci
autosómicos. También para loci autosómicos, Sokal et al.
(1989) encuentran algunas clinas longitudinales que podrían sustentar la
hipótesis de la difusión démica, pero sobre todo una
mayoría de clinas latitudinales que, en su opinión,
podrían deberse a los efectos de posibles presiones selectivas.
Richards et al. (1996), a partir de ADN mitocondrial,
concluyen que la expansión de la agricultura en Europa fue esencialmente
un proceso de aprendizaje por las poblaciones autóctonas, con muy poco
flujo génico. Esta interpretación es discutida por Cavalli-Sforza
y Minch (1997), quienes argumentan que estos resultados entran en
contradicción con otros obtenidos a partir de loci autosómicos y
del cromosoma Y, con los que se observa una clina desde el Próximo
Oriente hacia el Oeste de Europa. Entre los resultados asociados al cromosoma Y
que apoyan el concepto de la difusión démica se encuentran los
reflejados en los trabajos de Semino et al. (1996) y Quintana-Murci et al. (1999). También Chikhi et
al. (1998), a
partir de varios microsatélites, minisatélites y el gen DQA1,
apoyan la hipótesis de la expansión de un grupo Neolítico
hacia el Oeste y el Norte de Europa.
En la línea de Richards et
al. (1996), para
Torroni et al.
(1998) los resultados obtenidos a partir de ADN mitocondrial muestran que una
población del Paleolítico que viviría en la
Península Ibérica y el Sudoeste de Francia, al final de la
última glaciación, habría contribuído
sustancialmente al patrimonio genético de las poblaciones modernas del
Centro y el Norte de Europa. Esta opinión concuerda con la manifestada
por Otte (1990), para quien la difusión de la industria Magdaleniense
muestra que hubo una gran expansión poblacional en Europa, desde el
Sudoeste hacia el Centro, coincidiendo con el final de la última
glaciación. Por el contrario, Simoni et al. (2000) recopilan un gran
número de secuencias de ADN mitocondrial, concluyendo que la
heterogeneidad genética en Europa se encuentra sobre todo asociada a
clinas longitudinales, originadas por la primera colonización europea
durante el Paleolítico y por la difusión démica del
Neolítico.
Con este trabajo se pretende
realizar alguna aportación al debate a la luz del modelo de aislamiento
por distancia de Malécot, analizando las clinas existentes, su
orientación y los parámetros de este modelo, utilizando con este fin
los datos recopilados para una serie de polimorfismos séricos de loci
autosómicos.
Material y métodos
Se han considerado 16 sistemas
polimórficos, con un total de 69 alelos o haplotipos (Tabla 1), de los que se han registrado un
promedio de 37 poblaciones europeas hasta completar 2502 frecuencias
génicas. Varios países han sido divididos en regiones, con el fin
de evitar en lo posible la comparación de entidades geográficas
muy desiguales. En todo caso, se ha evitado el uso de localidades aisladas o regiones
geográficas muy pequeñas, que podrían originar un sesgo
importante debido a los errores de muestreo. Por ello, se han considerado
regiones relativamente amplias y se han calculado las frecuencias en cada una
de ellas como la media de las frecuencias de todas las muestras disponibles.
Esto ha implicado una reducción en el número de poblaciones en
los análisis, pero al mismo tiempo garantiza una mayor representatividad
de las muestras. Se han asignado las coordenadas geográficas
correspondientes a la capital o la ciudad más importante de cada una de
las regiones. Los datos se han tomado de diferentes recopilaciones (Mourant et
al., 1976;
Steinberg y Cook, 1981; Tills et al., 1983; Roychoudhury y Nei, 1988; Tsuji et
al., 1992;
Cavalli-Sforza et al.,
1994; Charron, 1997).
La detección de las clinas
se ha realizado mediante correlaciones entre las frecuencias génicas y
las coordenadas geográficas, que se han rotado a intervalos de 15 grados
con el fin de conocer su orientación con suficiente precisión
para la aplicación de los posteriores tratamientos estadísticos.
Para cada alelo y haplotipo se ha elaborado la matriz R de parentesco
(Harpending y Jenkins, 1973), a partir de la cual se ha obtenido el valor de la
varianza estandarizada de las frecuencias génicas (FST) y, por regresión, los valores de
los parámetros a
y b del
modelo de aislamiento por distancia de Malécot, de acuerdo a la
fórmula de cálculo del coeficiente medio de parentesco
![]()
donde a es una medida del parentesco
local y coincide con la varianza estandarizada de las frecuencias
génicas (FST), b es la tasa de declive exponencial del parentesco con la
distancia y d es la distancia.
Todos los cálculos se han
realizado con programas diseñados por los autores de este trabajo, salvo
la regresión exponencial, para la que se ha utilizado el programa
estadístico SPSS.
Resultados
En la tabla 1 se muestran los alelos y haplotipos analizados, el número de poblaciones consideradas para cada uno de ellos, su correlación con las coordenadas geográficas, el ángulo de las mismas si se ha detectado clina, los parámetros a y b del modelo de aislamiento por distancia y la frecuencia génica promedio. Se han detectado 44 clinas entre los 69 alelos (41 clinas en 62 alelos si sólo se considera un alelo en los locus dialélicos). Esta proporción es mucho mayor que la observada por Sokal et al. (1989) (14 entre 59) para polimorfismos muy similares. Una explicación posible para esta diferencia se basa en el tratamiento de las muestras. En el trabajo de Sokal et al (1989) se consideran por separado todas las comarcas o localidades, en tanto que en este trabajo se han agrupado por regiones, con el objetivo antes mencionado de minimizar el error de muestreo. Sin embargo, la orientación de las clinas es muy similar, resultando también la mayor parte de los gradientes alineados en posiciones próximas al eje latitudinal, en tanto que las clinas longitudinales se observan en menor número.
|
Alelo/haplotipo |
Poblaciones |
R |
Rotación |
a |
b |
Frecuencia |
|
ABO*A1 |
39 |
|
|
0,012 |
0,004 |
0,199 |
|
ABO*A2 |
39 |
0,504* |
0 |
0,014 |
0,014 |
0,058 |
|
ABO*B |
39 |
0,484* |
105 |
0,016 |
0,003 |
0,082 |
|
ABO*O |
39 |
0,474* |
270 |
0,016 |
0,003 |
0,662 |
|
ACP1*A |
38 |
0,585** |
0 |
0,018 |
0,004 |
0,331 |
|
ACP1*B |
38 |
0,547** |
180 |
0,018 |
0,004 |
0,615 |
|
ACP1*C |
38 |
|
|
0,009 |
0,007 |
0,053 |
|
ADA*1 |
36 |
0,666*** |
255 |
0,013 |
0,003 |
0,932 |
|
ADA*2 |
36 |
0,653*** |
75 |
0,013 |
0,003 |
0,068 |
|
AK1*1 |
42 |
|
|
0,004 |
0,006 |
0,963 |
|
AK1*2 |
42 |
|
|
0,004 |
0,006 |
0,037 |
|
FY*A |
40 |
0,767*** |
30 |
0,036 |
0,002 |
0,434 |
|
FY*B |
40 |
0,763*** |
210 |
0,036 |
0,002 |
0,565 |
|
GC*1 |
43 |
0,546** |
15 |
0,021 |
0,005 |
0,717 |
|
GC*2 |
43 |
0,546** |
195 |
0,021 |
0,005 |
0,283 |
|
GM* 1,17 21,28 |
31 |
|
|
0,027 |
0,003 |
0,215 |
|
GM* 1,2,17 21,28 |
31 |
0,808*** |
330 |
0,022 |
0,004 |
0,074 |
|
GM* 3 5* |
31 |
0,700*** |
135 |
0,031 |
0,003 |
0,700 |
|
GM* 1,17 5* |
31 |
0,625** |
195 |
0,012 |
0,003 |
0,007 |
|
GM* 1,17
10,11,13,15,16 |
31 |
|
|
0,024 |
0,043 |
0,003 |
|
HLAA*1 |
34 |
|
|
0,013 |
0,003 |
0,142 |
|
HLAA*2 |
34 |
0,567** |
15 |
0,005 |
0,010 |
0,283 |
|
HLAA*3 |
34 |
0,704*** |
15 |
0,011 |
0,004 |
0,137 |
|
HLAA*9 |
34 |
0,512* |
165 |
0,004 |
0,003 |
0,111 |
|
HLAA*10 |
34 |
0,439* |
150 |
0,010 |
0,004 |
0,061 |
|
HLAA*11 |
34 |
|
|
0,008 |
0,020 |
0,057 |
|
HLAA*28 |
34 |
|
|
0,004 |
0,042 |
0,040 |
|
HLAA*29 |
34 |
0,637** |
210 |
0,025 |
0,002 |
0,032 |
|
HLAA*30 |
34 |
0,548** |
180 |
0,054 |
0,061 |
0,030 |
|
HLAA*31 |
34 |
|
|
0,010 |
0,316 |
0,019 |
|
HLAA*32 |
34 |
0,558** |
150 |
0,016 |
0,022 |
0,030 |
|
HLAB*5 |
34 |
0,752*** |
165 |
0,023 |
0,002 |
0,080 |
|
HLAB*7 |
34 |
0,778*** |
345 |
0,019 |
0,003 |
0,110 |
|
HLAB*8 |
34 |
0,537* |
345 |
0,019 |
0,004 |
0,096 |
|
HLAB*13 |
34 |
0,467* |
90 |
0,013 |
0,003 |
0,029 |
|
HLAB*14 |
34 |
0,574** |
210 |
0,010 |
0,003 |
0,031 |
|
HLAB*15 |
34 |
0,776*** |
0 |
0,013 |
0,003 |
0,055 |
|
HLAB*16 |
34 |
0,498* |
135 |
0,010 |
0,003 |
0,040 |
|
HLAB*17 |
34 |
|
|
0,011 |
0,329 |
0,044 |
|
HLAB*18 |
34 |
0,516* |
180 |
0,038 |
0,004 |
0,063 |
|
HLAB*21 |
34 |
0,684*** |
195 |
0,009 |
0,033 |
0,020 |
|
HLAB*22 |
34 |
|
|
0,007 |
0,004 |
0,020 |
|
HLAB*27 |
34 |
0,605** |
15 |
0,010 |
0,020 |
0,039 |
|
HLAB*35 |
34 |
0,713*** |
135 |
0,016 |
0,002 |
0,092 |
|
HLAB*40 |
34 |
0,764*** |
0 |
0,017 |
0,003 |
0,044 |
|
Hp*1 |
52 |
|
|
0,004 |
0,004 |
0,384 |
|
Hp*2 |
52 |
|
|
0,004 |
0,004 |
0,616 |
|
JK*A |
24 |
|
|
0,005 |
0,008 |
0,501 |
|
JK*B |
24 |
|
|
0,007 |
0,009 |
0,494 |
|
MNS*MS |
38 |
|
|
0,006 |
0,149 |
0,248 |
|
MNS*Ms |
38 |
|
|
0,011 |
0,012 |
0,313 |
|
MNS*NS |
38 |
0,556** |
60 |
0,014 |
0,004 |
0,088 |
|
MNS*Ns |
38 |
0,428* |
300 |
0,012 |
0,046 |
0,351 |
|
P1*1 |
42 |
0,496** |
195 |
0,012 |
0,004 |
0,491 |
|
P1*2 |
42 |
0,496** |
15 |
0,012 |
0,004 |
0,509 |
|
PI*M |
17 |
0,810** |
60 |
0,027 |
0,002 |
0,923 |
|
PI*F |
17 |
|
|
0,058 |
0,115 |
0,015 |
|
PI*S |
17 |
0,786** |
210 |
0,049 |
0,002 |
0,051 |
|
PI*Z |
17 |
|
|
0,004 |
0,022 |
0,008 |
|
RH*CDE |
47 |
0,397* |
195 |
0,024 |
0,390 |
0,007 |
|
RH*CDe |
47 |
|
|
0,011 |
0,006 |
0,437 |
|
RH*Cde |
47 |
|
|
0,006 |
0,015 |
0,016 |
|
RH*cDE |
47 |
0,526** |
15 |
0,010 |
0,003 |
0,129 |
|
RH*cDe |
47 |
0,467* |
180 |
0,013 |
0,004 |
0,035 |
|
RH*cdE |
47 |
|
|
0,005 |
0,420 |
0,007 |
|
RH*cde |
47 |
0,386* |
255 |
0,018 |
0,005 |
0,362 |
|
TF*C |
40 |
|
|
0,007 |
1,231 |
0,992 |
|
TF*B |
40 |
0,421* |
255 |
0,004 |
0,003 |
0,006 |
|
TF*D |
40 |
|
|
0,052 |
0,288 |
0,004 |
Tabla 1. Número
de poblaciones, correlación clinal, patrones de los gradientes, valores
de los parámetros a y b de Malécot y
frecuencias de los alelos analizados (* p<0,01; ** p<0,001; ***
p<0,0001).
Table 1. Number of
populations, clinal correlation, patterns of gradients, Malécot's a and b values and
frequencies of alleles analyzed (* p<0.01; ** p<0.001; *** p<0.0001).
En la figura 1 se observa la
orientación de las clinas detectadas. No se ha eliminado un alelo en los
locus dialélicos para favorecer la simetría de la representación,
pero su efecto no es importante para la interpretación del
gráfico. Puede apreciarse claramente que el mayor número de
clinas aparecen con una tendencia creciente hacia la zona situada entre el
Norte y el Norte-Nordeste (11), junto a la tendencia opuesta, entre el Sur y el
Sur-Sudoeste (13). En conjunto, predominan las clinas latitudinales respecto a
las clinas longitudinales.
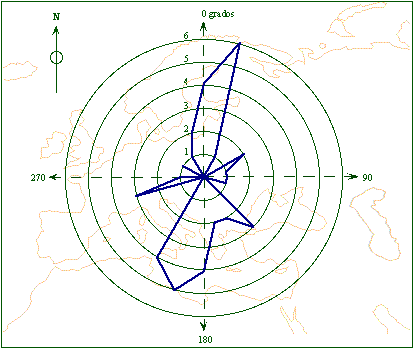
Figura 1. Gráfico
direccional que muestra los patrones de los gradientes de un grupo de loci
serológicos.
Figure 1. Directional plot showing the pattern of gradients for a group of serological loci.
Los parámetros a y b del modelo de aislamiento por distancia
de Malécot han mostrado valores diferentes entre el grupo de alelos que
presentan clinas significativas y el grupo de alelos que no las presentan
(Figuras 2 y 3).
La distribución de frecuencias del parámetro a entre los alelos que presentan clinas muestra una forma regular de acuerdo a una distribución normal (Z de Kolmogorov-Smirnof: 1,11; p: no significativa), en tanto que los alelos que no presentan clinas se distribuyen de forma más irregular entre valores más bajos, con una moda en 0,0075 y los valores más altos, por encima de 0,0400 (Figura 2). El test U de Mann-Whitney reveló unas diferencias significativas (p<0,01) entre ambos grupos de alelos. El parámetro a coincide con el valor de FST, de modo que un valor bajo implica una escasa heterogeneidad entre las poblaciones analizadas. Un valor por encima de la media, por el contrario, implica un elevado grado de heterogeneidad entre el conjunto de poblaciones consideradas.
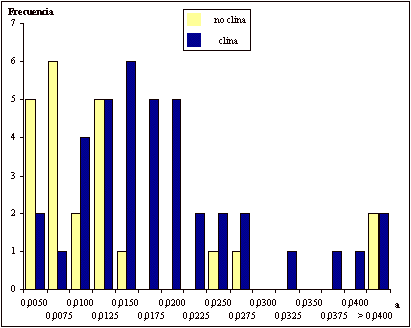
Figura 2.
Distribución de frecuencias del parámetro a de
Malécot entre alelos con clinas y alelos sin clinas.
Figure 2. Frequency distribution of Malécot's a between clinal and non clinal alleles.
También se han observado diferencias estadísticamente significativas para el parámetro b mediante el test U de Mann-Whitney (p<0,0001), de tal modo que los alelos que muestran clinas presentan mayoritariamente el valor modal (0,004) y los alelos sin distribución clinal presentan un rango de valores mayor, con un importante número de casos por encima de 0,030 (Figura 3). Un valor elevado de b implica una escasa afinidad genética con poblaciones distantes, lo que puede indicar patrones de variación local. Por el contrario, no se han detectado diferencias estadísticamente significativas mediante el test U de Mann-Whitney en los valores de a y b entre los alelos con clinas longitudinales y los alelos con clinas latitudinales.
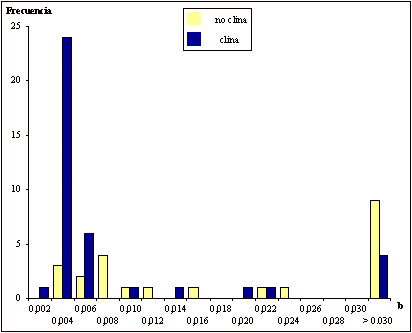
Figura 3.
Distribución de frecuencias del parámetro b de
Malécot entre alelos con clinas y alelos sin clinas.
Figure 3. Frequency distribution of Malécot's b between clinal and non clinal alleles.
Los valores altos de a coincidentes con frecuencias
génicas promedio altas, se corresponden con alelos o haplotipos en los
que existen diferencias muy importantes en relación con Asia o
Africa (FY*A, FY*B, GM*3 5) y van
asociados con clinas muy significativas (p<0,0001). Cuando la frecuencia es
baja, pueden existir diferencias importantes con otros continentes (HLAA*30,
TF*D) o no (HLAB*18, PI*F, PI*S).
Los valores altos de b se corresponden en algunos casos
con valores altos de a
(HLAA*30, PI*F, TF*D) e implican patrones locales de variación, con alto
grado de heterogeneidad genética. En el resto de alelos o haplotipos no
existen patrones claros, salvo para HLAB*21, en la que el valor elevado de b se asocia con una clina
Norte-Sur, mostrando una tendencia de aumento de frecuencias hacia la zona de
valores más altos de todo el mundo, la región del Cuerno de
Africa.
En conjunto, casi todos los
valores elevados de a
y b en alelos
asociados con clinas pueden explicarse a partir de flujos génicos con
una trascendencia local originados en regiones con grandes diferencias en las
frecuencias génicas.
Discusión
Como en el trabajo de Sokal et al
(1989), la mayor parte de las clinas detectadas son latitudinales. La
interpretación de las clinas latitudinales para esos autores se basa en
presiones selectivas no determinadas. Sin embargo, la mayor parte de los
gradientes encontrados en Europa no se reproducen en otros continentes.
Teniendo en cuenta lo reducido del ámbito geográfico europeo, se
hace difícil admitir para Europa más clinas latitudinales que en
ninguna otra región del mundo. Considerando además que en general
el modelo de aislamiento por distancia muestra valores similares de los parámetros a y b para estas clinas y las clinas
longitudinales, cabe pensar que los procesos que hayan afectado a su
distribución geográfica hayan sido los mismos, fundamentalmente
el flujo génico y la deriva genética.
Si se admite que los patrones de
heterogeneidad genética en Europa han sido determinados por estas dos
presiones evolutivas, puede establecerse una medida indirecta de la
acción de la deriva como el número de alelos que se han visto
afectados por procesos de muestreo poblacional, como el efecto fundador o el
efecto de cuello de botella. Así, un gran número de clinas
latitudinales implicará un considerable efecto de la deriva genética
en esa dirección, más importante que el efecto en relación
con la longitud, donde tan sólo unos pocos alelos se han visto
afectados. El proceso de poblamiento de Europa desde los refugios del Sur hacia
el Centro y el Norte del continente, inmediatamente después del fin de
la última glaciación, pudo haber determinado un efecto fundador
tan importante como para dejar su huella en un gran número de alelos. El
muestreo gamético asociado a este proceso podría haber creado
unas diferencias importantes entre el Norte y el Sur de Europa, que con el paso
del tiempo se han ido suavizando por la acción del flujo génico,
dando lugar a una serie de gradientes en los que se verifica el modelo de
aislamiento por la distancia. De hecho, la distribución de frecuencias
del parámetro a, que como se ha dicho, equivale a FST, sigue
una distribución normal, como cabría esperar si todas las clinas
se hubiesen originado en un mismo proceso. En definitiva, los loci
autosómicos analizados corroboran la hipótesis de Richards et
al. (1996) y
Torroni et al.
(1998), de tal modo que la estructura genética de las poblaciones
europeas vendría determinada en su mayor parte por la
colonización de Europa desde el Sur hace unos 10.000 años.
Las clinas longitudinales, por su
parte, podrían estar indicando el efecto de la difusión
démica del Neolítico desde el Este de Europa, cuya
ubicación es posterior en el tiempo y probablemente fue menos importante
que el proceso anterior, como se deduce por el menor número de clinas
detectado. Durante ese período se encontraba ya poblado todo el
continente y por tanto el efecto de la deriva pudo tener una trascendencia
menor.
Agradecimientos. Este trabajo
ha sido financiado por el proyecto UPV 154.310-EA082/98 de la Universidad del
País Vasco.
Bibliografía
Ammerman,
A. J., Cavalli-Sforza, L. L., 1984, The Neolithic Transition and the Genetics
of Population in Europe. (Princeton, USA: Princeton University Press).
Barbujani,
G., Oden, N. L., Sokal, R. R., 1989, Detecting regions of abrupt change in maps
of biological variables. Syst. Zool., 38:376-389.
Cavalli-Sforza,
L. L., Menozzi, P., Piazza, A., 1994, The History and Geography of Human Genes.
(Princeton, USA: Princeton University Press).
Cavalli-Sforza,
L. L., Minch, E., 1997, Paleolithic and Neolithic lineages in the European mitochondrial
gene pool, Am. J. Hum. Genet., 61:247-251.
Charron,
D., 1997, HLA. Genetic Diversity of HLA Functional and Medical Implication.
(Paris: EDK).
Chikhi,
L., Destro-Bisol, G., Bertorelle, G., Pascali, V., Barbujani, G., 1998, Clines
of nuclear DNA markers suggest a largely Neolithic ancestry of the European
gene pool. Proc. Natl. Acad. Sci. USA, 95:9053-9058.
Harpending,
H., Jenkins, T., 1973, Genetic distance among southern African populations. En
Methods and Theories of Anthropological Genetics, editado por M. H. Crawford y
P. L. Workman (Albuquerque: University of New Mexico Press) pp. 177-199.
Menozzi,
P., Piazza, A., Cavalli-Sforza, L. L., 1978, Synthetic maps of human gene
frequencies in Europeans. Science, 201:786-792.
Mourant,
A. E., Kopec, A. C., Domaniewska-Sobczac, K., 1976, The Distribution of the
Human Blood Groups and Other Polymorphisms. Oxford Monographs on Medical
Genetics. (Oxford, England: Oxford University Press).
Otte,
M., 1990, The northwestern European plain around 18,000 BP. En The world at
18,000 BP. Vol. 1, editado por O. Soffer y C. Gamble (London: Unwin Hyman) pp.
54-68.
Piazza,
A., Menozzi, P., Cavalli-Sforza, L. L., 1981, Synthetic gene frequency maps of
man and selective effects of climate. Proc. Natl. Acad. Sci. USA, 78:2638-2642.
Quintana-Murci,
L., Semino, O., Minch, E., Passarimo, G., Brega, A., Santachiara-Benerecetti,
A. S., 1999, Further characteristics of proto-European Y chromosomes, European
Journal of Human Genetics, 7:603-608.
Rendine,
S., Piazza, A., Menozzi, P., Cavalli-Sforza, L. L., 1999, A problem with
synthetic maps: Reply to Sokal et al. Human Biology, 71:15-25.
Richards,
M., Côrte-Real, H., Forster, P., Macaulay, V., Wilkinson-Herbots, H.,
Demaine, A., Papiha, S., et al (1996) Paleolithic and Neolithic lineages in the
European mitochondrial gene pool. Am. J. Hum. Genet., 59:185-203.
Roychoudhury,
A. K., Nei, M., 1988, Human Polymorphic Genes: World Distribution.( Oxford,
England: Oxford University Press).
Semino,
O., Passarino, G., Brega, A., Fellous, M., Santachiara-Benerecetti, A. S.,
1996, A view of the Neolithic demic diffusion in Europe through two Y
chromosome-specific markers. Am. J. Hum. Genet., 59:964-968.
Simoni,
L., Calafell, F., Pettener, D.,
Bertranpetit, J., Barbujani, G., 2000, Geographic patterns of mtDNA diversity
in Europe. Am. J. Hum. Genet., 66:262-278.
Sokal,
R. R., Harding, R. M., Oden, N. L., 1989, Spatial patterns of human gene
frequencies in Europe. American Journal of Physical Anthropology, 80:267-294.
Sokal,
R. R., Oden, N. L., 1978, Spatial autocorrelation in biology. 1. Methodology.
Biol. J. Linn. Soc., 10:199-228.
Sokal,
R. R., Oden, N. L., Thomson, B. A., 1999a, A problem with synthetic maps. Human
Biology, 71:1-13.
Sokal,
R. R., Oden, N. L., Thomson, B. A., 1999b, Letter to the Editor. Problems with
synthetic maps remain: Reply to Rendine et al. Human Biology, 71:447-453.
Steinberg,
A. G., Cook, C. E., 1981, The Distribution of the Human Immunoglobulin
Allotypes. (Oxford, England: Oxford University Press).
Tills,
D., Kopec, A. C., Tills, R. E., 1983, The Distribution of the Human Blood
Groups and Other Polymorphisms: Supplement 1. Oxford Monographs on Medical
Genetics. (Oxford, England: Oxford University Press).
Torroni,
A., Bandelt, H., Dúrbano, L., Lahermo, P., Moral, P., Sellito, D.,
Rengo, C., Forster, P., Savontaus, M., Bonné-Tamir, B., Scozzari, R.,
1998, mtDNA analysis reveals a major Late Paleolithic population expansion from
Southwestern to Northeastern Europe. Am. J. Hum. Genet., 62:1137-1152.
Tsuji,
K., Aizawa, M., Sasazuki, T., (Eds.) 1992, HLA 1991. (Oxford, England: Oxford
University Press).