Colloques du Groupement des Anthropologistes de Langue Française (GALF)
Fadhlaoui-Zid, K., Sanchez-Mazas, A., Buhler, S., Khodjet Ell khill, H., Ben Amor, M., Comas, D., Dugoujon, J.M., Slama, H., Ben Ammar Elgaaeid, A.mel, 2009, Caractérisation génétique des isolats berbérophones du Sud tunisien par confrontation des résultats de trois marqueurs polymorphes. Antropo, 18, 73-86. www.didac.ehu.es/antropo
Caractérisation génétique des isolats berbérophones du Sud tunisien par confrontation des résultats de trois marqueurs polymorphes
Genetic characterization of Berber isolates from Southern Tunisia by confrontation of results of three polymorphic markers
Karima Fadhlaoui-Zid1, Alicia Sanchez-Mazas2, Stéphane Buhler2, Houssein Khodjet Ell khill1, Mohamed Ben Amor1, David Comas3, Jean-Michel Dugoujon4, Hmida Slama5, A.mel Ben Ammar Elgaaeid1
1 Laboratoire de Génétique, Immunologie et Pathologies humaines, Faculté des Sciences de Tunis. Université Tunis El Manar 1060 Tunis, Tunisie.
2 Laboratoire d’Anthropologie, Génétique et Peuplements (AGP) Département d’Anthropologie et d’Ecologie, Université de Genève, Suisse.
3 Unitat de Biologia Evolutiva. Universitat Pompeu Fabra. Doctor Aiguader 80, 08003 Barcelona, Spain.
4 Centre d’Anthropologie, UMR 85555, Toulouse, France.
5 Centre National de Transfusion Sanguine de Tunis. La Rabta. Tunis. Tunisie.
Auteur chargé de la correspondance: Karima Fadhlaoui-Zid. Avenue de l’indépendance, Résidence Wafaa, Bloc E, Appartement E5. 2000. Bardo. Tunis. TUNISIE. Adresse électronique: karimafadhlaoui@yahoo.fr
Mots-clés: Berbères, Tunisie, Afrique du Nord, marqueurs génétiques, allotypes GM, HLA-DRB1, HLA-DQB1, ADNmt.
Keywords: Berbers, Tunisia, North Africa, genetic polymorphisms, GM allotypes, HLA-DRB1, HLA-DQB1, mtDNA.
Résumé
Dans cette étude, nous nous sommes intéressés au polymorphisme de trois marqueurs génétiques (Allotypes GM, HLA Classe II et ADNmt) au sein de trois isolats berbères (Chenini-Douiret, Matmata et Sened) localisés dans le Sud tunisien. Notre objectif est d’évaluer l’impact génétique des invasions qu’a connues la Tunisie après son peuplement berbère en estimant la diversité génétique de ces trois isolats et en comparant ces populations berbères à d’autres populations nord-africaines.
Les résultats obtenus pour les trois systèmes sont conformes entre eux et montrent que, malgré leur proximité géographique et la communauté linguistique, les trois populations berbères du Sud tunisien sont génétiquement très différenciées entre elles. Cette différenciation pourrait être due à l'effet de leur isolement et de leur endogamie, conduisant à leur divergence rapide les unes des autres. Les Berbères de Chenini-Douiret, très peu diversifiés et très divergents du point de vue génétique quel que soit le marqueur étudié, constituent un isolat. Lorsque l’on compare ces groupes berbères avec d’autres populations nord-africaines, les populations berbères du Sud tunisien apparaissent plus hétérogènes que celles du Maroc et d’Algérie. Outre l’effet de la dérive génétique, cette diversité peut refléter des contributions génétiques inégales d’origines européenne, sub-saharienne et/ou moyen-orientale ayant eu lieu au cours de la préhistoire et de l’histoire. Ainsi, la population tunisienne, à l’instar de la population nord-africaine dans son ensemble, est en réalité une mosaïque de populations.
Abstract
In this study, we are interested to polymorphism of three genetic markers (GM Allotypes, HLA Classe II and mtDNA) in three Berber isolates (Chenini-Douiret, Matmata and Sened) localed in the South of Tunisia. Our objective is to evaluate the genetic impact of the invasions which Tunisia has knew after the Berber settlement by considering genetic diversity of these three isolates and by comparing these Berber populations to other North-African populations.
Results obtained for the three systems are in agreement and show that, in spite of their geographical proximity and the great resemblance of their language, the three Berber populations of the South of Tunisia are genetically very differentiated. This differentiation could be due to the effect of their isolation and their endogamy. Berber of Chenini-Douiret, very little diversified and genetically very divergent whatever the marker studied, could be considered as an isolate. When we compared these Berber groups with other North-African populations, the Berber populations of the South of Tunisia appear more heterogeneous than those of Morocco and Algeria. In addition to the effect of the genetic drift, this diversity can reflect unequal genetic contributions of European, sub-Saharan and/or middle-Eastern components that took place during prehistory history. Thus, the Tunisian population, like the North-African populations is a mosaic of populations.
Introduction
L’anthropologie utilise, dans son approche biologique, la génétique pour comprendre les mécanismes de l’évolution de l’espèce humaine ainsi que pour décrire la diversité génétique des populations humaines. Ceci lui permet de proposer, à la lumière des autres disciplines comme l’archéologie, la linguistique et l’ethnologie, des hypothèses de reconstruction de l’histoire du peuplement humain ainsi que des interactions Homme-Milieu (sélection naturelle, influence culturelle…..).
Au cours des dernières décennies, l’anthropologie génétique a produit de nombreux travaux (voir pour l’essentiel Cavalli-Sforza et al., 1994). Les données se sont accumulées avec le développement de techniques de biologie moléculaire puissantes, fournissant de nouveaux outils pour l’analyse génétique, ainsi que les modèles théoriques d’évolution fidèles et robustes nécessaires à l’interprétation de nos résultats dans un contexte historique.
Une analyse "système par système" des polymorphismes génétiques les mieux connus à l’échelle mondiale nous permet, d’avoir une vue à la fois générale et détaillée de la diversité génétique humaine. L’étude des marqueurs génétiques dans les populations nord-africaines et en particulier la population berbère a été envisagée dans l’objectif de mieux comprendre l’histoire du peuplement de l’Afrique du Nord. Cette région est géographiquement limitée au Nord par la Méditerranée et au Sud par le désert du Sahara, qui a limité les mouvements humains et les échanges avec les populations sub-sahariennes. L’histoire rapporte l’occupation de l’Afrique du Nord par les Phéniciens, les Romains, les Vandales et d’autres peuples dont l’impact démographique reste inconnu. Postérieurement, l’islamisation des peuples berbères originaires de la zone provoqua un grand changement culturel dû à l’influence arabe. aujourd’hui, la langue berbère et la langue arabe cohabitent dans les populations d’Afrique du Nord. Les possibles différences entre les deux groupes de populations arabophones et berbérophones ne sont pas encore bien établies. Par ailleurs, les populations de cette région ont eu d’importants contacts avec d’autres populations méditerranéennes, principalement les populations européennes, mais aussi certains échanges avec les populations sub-sahariennes.
Pour analyser l’histoire du peuplement des isolats berbérophones du Sud tunisien (Chenini-Douiret, Sened et Matmata), nous avons étudié le polymorphisme de trois marqueurs: les allotypes Gm des immunoglobulines, le système HLA et l’ADN mitochondrial. Le choix de ces marqueurs est justifié par leur degré de polymorphisme élevé et de ce fait ils sont de nos jours les systèmes génétiques les plus informatifs pour étudier les relations historiques ou géographiques entre populations humaines.
Les allotypes GM sont codés par des gènes liés sur le chromosome 14 et dont les allèles sont transmis dans des combinaisons fixes appelées haplotypes. On reconnaît aujourd’hui 15 haplotypes GM communs dont la distribution qualitative et quantitative varie fortement d’une population à l’autre, permettant de définir trois grands groupes: les haplotypes GM fréquemment rencontrés dans les populations européennes, ceux fréquents en Asie et ceux communs aux populations sub-sahariens.
Le système HLA constitue l’un des systèmes les mieux caractérisés tant au niveau populationnel que moléculaire ou fonctionnel. L’existence d’un polymorphisme extrêmement étendu et de déséquilibres de liaison constituent les principaux avantages de ce système. Les études de polymorphisme HLA permettent ainsi de suivre les migrations de populations à travers l'histoire et ont donc un grand intérêt en anthropologie.
L’ADN mitochondrial présente plusieurs avantages. Il n'est hérité que par le voie matrilinéaire (pas de recombinaison) et les mutations dans le premier segment hypervariable (HVSI) de la région de contrôle sont accumulés à une vitesse plus importante que dans les autres parties du génome humaine, ceci permet une possible différentiation entre les populations plus proches. De plus les haplogroupes de l’ADN mitochondrial sont caractéristiques de continents.
Populations et Méthodes
Populations étudiées
La population berbère analysée dans le présent travail est originaire du Sud tunisien. Cette population appartient à trois isolats différents Matmata, Chenini-Douiret à l’Est de Tataouine et Sened à Gafsa (Figure 1).
Les prélèvements sanguins ont été effectués sur des volontaires (au consentement éclairé). Les individus répondaient à 3 critères: ils parlent le Berbère (nommé Chelha), ils sont originaires de la région et ils sont non apparentés.
Méthodes
Système GM
Pour le système GM, 134 sérum-tests (51 de Chenini-Douiret, 63 de sened et 20 de Matmata) ont été typé pour les allotypes G1M(1,2,3,17), G2M(23) et G3M(5,6,10,11,13,14,15,16,21,24,28) selon la méthode classique de l’inhibition de l’hémagglutination (Field et Dugoujon, 1989).
Génotypage HLA-DRBI et HLA-DQBI
Au total, 104 individus (52 de Chenini-Douiret et 52 de sened) ont été typés pour les gènes DRB1 et DQB1 du HLA classe II. Le génotypage a été réalisé par PCR/SSO.
ADN mitochondrial (ADNmt)
L’ADN a été extrait de 155 échantillons sanguins de Berbères du Sud tunisiens (53 de Chenini-Douiret, 53 de sened et 49 de Matmata). L’extraction a été réalisée à partir de sang total selon la méthode phénol-chloroforme. La zone hypervariable I de l’ADNmt a été amplifiée par PCR avec les amorces: L15996 (5’-CTCCACCATTAGCACCCAAAGC-3’) et H16401 (5’-TGATTTCACGGAGGATGGTG-3’), produisant un fragment de 445 bases. Les produits de PCR ont été purifiés avec le kit QiAEX II (Qiagen) et 10 μl de l’ADNmt ont été séquencés (ABI377 Applied Biosystems) avec les amorces H et L utilisées pour la PCR. Les séquences ainsi obtenues couvrent un total de 360 bases, de 16024 à L16391 et ont été comparées à la séquence de référence Sequence (CRS; Anderson et al., 1981; Andrews et al., 1999). Elles sont disponibles sur le site: http://www.upf.es/cexs/recerca/bioevo/index.htm (Fadhlaoui-Zid et al., 2004).
Analyse statistique
Les fréquences alléliques et haplotypiques sont calculées à l’aide du programme ARLEQUIN version 2.000 (Schneider et Coll. 2000). Des distances génétiques de Reynolds (Reynolds et al., 1983), directement liées aux FSTs, sont calculées et utilisées pour représenter graphiquement, sur deux dimensions, les relations entre populations par la méthode d’analyse d’échelonnement multidimensionnel (ou MDS) (Kruskal, 1964). Les arbres phylogénétiques ont été construits à l’aide du logiciel Neighbor du package Phylip et Treview (Saitou N, 1987).
La structure génétique et la différenciation des populations en tenant compte de structures hiérarchiques linguistiques définies a priori sont étudiées à l’aide d’une analyse de la variance moléculaire ou AMOVA (Excoffier et Coll. 1992).
Figure 1. Localisation géographique des différents isolats berbères étudiés.
Figure 1. Geographical location of the localities sampled.
Résultats et Discussion
Système GM
Des études antérieures sur le polymorphisme du système GM ont été entreprises dans différents groupes de la population tunisienne (Lefranc et Coll. 1979; Lefranc. 1980; Yacoubi et Coll. 2001; Chaâbani et Coll. 1984). Afin de comparer la répartition des haplotypes GM entre les trois isolats berbères analysés dans la présente étude et les autres groupes tunisiens de la littérature, nous avons représenté les haplotypes GM en fonction de leurs fréquences (Figure 2).
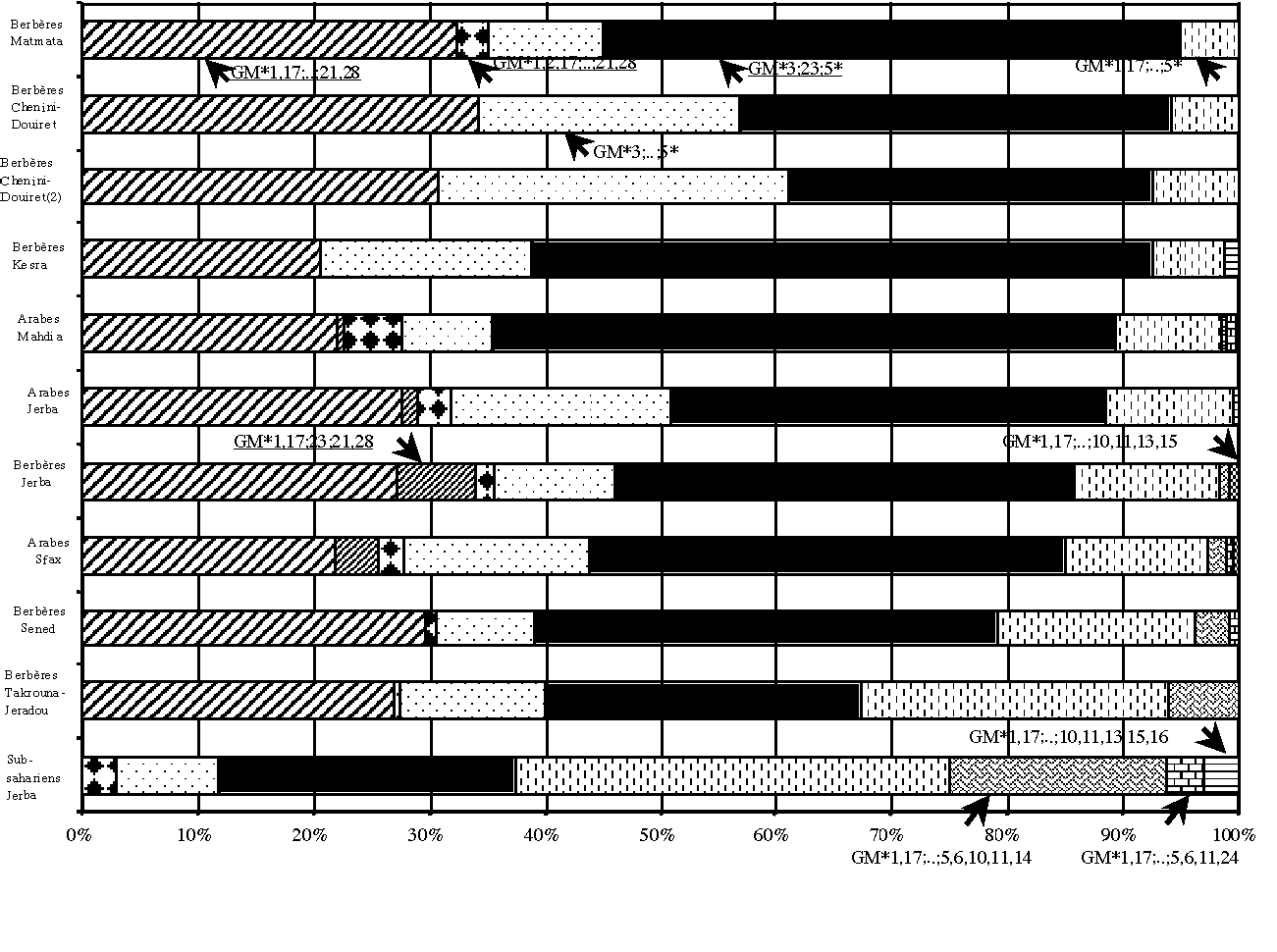
Figure 2. Répartition des haplotypes GM dans les groupes de la population tunisienne.
Figure 2. GM haplotypes repartition in the groups of Tunisian population.
Pour les Berbères de Matmata, les Arabes de Mahdia et les Berbères de Kesra, les fréquences combinées des haplotypes GM*3;23;5* et GM*3;..;5*, fréquents dans les populations européennes, sont respectivement de 0.600, 0.617 et 0.719. Ces valeurs sont proches de celles des Européens (0.72 à 0.83) et plus élevées que celles trouvées dans les autres populations tunisiennes. De plus, chez ces trois groupes, l’haplotype GM*3;23;5* est plus représenté que l’haplotype GM*3;..;5*; ceci est habituellement observé chez les Européens (Sanchez-Mazas & Pellegrini. 1990; Steinberg & Cook. 1981). Ces résultats montrent, que ces trois populations ont un caractère européen.
Les Berbères de Sened et Takrouna-Jeradou présentent une composante sub-saharienne importante (fréquences relativement élevées des haplotypes GM*1,17;..;5* et GM*1,17;..;5,6,10,11,14 caractéristiques des Africains sub-sahariens).
les Berbères de Chenini-Douiret sont particuliers étant donné qu’ils ont un rapport de fréquences de l’haplotype GM*3;23;5* / GM*3;..;5* plus faible (1 pour ceux analysés par Chaâbani et Coll. en 1984 et 1.6 dans notre cas) que dans les autres populations tunisiennes (2 à 6.9). Ce groupe de Berbères présente aussi des fréquences élevées de l’haplotype GM*1,17;..;21,28 (plus que 30%).
Ainsi, les populations tunisiennes présentent des profils génétiques très diversifiés. De nombreux haplotypes GM fréquemment observés en Europe, au Proche Orient ou en Afrique sub-saharienne sont présents dans ces populations.
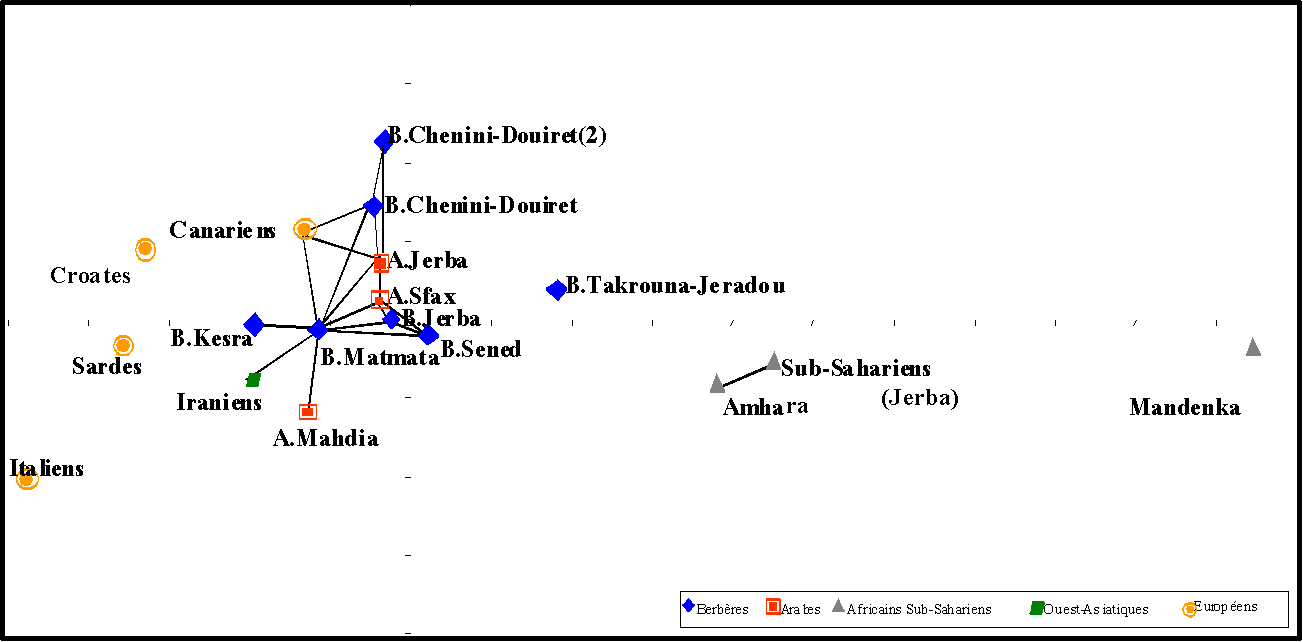
Afin de positionner les différents groupes de la population tunisienne par rapport à d’autres populations, une analyse MDS (Figure 3) a été réalisée sur la base des distances génétiques « Fst » calculées à partir des fréquences des haplotypes GM, entre les différentes populations tunisiennes et sept populations situées dans la même zone géographique que la Tunisie et pour lesquelles les fréquences des différents haplotypes GM sont détaillées dans la littérature: Africaines sub-sahariennes [ Amhara Ethiopiens (Dugoujon et Coll. sous presse) et Mandenka Sénégalais (Blanc et Coll. 1990) ]; Moyen-Orientale [Iraniens (Van Loghem et Coll. 1977) ] et Européennes [ Italiens, Sardes (Piazza et Coll. 1976), Croates (Borot et Coll. 1991) et Canariens (Esteban et Coll. 1998) ].
Globalement, cette analyse montre que les populations tunisiennes se projettent entre les Européens et les Asiatiques de l’Ouest d’une part et entre les Européens et les Africains sub-sahariens d’autre part. Ceci met en évidence que sur les grandes distances les effets géographiques Est-Ouest et Nord-Sud sont vérifiés.
Figure 3: Analyse d’échelonnement multidimensionnel (MDS) à partir des distances génétiques de Reynolds calculées entre paires de populations. Les populations présentant des valeurs non significatives de FSTs sont connectées entre elles, deux à deux. A et B désignent respectivement Arabes et Berbères.
Figure 3: Multidimensional scaling analysis (MDS) based on Reynolds genetic distances calculated between pairs of populations. Links between populations designate non-significant FSTs between them. A and B respectively designed Arabs and Berbers.
Système HLA
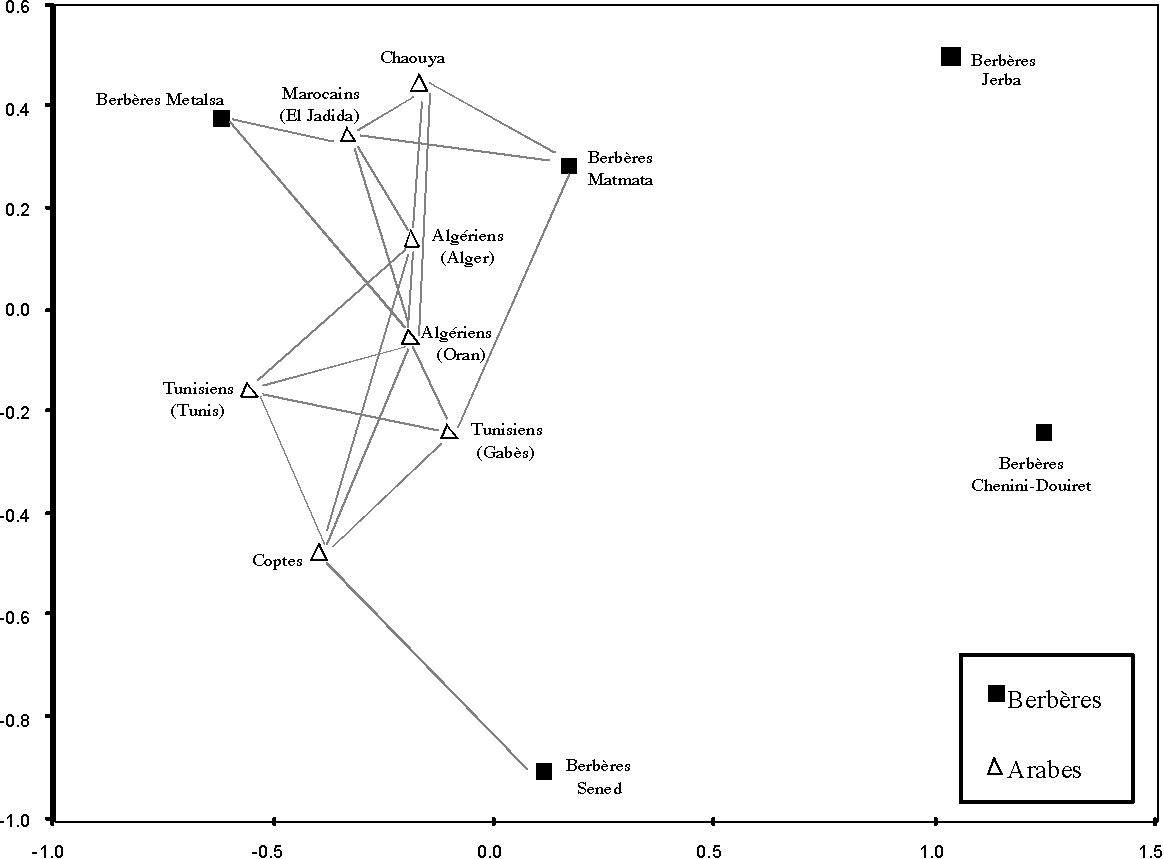
Une analyse MDS sur la base des distances génétiques FSTs calculées entre douze groupes (Arabes et berbères) (Tableau 1) de l’Afrique du Nord est représentée au niveau de la figure 6. Les populations présentant des valeurs non significatives de FSTs sont connectées entre elles, deux à deux au niveau du MDS. Cette représentation montre que les Berbères de Chenini-Douiret et ceux de Jerba sont isolés (ils présentent des valeurs de FSTs significatives avec toutes les autres populations nord-africaines).
De plus, il apparaît nettement que les populations berbères en général sont plus diversifiées sur la base des marqueurs HLA que les populations arabes. Toutes les populations berbères de Tunisie présentent des distances génétiques significatives entre elles. Ceci n’était pas évident avec le système GM qui est moins polymorphe, moins discriminant mais neutre.
|
Populations |
Pays, Localité |
N |
Références |
|
Berbères Chenini-douiret |
Tunisie, Chenini et Douiret |
52 |
Présente étude |
|
Berbères Sened |
Tunisie, Sened |
52 |
Présente étude |
|
Berbères Tunisie, |
Tunisie, Matmata |
81 |
Abdennaji Guenounou et al., 2005 |
|
Berbères Jerba |
Tunisie, île de Jerba |
55 |
Abdennaji Guenounou et al., 2005 |
|
Tunisiens |
Tunisie, Gabès |
96 |
Abdennaji Guenounou et al., 2005 |
|
Tunisiens |
Tunisie, Tunis |
102 |
Hmida et al., 1995; Hors et al., 1997 |
|
Algériens |
Algérie, Alger |
106 |
Arnaiz-Villena et al., 1995 |
|
Algériens |
Algérie, Oran |
99 |
Djoulah et al., 1994; Hors et al., 1997 |
|
Marocains |
Maroc, El Jadida |
95 |
Gomez-Casado et al. 2000* |
|
Chaouya |
Maroc |
99 |
Oumhani et al., 2002* |
|
Berbères Metalsa |
Maroc, Nador |
99 |
Oumhani et al., 2002* |
|
Coptes Egyptiens
|
Egypte, Caire et Alexandrie |
40 |
Hors et al., 1997
|
Tableau 1. Populations considérées dans cette étude pour la comparaison de HLA-DRB1. N: taille de l’échantillon. * données du 13ème IHWC.
Table 1. Populations compared in this study for HLA-DRB1. N: sample size. *13th IHWC data.
Figure 3. Analyse d’échelonnement multidimensionnel (MDS) à partir des distances génétiques de Reynolds calculées entre paires de populations. Les populations présentant des valeurs non significatives de FSTs sont connectées entre elles, deux à deux.
Figure 3. Multidimensional scaling analysis (MDS) based on HLA-DRB1 allelic frequencies and Reynold’s genetic distances. based on Reynolds genetic distances calculated between pairs of populations. Links between populations designate non-significant FSTs between them.
ADN mitochondrial
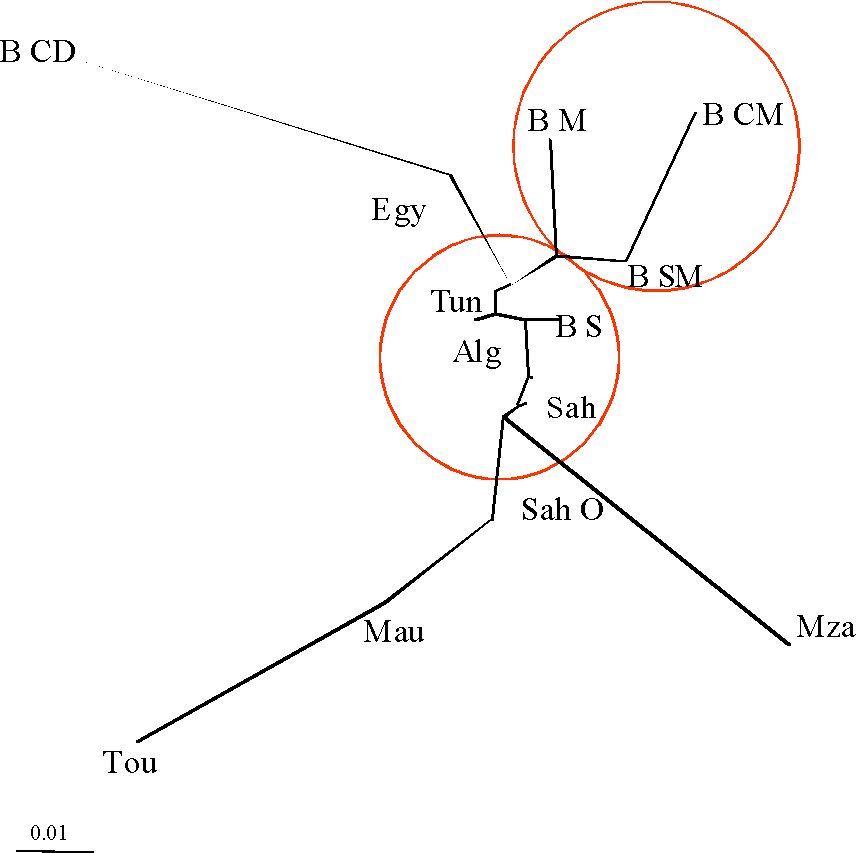
Un premier arbre Neighbor-Joining a été réalisé sur la base des distances génétiques calculées à partir des fréquences des haplotypes HVSI en ne considérant que les populations nord africaines (Tableau 2, Figure 4). Ce premier arbre révèle deux groupes de populations. Un groupe comprenant les populations Berbères de Matmata et du Centre et du Sud du Maroc. L’autre groupe rassemble la population Berbères de Sened avec les Tunisiens, les Algériens, les Arabes marocains et les Sahraouis. Un autre élément est révélé dans la Figure 4: On constate que les Berbères de Chenini-Douiret tout comme les Touaregs ou encore les Mzab, sont les plus différenciés par rapport à l’ensemble des populations de l’Afrique du Nord.
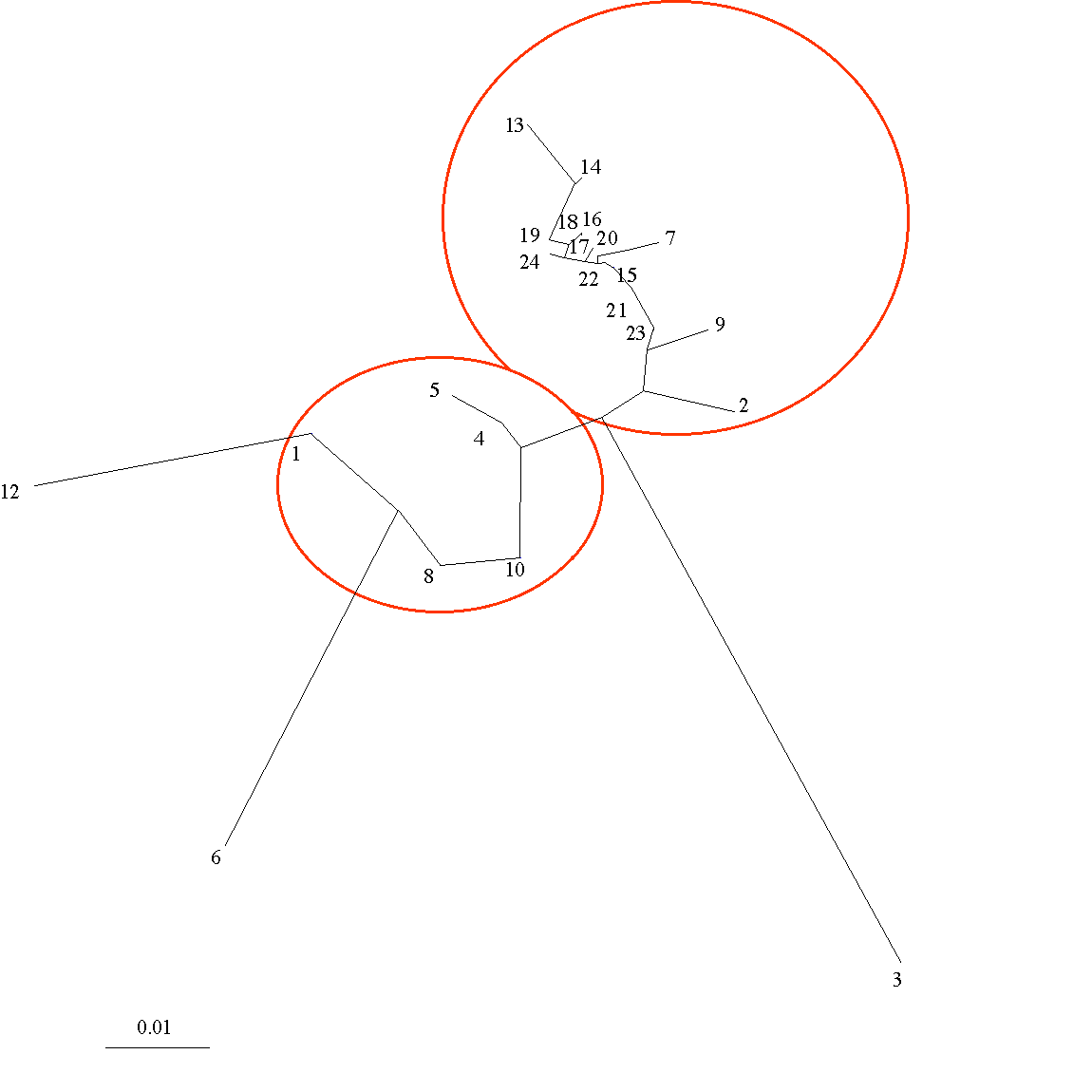
Nous avons ensuite réalisé un deuxième arbre Neighbor-Joining afin de situer les populations nord africaines et particulièrement les Berbères par rapport aux populations de la Méditerranée de l’Ouest (Tableau 2, Figure 5). Cette représentation confirme la différenciation génétique des Berbères de Chenini-Douiret, des Mzab et des Touaregs vis-à-vis de toutes les autres populations comparées (nord-africaines et européennes) et met en évidence le regroupement des Berbères de Matmata qui se caractérisent par la fréquence assez élevée de l’haplogroupe européen V (15.1%) et ceux du centre et du Sud du Maroc avec les populations Sud-Ouest européennes. Les populations européennes apparaissent génétiquement très proches par comparaison aux populations nord-africaines.
|
N° |
Populations |
N |
Références |
|
1 |
Berbères Sened |
55 |
Présente étude |
|
2 |
Berbères Matmata |
53 |
Présente étude |
|
3 |
Berbères Chenini-Douiret |
53 |
Présente étude
|
|
4 |
Tunisiens |
47 |
Plaza et al., 2003 |
|
5 |
Algériens |
47 |
Plaza et al., 2003 |
|
6 |
Mzab |
85 |
Côrte Real et al., 1966 |
|
7 |
Berbères marocains (Centre) |
64 |
Plaza et al., 2003; Rando et al., 1998
|
|
8 |
Arabes marocains |
50 |
Plaza et al., 2003; Rando et al., 1998 |
|
9 |
Berbères marocains (Sud) |
50 |
Brakez et al., 2000 |
|
10 |
Sahraouis |
81 |
Plaza et al., 2003; Rando et al., 1998 |
|
11 |
Touaregs |
26 |
Watson et al., (1996) |
|
12 |
Mauritaniens |
30 |
Rando et al., 1998 |
|
13 |
Basques |
173 |
Plaza et al., 2003; Côrte Real et Coll. 1966, Crespillo et al., 2000; López-Soto et al., 2000; A.Alonso., 2000 (communication personnelle) |
|
14 |
Galiciens |
103 |
Crespillo et al., 2000; A.Alonso (communication personnelle); Salas et al., 1998 |
|
15 |
Portugais |
295 |
Côrte Real et al., 1966; Pereira et al., 2000 |
|
16 |
Andalous |
158 |
Plaza et al., 2003; Côrte Real et al., 1966; Crespillo et Coll. 2000; López-Soto et al., 2000; A.Alonso (communication personnelle) |
|
17 |
Espagnols |
50 |
Crespillo et al., 2000; A.Alonso (communication personnelle) |
|
18 |
Valenciens |
30 |
A.Alonso (communication personnelle) |
|
19 |
Catalans |
78 |
Plaza et al., 2003; Côrte Real et al., 1966; Crespillo et al., 2000; A.Alonso (communication personnelle) |
|
20 |
Sardes |
73 |
Di Rienzo et Wilsom. 1991 |
|
21 |
Toscans |
49 |
Francalacci et al., 1996 |
|
22 |
Italiens du Centre |
83 |
Tagliabracci et al., 2001 |
|
23 |
Italiens du Sud |
37 |
Rikards et al., 2000 et communication personnelle |
|
24 |
Siciliens |
169 |
Cali et al., 2001; Rikards et al., 2000 et communication personnelle |
Tableau 2. Populations considérées dans cette étude pour la comparaison de l’ADNmt. N: taille de l’échantillon.
Table 2. Populations compared in this study for mtDNA. N: sample size.
Figure 4. Arbre Neighbor-Joining reliant les populations nord-africaines. Populations représentées: B CD: Berbères de Chenini-Douiret; BM: Berbères de Matmata; B CM: Berbères du centre du Maroc; B SM: Berbères du Sud du Maroc; Egy: Egyptiens; Tun: Tunisiens; Alg: Algériens; B S: Berbères de Sened; Sah: Sahraouis; Mza: Mzab; Mau: Mauritaniens; Tou: Touareg.
Figure 4. Neighbor-Joining tree of North African populations. Populations studied: B CD: Chenini-Douiret Berbers; BM: Matmata Berbers; B CM: Center Moroccan Berber; B SM: South Moroccan Berbers; Egy: Egyptians; Tun: Tunisians; Alg: Algerians; B S: Sened Berbers; Sah: Saharawi; Mza: Mozabites; Mau: Mauritanians; Tou: Tuareg.
Analyse de la variance moléculaire (AMOVA)
En plus des trois isolats berbères étudiés dans le présent travail, d’autres populations nord-africaines ont été considérées dans cette analyse. Ces populations ont été rassemblées selon des critères linguistiques et culturels en deux groupes (Arabes et Berbères) et une analyse de variance a été réalisée pour chaque groupe. Les résultats obtenus montrent que quel que soit le marqueur analysé, les Berbères sont plus diversifiés que les Arabes. En effet, dans le groupe berbère 12.21%, 1,74%, 2,04% et 6.16% (respectivement pour GM, HLA- DRB1, HLA-DQB1et ADNmt ) de la variance génétique sont dus aux différences entre les populations considérées alors que dans le groupe arabe 4.04%, 0,35%, 1,18% et 1.54% de la variance génétique sont attribués aux différences entre les populations analysées.
La confrontation de ces deux groupes, révèle que les arabes et les Berbères en Afrique du nord ne sont pas génétiquement différenciés (Tableau 3).
L’absence de différenciation génétique entre les Berbères et les Arabes de l’Afrique du Nord a aussi été observée à travers l’analyse d’autres marqueurs génétiques comme les marqueurs classiques (Bosch et Coll. 1997); les STR autosomiques (Bosch et Coll. 2000), le polymorphisme des insertions Alu (Comas et Coll. 2000) et le chromosome Y (Bosch et Coll. 2001). Ces observations pourraient être expliquées par deux l’hypothèses: La contribution moyen-orientale ancienne (probablement capsienne) à l’origine des Berbères (Renfrew. 1991; Camps. 1995), l’origine berbère des populations dites arabes f’Afrique du Nord en sachant que ces deux hypothèses ne sont pas exclusives.
Figure 5. Arbre Neighbor-Joining reliant les populations ouest méditerranéennes. Les populations correspondant aux numéros sont présentées dans le tableau 2.
Figure 5. Neighbor-Joining tree of West Mediterranean populations. Populations numerous are in table 2.
|
Marqueur |
n |
FST(%) |
Sign |
FCT(%) |
Sing |
|
GM |
|
|
|
|
|
|
Berbères |
7 |
12.21 |
*** |
|
|
|
Arabes |
11 |
4.04 |
*** |
|
|
|
Arabes vs Berbères |
18 |
|
|
3.88 |
n.s |
|
HLA |
|
|
|
|
|
|
HLA- DRB1 (haute résolution) |
|
|
|
|
|
|
Berbères |
5 |
1,74 |
*** |
|
|
|
Arabes |
7 |
0,35 |
*** |
|
|
|
Arabes vs Berbères |
12 |
|
|
0,07 |
n.s |
|
HLA- DQB1 (haute résolution) Berbères |
5 |
2,04 |
*** |
|
|
|
Arabes |
7 |
1,18 |
*** |
|
|
|
Arabes vs Berbères |
12 |
|
|
0 |
n.s |
|
ADNmt |
|
|
|
|
|
|
Berbères |
8 |
6.16 |
** |
|
|
|
Arabes |
5 |
1.54 |
** |
|
|
|
Arabes vs Berbères |
14 |
|
|
-0.20 |
n.s |
Tableau 3. Diversité génétique entre (FCT) et à l’intérieur (FSC) des groupes linguistiques. n: nombre de populations. **: 0.001<P<0.01; ***: P<0.001; n.s.: non significatif (P>0.05)
Table 3. Genetic diversity among populations (FCT) and within (FSC) linguistic groups.
Conclusion
Les résultats obtenus pour les trois systèmes sont concordants et montrent que, malgré leur proximité géographique et la grande ressemblance de leur parler, les trois populations berbères du Sud tunisien sont génétiquement très distinctes. Cette divergence est explicable par leur isolement et leur endogamie, facteurs conduisant à une différenciation rapide. Le cas extrême est celui des Berbères de Chenini-Douiret, très peu diversifiés et très divergents du point de vue génétique quel que soit le marqueur étudié; il s’agit clairement d’un isolat. Lorsque l’on compare ces groupes berbères avec d’autres populations nord-africaines, les populations berbères du Sud tunisien apparaissent plus hétérogènes que celles du Maroc et d’Algérie. Outre l’effet de la dérive génétique, cette diversité peut refléter des contributions génétiques inégales d’origines européenne, sub-saharienne et/ou moyen-orientale ayant eu lieu au cours de la préhistoire et de l’histoire. Ainsi, la population tunisienne, à l’instar de la population nord-africaine dans son ensemble, est en réalité une mosaïque de populations. Nos résultats montrent qu’on ne saurait définir de « profil génétique berbère » représentatif des populations berbères ancestrales. Plus généralement, on peut se demander s’il est pertinent de considérer les profils génétiques de populations culturellement et/ou géographiquement isolées comme références pour des périodes anciennes, comme les Basques en Europe pour représenter les populations de la période pré-néolithique.
Bibliographie
Abdennaji Guenounou, B., Yacoubi Loueslati, B., Buhler, S., Hmida, S., Ennafaa, H., Khodjet-Elkhil, H., Moojat, N., Dridi, A., Boukef, K., Ben Ammar Elgaaied, A. et Sanchez-Mazas,A., 2006, HLA class II genetic diversity in Southern Tunisia and the Mediterranean area. Int J Immunogenet, 33(2), 93-103.
Alonso, A., 20000 communication personnelle.
Anderson, S., Bankier, A.T., Barrell, B.G., Bruijn, M.H.L., Coulson, A.R., Drouin, J., Eperon, I.C., Nierlich, D.P., Roe, B.A., Sanger, F., Schreier, P.H., Smith, A.J.H., Staden, R. and Young, I.G., 1981, Sequence and organization of the human mitochondrial genome. Nature, 290, 457-465.
Andrews, R.M., Kubacka, I., Chinnery, P. F., Lightowlers, R. N., Turnbull, D. M. and Howell, N., 1999, Reanalysis and revision of the Cambridge reference sequence for human DNA. Nat Genet, 23, 147.
Arnaiz-Villena, A., Benmamar, D., Alvarez, M., Diaz-Campos, N., Varela, P., Gomez-Casado, E. and Martinez-Laso, J., 1995, HLA allele and haplotype frequencies in Algerians. Relatedness to Spaniards and Basques. Human Immunology, 43, 259.
Blanc, M., Sanchez-Mazas, A., Van Blyenburgh, N.H., Sevin, A., Pison, G., Langaney, A., 1990, Interethnic genetic differentiation: Gm polymorphism in eastern Senegal. Am J Hum Genet, 46, 383-392.
Borot, N., Dugoujon, J.M., Janicijevic, B. and Coll., 1991, Gm and Km immunoglobulin allotypes in populations on the island of Pag, Croatia. College of Anthropology, 15, 247-255.
Bosch, E., Calafell, F., Perez-Lezaun, A., Comas, D., Mateu, E. & Bertranpetit, J., 1997, Population history of North Africa: evidence from classical genetic markers. Hum. Biol, 69, 295-311.
Bosch, E., Calafell, F., Perez-Lezaun, A., Clarimon, J., Comas, D., Mateu, E., Martinez-Arias, R., Morera, B., Brakez, Z., Akhayat, O., Sefiani, A., Hariti, G., Cambon-Thomsen, A. & Bertranpetit, J., 2000, Genetic structure of north-west Africa revealed by STR analysis. Eur J Hum Genet, 8, 360-366.
Bosch, E., Calafell, F., Comas, D., Oefner, P.J., Underhill, P.A. & Bertranpetit, J., 2001, High-resolution analysis of human Y-chromosome variation shows a sharp discontinuity and limited gene flow between northwestern Africa and the Iberian Peninsula. Am J Hum Genet. 68,1019-1029.
Brakez, Z., Bosch, E., Izaabel, H., Akhayat, O., Comas, D., Bertranpetit, J. and Calafell, F., 2001, Human mitochondrial DNA sequence variation in the Moroccan population of the Souss area, Ann Hum Biol, 28, 295-307.
Cali, F., Le Roux, M.G., D’Anna, R., Flugy, A., De Leo, G., Chiavetta, V., Ayala, G.F. & Romano, V., 2001, MtDNA control region and RFLP data for Sicily and France, Int J Legal Med. 114, 229-231.
Camps, G. (1995). Les Berbères, Mémoire et identité. Editions France (Paris).
Cavalli-Sforza, L.L., Menozzi, P. & Piazza, A.,1994, The History and Geography of Human Genes, Princeton, New Jersey: Princeton University Press.
Chaabani, H., Helal, A.N., Van Loghem, E., Langaney, A., Benammar Elgaaied, A., Rivat Peran, L., Lefranc, G., 1984, Genetic study of Tunisian Berbers. I. Gm, Am and Km immunoglobulin allotypes and ABO blood groups. J Immunogenet, 11, 107-113.
Comas, D., Calafell, F., Benchemsi, N., Helal, A., Lefranc, G., Stoneking, M., Batzer, M.A., Bertranpetit, J. and Sajantila, A., 2000, Alu insertion polymorphisms in NW Africa and the Iberian Peninsula: evidence for a strong genetic boundary through the Gibraltar Straits. Hum Genet, 107, 312-319.
Côrte-Real, H.B., Macaulay, V.A., Richards, M.B., Hariti, G., Issad, M.S., Cambon-Thomsen, A., Papiha, S., Bertranpetit, J. and Sykes, B.C., 1996, Genetic diversity in the Iberian Peninsula determined from mitochondrial sequence analysis, Ann Hum Genet, 60, 331-350.
2000, Mitochondrial DNA sequences for 118 individuals from northeastern Spain. Int J Legal Med, 114,130-132.
., 1991, Branching pattern in the evolutionary tree for human mitochondrial DNA.Proc Natl Acad Sci USA, 88,1597-1601.
Djoulah, S., Sanchez-Mazas, A., Khalil, I., Benhamamouch, S., Degos, L., Deschamps, I. and Hors, J., 1994, HLA-DRB1, DQA1 and DQB1 DNA polymorphisms in healthy Algerian and genetic relationships with other populations. Tissue Antigens, 43, 102.
Dugoujon, J.M., Hazout, S., Loirat, F., Mourrieras, B., Crouau-Roy, B., Sanchez-Mazas, A., 2004, Gm haplotype diversity of 82 populations over the world. Am J Phys Anthropol, 125(2), 175-92.
Esteban, E., Dugoujon, J.M., Valveny, N., Conzalez-Reimers, E. and Moral, P.,1998, Spanish and African contribution to the genetic pool of the Canary islanders: data from GM and KM haplotypes and RFLPs in the immunoglobulin IGHG loci. Ann Hum Genet, 62, 33-45.
Excoffier, L., Smouse, P. and Quattro, J.M., 1992, Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics, 131, 479-491.
Fadhlaoui-Zid, K., Plaza, S., F, Calafell., Ben Amor, M., Comas,D., and. Bennamar El gaaied, A., 2004, Mitochondrial DNA Heterogeneity in Tunisian Berbers. Annals of Human Genetics, 68, 222–233.
Field, L.L. and Dugoujon, J.M., 1989, Immunoglobulin allotyping (Gm and Km) of GAW5 families. Genet Epidemiol, 6, 31-34.
Gomez-Casado, E., del Moral, P., Martinez-Laso, J., Garcia-Gomez, A., Allende, L., Silvera-Redondo, C., Longas, J., Gonzalez-Hevilla, M., Kandil, M., Zamora, J. and Arnaiz-Villena, A., 2000, HLA genes in Arabic-speaking Moroccans: close relatedness to Berbers and Iberians. Tissue Antigens, 55, 239.
Hmida, S., Gauthier, A., Dridi, A., Quillivic, F., Genetet, B., Boukef, K. and Semana, G., 1995, HLA class II gene polymorphism in Tunisians. Tissue Antigens, 45, 63.
Hors, J., El Chenawi, F., Djoulah, S., Hafez, M., Abbas, F., El Borai, M.-H., Kamel, M., Abbal, M., A., C.-T., Mercier, P., Reviron, D., Magzoub, M.-A., Rosner, G., Delgado, J.-C., Yunis, E., Raffoux, C., Tamouza, R., Izaabel, H., Hmida, S., Benhamamouch, S., Bessaoud, K., Langaney, A. and Sanchez-Mazas, A.,1997, HLA in North African populations: 12th International Histocompatibility Workshop: NAFR report. In: Genetic diversity of HLA: functional and medical implication. Proceedings of the 12th International Histocompatibility Workshop and Conference (Paris, June 1996) (ed. by D. Charron), p. 328. EDK, Paris.
Kruskal, J., 1964, Multidimensional scaling by optimizing goodness of fit to a nonmetric hypothesis. Psychometrika, 29, 1-27.
Lefranc, G., De Lange, G., Rivat, L., Langaney, A., Lefranc, M.P., Ellouze, F., Sfar, G., Sfar, M and Van Loghem, E., 1979, Gm, Am and Km immunoglobulin allotypes of two populations inTunisia. Hum Genet, 50, 199-211.
Lefranc, m.p., 1980,. Les allotypes gm, am, et km des immunoglobulines humaines dans deux échantillons de la population tunisienne (Mahdia et sfax): contribution à la connaissance séroanthropologique de cette population et à la génétique des immunoglobulines. Thèse de doctorat paris-sud.
López-Soto, M. andSanz, P., 2000, Mitochondrial DNA polymorphisms in individuals living in Andalusia (south Spain) and Extremadura (western Spain). In: Cuadernos Medicina Forense. pp. 174-24.
Loveslati, B.Y., Sanchez-Mazas, A., Ennafaa, H., Marrakchi, R., Dugoujon, J.M., Lefranc, J.M and Elgaaied, A.B., 2001, A study of Gm allotypes and immunoglobulin heavy gamma IGHG genes in Berbers, Arabs and sub-Saharan Africans from Jerba Island, Tunisia. Eur J Immunogenet, 28, 531-538.
Oumhani, K., Canossi, A., Piancatelli, D., Di Rocco, M., Del Beato, T., Liberatore, G., Aureli, A., Benjoaud, A., El Aouad, R., Adorno, D., Casciani, C.U. and Ben Jouad, A.E., 2002, Sequence-Based analysis of the HLA-DRB1 polymorphism in Metalsa Berber and Chaouya Arabic-speaking groups from Morocco. Human Immunology, 63, 129.
., 2000, Diversity of mtDNA lineages in Portugal: Not a genetic edge of European variation. Ann Hum Genet, 64, 491-506.
Piazza, A., Van Loghem, E., De Lange, G., Curtoni, E.S., Ulizzi, L. and Terrenato, L.,1976, Immunoglobulin allotypes in Sardinia. Am J Hum Genet, 28, 77-86.
Plaza, S., Calafell, F., Helal, A., Bouzerna, N., Lefranc, G., Bertranpetit, J. and Comas, D., 2003, Joining the pillars of Hercules: mtDNA sequences show multidirectional gene flow in the western Mediterranean. Annals of Human Genetics, 67, 312.
Rando, J.C., Pinto, F., Gonzalez, A.M., Hernandez, M., Larruga, J.M., Cabrera, V.M. and Bandelt, H.-J., 1998, Mitochondrial DNA analysis of northwest African populations reveals genetic exchanges with European, near-eastern, and sub-Saharan populations. Annalsof Human Genetics, 62, 531-550
Renfrew, C., 1991, Before Babel: speculations on the origins of linguistic diversity. Cambridge Archaeol. J, 1, 3-23.
Reynolds, J., Weir, B. and Cockerham, C., 1983, Estimation for the coancestry coefficient: basis for a short-term genetic distance. Genetics, 105, 767-779
Rickards, O., Martínez labarga, C., Casalotti, R., Castellana, G., Tunzi sisto, A.M. and Mallegni, F., 2000, MtDNA variability in extinct and extant populations of Sicily and southern Italy. In: C.Renfrew and K.Boyle (Eds), Archaeogenetics: DNA and the population prehistory, pp. 175-183, McDolnald Institute Monographs, McDonald Institute for Archaeological Research, Cambridge.
Rickards, O., 2000 communication personelle.
Saitou, n. and Nei, m., 1987, The neighbor-joining method: a new method for reconstructing phylogenetic tree. Mol biol evol, 4, 406-425.
1998, MtDNA analysis of the Galician population: a genetic edge of European variation. Eur J Hum Genet, 6, 365-375.
Sanchez-Mazas, A., 1990, Polymorphisme des systèmes Rhésus, Gm et HLA et histoire du peuplement humain. Thèse de doctorat. Université de Genève.
Schneider, S., Roessli, D., and Excoffier, L., 2000, Arlequin ver. 2.000: A Software for Population Genetics Data Analysis (Genetics and Biometry Laboratory, University of Geneva, Switzerland).
Steinberg, A.G. and Cook, C.E.,1981, The distribution of the human immunoglobulin allotypes, Oxford, New-York, Toronto: Oxford University Press.
Tagliabracci, A., Turchi, C., Buscemi, L. and Sassaroli, C.,2001, Polymorphism of the mitochondrial DNA control region in Italians. Int J Legal Med, 14, 224-228.
Van Loghem, E., Lange, G., Matsumoto, H., Mizazaki, T. and Ohkura, K., 1977, Immunoglobulin haplotypes of two population groups in Iran. J Immunogenet, 4, 385-390.
Watson, E., Bauer, K., Aman, R., Weiss, G., von Haeseler, A., and Pääbo, S., 1996, mtDNA sequence diversity in Africa. American Journal of Human Genetics, 59, 437-444.