
Colloques du Groupement des Anthropologistes de Langue Française (GALF)
Coudray, C., Torroni, A., Achilli, A., Pala, M., Olivieri, A., Larrouy, G., Dugoujon, J.M., 2009, Les lignées mitochondriales et l’histoire génétique des populations berbérophones du nord de l’Afrique. Antropo, 18, 63-72. www.didac.ehu.es/antropo
Les lignées mitochondriales et l’histoire génétique des populations berbérophones du nord de l’Afrique
Mitochondrial lineages and genetic history of Berber-speaking populations from North Africa
Clotilde Coudray1, Antonio Torroni2, Alessandro Achilli2, Maria Pala2, Anna Olivieri2, Georges Larrouy1, Jean-Michel Dugoujon1
1Laboratoire d’Anthropobiologie, CNRS FRE 2960, 37 allées Jules Guesde, 31073 Toulouse, France
2Dipartimento di Genetica e Microbiologia, Università di Pavia, Via Ferrata 1, 27100 Pavia, Italie
Auteur chargé de la correspondance: Clotilde Coudray. E-mail: clotilde.coudray@wanadoo.fr
Mots clefs: Berbères, peuplement nord-africain, ADN mitochondrial
Key Words: Berbers, North-African settlement, mitochondrial DNA
Résumé
Divers marqueurs moléculaires peuvent être utilisés pour retracer la diversité génétique des populations humaines. Nous présentons ici nos travaux sur la diversité mitochondriale des populations berbérophones du nord de l’Afrique. Les analyses portent sur le séquençage de la région contrôle (HVS-I et HVS-II) et sur des RFLPs dans la région codante de l’ADN mitochondrial (ADNmt) de différentes populations berbères du Maroc et d’Egypte.
Au travers de comparaisons avec des populations géographiquement proches (notamment du sud-ouest de l’Europe, du Proche-Orient et des régions sub-sahariennes), notre objectif est de comprendre comment cette diversité a pu se mettre en place au nord de l’Afrique en rapport avec les divers mouvements de populations.
Nos résultats montrent que les populations nord-africaines occupent, dans la diversité mitochondriale globale, une position intermédiaire entre les populations européennes et sub-sahariennes et qu’il apparaît une différenciation génétique entre le nord-ouest et le nord-est du continent africain, les populations du Maghreb se rapprochant des populations européennes et du Moyen-Orient alors que les populations de Haute Egypte montrent plus d’affinités avec les populations sub-sahariennes et d’Afrique de l’Est. Nos travaux révèlent également une nette et significative différenciation génétique entre les Berbères du Maghreb et les Berbères égyptiens, ces derniers présentant plus d’affinités avec les populations est-africaines.
Abstract
Various molecular markers can be used to study the genetic diversity of human populations. Here, we present our work on the mitochondrial diversity of Berber-speakers from North Africa. Analyses were done after control region sequencing (HVSI and HVSII) and coding region RFLPs of the mitochondrial DNA (mtDNA) from various Berber populations from Morocco and Egypt.
Through comparison with geographically related populations (in particular from south-western Europe, the Middle East and sub-Saharan areas), our objective is to understand how this diversity was set up in North Africa in connection with the several migrations of different populations.
Our results show that North-Africans have, in the total mitochondrial diversity, an intermediate position between European and sub-Saharan populations. We also see a genetic differentiation between North-Western and North-Eastern African groups: populations from Maghreb are related to European and Middle Eastern populations whereas High Egyptians share more affinities with sub-Saharans and East Africans. Our data also reveal a clear and significant genetic differentiation between Berbers from Maghreb and Egyptian Berbers, the latter sharing more affinities with East-African populations.
Introduction
Si les vestiges archéologiques et paléoanthropologiques mis au jour témoignent de l’ancienneté (Paléolithique) de l’occupation du nord de l’Afrique par l’Homme, la question de l’évolution de ces groupes humains jusqu’à nos jours reste posée. Les raisons de ce questionnement sont directement liées aux évènements préhistoriques et historiques qui se sont succédé dans cette région depuis l’installation de ses plus anciens habitants, les ancêtres des Berbères. La présence de ces derniers au nord de l’Afrique daterait sans doute du Mésolithique (avec l’industrie capsienne, 10.000 – 4.700 ans BP), mais est néanmoins incontestablement marquée au Néolithique (vers 11.000 ans BP dans les régions sahariennes et 5.000 ans BP au Maghreb) par diverses manifestations culturelles et par l’introduction des langues afro-asiatiques. Ces hommes ont ensuite eu une histoire riche en invasions, conquêtes et tentatives d’assimilation par, tour à tour, des phéniciens, des romains, des vandales et des byzantins. Mais c’est la conquête arabe amorcée au VIIème siècle qui a été l’évènement le plus décisif, au cours duquel l’islamisation et l’arabisation ont déterminé durablement le destin des Berbères. Malgré une forte résistance, les Berbères durent se soumettre à l’autorité arabe. Les groupes réfractaires ont été chassés de leurs terres et contraints de se retirer dans des régions plus isolées. Ce passé mouvementé a eu une incidence directe sur la répartition géographique des communautés berbères qui se retrouvent aujourd’hui disséminées dans un vaste territoire s’étendant de la Mauritanie à l’Egypte (oasis de Siouah) et du désert du Sahara aux régions montagneuses de l’Atlas marocain.
Nos travaux sont réalisés dans le cadre d’un projet CNRS et EUROCORES «Origin of Man, Language and Languages» (OMLL) ayant pour but de décrire la diversité génétique et linguistique des Berbères et de retracer leurs degrés de parenté avec des populations géographiquement proches, de culture et de langue différentes. Nous partons de l’hypothèse qu’au cours du temps, les différentes populations qui ont migré au nord de l’Afrique, en plus de leur impact démographique, ont très certainement laissé leurs empreintes dans le patrimoine génétique des populations berbères. Ainsi, afin de retracer « l’histoire génétique » des Berbères, différents marqueurs informatifs en génétique des populations humaines (microsatellites, allotypes GM, polymorphismes de l’ADN mitochondrial, du chromosome Y…) peuvent être étudiés (Plaza et al., 2003 ; Cruciani et al., 2004 ; Dugoujon et al., 2004 ; Coudray et al., 2006a ; Coudray, 2006b, Olivieri et al., 2006 ; Coudray et al., 2007a et b ). Nous présentons ici nos résultats d’analyse concernant les polymorphismes de l’ADN mitochondrial.
L’ADN mitochondrial (ADNmt) est une molécule circulaire de 16569pb présente dans les mitochondries de chaque cellule et de transmission uniquement maternelle. Son polymorphisme est défini par diverses mutations dans la séquence ADN (notamment dans les fragments hypervariables HVSI et HVSII de la région contrôle mais aussi dans la région codante). Les combinaisons de ces mutations permettent de décrire différents haplotypes que l’on peut regrouper, d’après leurs relations phylogénétiques, en haplogroupes. Ces haplogroupes mitochondriaux sont répartis en différentes familles très fréquentes dans certaines régions géographiques voire même dans certaines populations. Ainsi, les populations européennes ont des fréquences élevées d’haplogroupes H, HV, I, J, K, T, U, V, W et X. Les lignées fréquentes au Proche-Orient et au sud de l’Asie sont M et N (lignées ancestrales), E, F, G, P, Q, R, Y et Z. Une lignée est principalement détectée dans les populations nord-africaines : U6. Enfin, les régions sub-sahariennes présentent un pourcentage élevé d’haplogroupes de la famille L.
Divers travaux ont été conduits au nord de l’Afrique pour des populations du Maroc (Rando et al., 1998 ; Brakez et al., 2001 ; Kéfi et al., 2005), d’Algérie (Côrte-Real et al., 1996 ; Macaulay et al., 1999), de Tunisie (Fadhlaoui-Zid et al., 2004 ; Cherni et al., 2005 ; Loueslati et al., 2006), d’Egypte (Krings et al., 1999 ; Stevanovitch et al., 2004), ou plus globalement du pourtour méditerranéen (Plaza et al., 2003). Les auteurs montrent que les Berbères ont une structure mitochondriale composée aussi bien de lignées fréquemment observées en Europe (H, J, T, V), que d’haplogroupes présents à des taux élevés au Proche-Orient (M) et au sub-Sahara (L2, L3). L’haplogroupe U6 est détecté à des fréquences relativement élevées chez les Berbères du Maghreb, et jusqu’à 30% chez les Mozabites algériens.
Dans cette étude, nous verrons comment se situe la diversité mitochondriale des populations berbères parmi celle de populations géographiquement proches puis nous considèrerons plus précisément la distribution des haplogroupes mitochondriaux au sein des groupes berbérophones.
Matériels et Méthodes
Les populations berbérophones étudiées par notre équipe sont originaires du Maroc (Asni, Bouhria et oasis de Figuig) et d’Egypte (oasis de Siouah). Les individus ont été sélectionnés à partir d’une enquête linguistique réalisée sur le terrain. Il s’agit d’adultes sains, non apparentés et dont les quatre grands-parents sont nés dans la même région.
Les ADNs ont été directement extraits de prélèvements sanguins selon une méthode classique d’extraction au phénol-chloroforme. Des fragments de 892pb de la région contrôle de l’ADNmt (comprenant la séquence HVSI et une partie de la séquence HVSII) ont été amplifiés par PCR puis séquencés dans un Analyseur Génétique (Applied Biosystems). Des RFLPs ont ensuite été recherchés dans la région codante. L’assignation des haplotypes à un haplogroupe donné a été effectuée selon la classification de Torroni et al. (1996).
Notre base de données rassemble les fréquences des haplogroupes mitochondriaux de 41 populations (Figure 1) : 20 du nord de l’Afrique (Maroc, Algérie, Tunisie, Egypte, Mauritanie), 5 des régions sub-sahariennes (Cameroun, Angola, Kenya, Mozambique, Ethiopie), 6 du Proche- et Moyen-Orient (Yémen, Iraq, Israël, Jordanie, Turquie, Syrie) et 10 du sud de l’Europe (Grèce, Italie, Espagne, Portugal). Les références de chaque population sont : Asni, Bouhria, (Dugoujon et Torroni, non publié), Figuig (Dugoujon, non publié), Souss (Brakez et al., 2001), Arabes Maroc (Rando et al., 1998 ; Plaza et al., 2003), Saharaouis (Plaza et al., 2003), Ghardaïa (Macaulay et al., 1999), Algérie (Plaza et al., 2003), Douiret-Chenini, Sened, Matmata (Fadhlaoui-Zid et al., 2004), Berbères Jerba, Arabes Jerba (Loueslati et al., 2006), Arabes Tunisie (Plaza et al., 2003), Siouah (Dugoujon et Torroni, non publié), Gurna (Stevanovitch et al., 2004), Adaïma (Dugoujon, non publié), Mauritanie (Rando et al., 1998), Cameroun (Coia et al., 2005), Angola (Plaza et al., 2004), Kenya (Brandstatter et al., 2004), Mozambique (Pereira et al., 2001 ; Salas et al., 2002), Ethiopie, Yemen (Kivisild et al., 2004), Iraq, Israel, Jordanie (Richards et al., 2000), Syrie (Richards et al., 2000 ; Vernesi et al., 2001), Turquie (Richards et al., 2000 ; Di Benedetto et al., 2001 ; Quintana-Murci et al., 2004), Grèce (Richards et al., 2000 ; Vernesi et al., 2001), Italie (Torroni, non publié), Sardaigne, Sicile (Richards et al., 2000 ; Torroni, non publié), Basques (Richards et al., 2000), Espagne, Andalousie (Crespillo et al., 2000 ; Richards et al., 2000 ; Larruga et al., 2001 ; Plaza et al., 2003), Portugal (Pereira et al., 2000 ; Richards et al. 2000 ; González et al., 2003).
Pour les comparaisons entre populations, nous avons réalisé différentes Analyses en Composantes Principales (ACP). Ces analyses sont basées sur la projection graphique d’un dendrogramme construit à partir de la variabilité des fréquences des haplogroupes rencontrés dans l’ensemble des populations considérées.
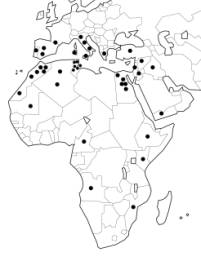
Figure 1. Localisation géographiques des populations incluses dans la base de données mitochondriales.
Figure 1. Geographical localization of the populations included in the mitochondrial database.
Résultats
La figure 2 présente une Analyse en Composantes Principales (ACP) portant sur les fréquences des haplogroupes mitochondriaux des 41 populations incluses dans la base de données (Coudray, 2006b) : 20 du nord de l’Afrique (Maroc, Algérie, Tunisie, Egypte, Mauritanie), 5 des régions sub-sahariennes (Cameroun, Angola, Kenya, Mozambique, Ethiopie), 6 du Proche- et Moyen-Orient (Yémen, Iraq, Israël, Jordanie, Turquie, Syrie) et 10 du sud de l’Europe (Grèce, Italie, Espagne, Portugal).
Cette ACP permet de mettre en évidence 3 ensembles de populations : un premier groupe, « (1) », comprend toutes les populations européennes et du Moyen-Orient ainsi que les populations nord-ouest africaines et les Berbères égyptiens de Siouah ; un deuxième groupe, « (2) », rassemble les trois populations de Haute Egypte (Adaïma), les Berbères de Sened (Tunisie) et la population d’Ethiopie ; à noter que la population du Yémen se situe entre ces deux premiers groupes avec un rapprochement avec le premier selon l’axe 1 et un rapprochement avec le second selon l’axe 2 ; enfin, un dernier ensemble, « (3) », regroupe les quatre populations sub-sahariennes du Cameroun, d’Angola, du Mozambique et du Kenya. Si on regarde la disposition des groupes on constate que les groupes « (1) » et « (3) » sont les plus différenciés et que le groupe « (2) » occupe une place intermédiaire. De plus, si on regarde plus précisément le groupe « (1) » on peut remarquer que les populations européennes se situent vers une extrémité de cet ensemble tandis que les populations nord-africaines sont à l’autre extrémité. Les haplogroupes mitochondriaux associés à cette disposition des populations sur l’ACP sont les haplogroupes fréquents en Europe (comprenant aussi les U6 détectés au nord de l’Afrique) pour le groupe « (1) », les haplogroupes M1 pour le groupe « (2) » et enfin les haplogroupes de la famille L pour le groupe « (3) ». En effet, on observe une nette différenciation entre les pools mitochondriaux sub-sahariens (sauf d’Ethiopie) composés de plus de 90% d’haplogroupes L et les patrimoines génétiques observés sur la côte nord de la Méditerranée composés de plus de 90% d’haplogroupes observés à des fréquences élevées en Europe ou Eurasie (H, HV, J, T, U*…). A ces gradients de fréquences nord-sud et sud-nord, s’ajoute le gradient des haplogroupes M1 (fréquents en Afrique de l’Est) d’ouest en est : 0 à 12% au Maghreb, 12 à 24% en Haute Egypte et 17% en Ethiopie.
Ainsi, cette première ACP montre : que les populations européennes et sub-sahariennes sont les plus génétiquement différenciées ; qu’il y a une différenciation entre la population d’Ethiopie et les autres populations sub-sahariennes et que les populations nord-africaines occupent une place intermédiaire : les groupes nord-ouest africains se rapprochent des populations européennes et du Moyen-Orient tandis que les populations de Haute Egypte présentent plus d’affinités avec les groupes sub-sahariens.
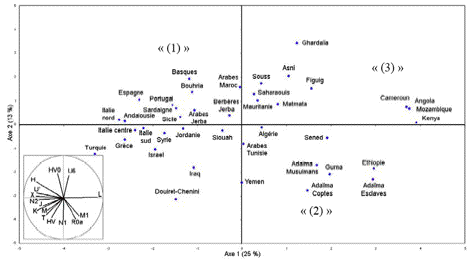
Figure 2. Analyse en Composantes Principales de la distribution des haplogroupes mitochondriaux de 41 populations africaines, proche-orientales et européennes. L’ACP a été tracée à partir des fréquences des haplogroupes mitochondriaux de 41 populations humaines d’Afrique (régions Nord et sub-Sahara), du Proche et Moyen-Orient et du sud de l’Europe (voir les références dans la partie Matériels et Méthodes). Les variables (haplogroupes) expliquant la disposition des populations le long des axes de l’ACP sont aussi reportées. La description des groupes de populations « (1) », « (2) » et « (3) » est présentée dans le texte.
Figure 2. Principal Component Analysis of the mitochondrial haplogroup distribution on 41 African, Near-Eastern and European populations. The PCA was drawn from the mitochondrial haplogroup frequencies of 41 human populations from Africa (Northern and sub-Saharan Africa), from Near-East and Middle-East and from southern Europe (for the references, see the Materials and Methods part). The variables (haplogroups) explaining the population locations along the axes are also showed. Description of the populations groups "(1)", "(2)" and "(3)" is presented in the text.
La 2ème étude réalisée concerne la distribution des haplogroupes mitochondriaux des 10 populations berbérophones de la base de données (Maroc, Algérie, Tunisie, Egypte). Quatre de ces populations ont été typées par notre équipe (Asni, Bouhria, Figuig et Siouah ; Dugoujon non publié). L’ACP permet de constater que selon l’axe 1, les Berbères tunisiens de Douiret-Chenini sont les plus différenciés, et que selon l’axe 2, on observe une différenciation entre les Berbères égyptiens de Siouah et les autres populations berbérophones (Figure 3). Cette disposition des populations sur l’ACP est expliquée par la distinction entre les haplogroupes HV, J, K, M, T, et U* et les haplogroupes HV0, L, M1, N1, N2, R0a, U6, et X. Ainsi, les Berbères de Douiret-Chenini se différencient des autres populations en raison d’une plus faible fréquence d’haplogroupes H mais aussi de fréquences plus élevées pour les familles K, HV et T.
Si on regarde la structure génétique mitochondriale générale des populations berbères, on constate qu’elle est composée majoritairement d’haplogroupes fréquents dans les populations européennes (de 44 à 84%), de 13 à 47% d’haplogroupes L très fréquents dans les populations sub-sahariennes, de 0 à 16% de M1 détectés principalement dans les populations est-africaines, de 0 à 28% de U6 (nord Afrique) et de 0 à 8% de M, N ou X (Eurasie).
Nous avons ensuite cherché à affiner cette différenciation mitochondriale entre les Berbères du Nord-Ouest et du Nord-Est, en réalisant un network pour chacune des 4 populations étudiées par notre équipe (résultats non montrés) : Asni (53 individus, 41 haplotypes différents), Bouhria (72 individus, 49 haplotypes), Figuig (115 individus, 41 haplotypes) et Siouah (80 individus, 26 haplotypes).
Quatre points essentiels sont à souligner sur ces networks, marquant la différenciation observée. Tout d’abord, les haplogroupes H fréquents dans les populations européennes ont été observés au Maroc autour de 24 à 38% alors qu’un seul individu porte cet haplogroupe à Siouah. Ensuite, les haplogroupes L pris dans leur ensemble ont une fréquence de 14 à 47% au Maroc et de 24% à Siouah. Leur répartition qualitative est aussi différente d’une région à l’autre. Par exemple L1b, observé dans les populations d’Afrique Centrale et Occidentale est plus fréquent au Maghreb (7 à 10%) qu’à Siouah (1%). A l’inverse, les haplogroupes L3i et L4g, rencontrés dans les populations est-africaines, sont détectés uniquement chez les Berbères égyptiens. Ensuite, l’haplogroupe M1 a une fréquence quatre fois plus élevée à Siouah (16%) qu’au Maroc. Enfin, l’haplogroupe U6a est observé seulement dans les populations marocaines.
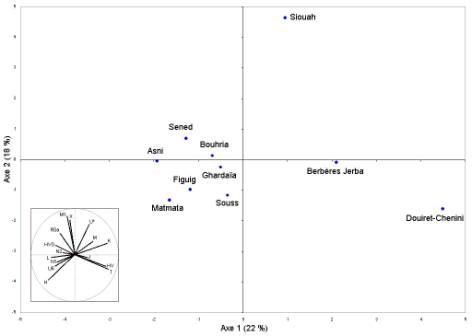
Figure 3. Analyse en Composantes Principales de la distribution des haplogroupes mitochondriaux de 10 populations berbérophones nord-africaines. L’ACP a été tracée à partir des fréquences des haplogroupes mitochondriaux de 10 populations berbérophones du Maroc (Asni, Bouhria, Figuig, Souss), d’Algérie (Ghardaïa), de Tunisie (Matmata, Jerba, Sened) et d’Egypte (Siouah). Pour les références des populations analysées, voir la partie Matériels et Méthodes. Les variables (haplogroupes) expliquant la disposition des populations le long des axes de l’ACP sont aussi reportées.
Figure 3. Principal Component Analysis of the mitochondrial haplogroup distribution on 10 Berber-speaking populations from North Africa. The PCA was drawn from the mitochondrial haplogroup frequencies of 10 Berber-speaking populations from Morocco (Asni, Bouhria, Figuig, Souss), Algeria (Ghardaïa), Tunisia (Matmata, Jerba, Sened) and Egypt (Siwa). For the population references, see the Materials and Methods part. The variables (haplogroups) explaining the population locations along the axes are also showed.
Discussion
Les premières analyses réalisées visaient à situer la diversité génétique des communautés nord-africaines dans un contexte géographique, notamment par comparaison avec des groupes du sud de l’Europe et des régions sahariennes et sub-sahariennes. Ainsi, l’analyse des 41 populations composant notre base de données génétiques a révélé que les populations berbères sont génétiquement proches des populations du sud de l’Europe, mais significativement différenciées des groupes sub-sahariens. Ces résultats s’expliquent par la structure du patrimoine génétique des Berbères qui est en effet caractérisé par un pourcentage élevé de certains haplogroupes rencontrés communément dans les populations européennes et d’une proportion moins importante de marqueurs fréquemment observés dans les populations sub-sahariennes.
La proximité génétique observée entre les Berbères et les populations du sud de l’Europe révèle que ces groupes partageaient un ancêtre commun. Deux hypothèses sont discutées. L’une daterait ces origines communes du Paléolithique supérieur avec l’expansion d’hommes anatomiquement modernes, depuis le Proche-Orient, et s’étendant le long des deux rives de la Méditerranée. L’autre hypothèse soutient ces origines proche-orientales, mais les dateraient plutôt du Néolithique, il y a 10.000 ans BP. Quoiqu’il en soit, les polymorphismes communs aux populations berbères et sud-européennes ont également pu être introduits ou maintenus par des flux géniques à travers le détroit de Gibraltar. Par exemple, des échanges ont pu avoir lieu au cours de la préhistoire, lors de l’expansion des populations européennes à partir du refuge franco-cantabrique, il y a 20.000 ans BP. Ou alors ces échanges ont pu se produire au cours de l’histoire, lors de l’invasion et l’occupation pendant près de sept siècles (du VIIIème au XVème siècle) de la péninsule Ibérique par des troupes berbères musulmanes, almoravides puis almohades.
A l’inverse, la différenciation observée entre les populations nord-africaines et sub-sahariennes montre d’une part, que le peuplement de ces régions s’est fait par des vagues de migrations différentes et, d’autre part, qu’une diversité génétique était déjà appréciable en Afrique depuis des temps très anciens. Se pose alors la question du rôle du Sahara dans les mouvements et les échanges entre les populations. Il faut préciser que le Sahara n’a pas toujours été un désert car il a également connu des périodes de forte humidité offrant des espaces de verdure propices à l’installation des hommes et à la domestication des animaux. Il y a donc très certainement eu des échanges entre les populations préhistoriques africaines, échanges au cours desquels des marqueurs plus fréquemment observés au sub-Sahara auraient été introduits dans le patrimoine génétique des Berbères. Des contacts entre le nord de l’Afrique et de grands empires sub-sahariens (tels ceux du Ghana, du Mali, ou encore l’empire Songhaï) sont également rapportés par l’histoire, lors de commerces transsahariens d’or, de sel et d’esclaves.
La deuxième partie de notre étude visait à mesurer les relations de parenté entre les communautés berbères sur l’ensemble de leur domaine de répartition. Ainsi, nos résultats ont mis en évidence une différenciation génétique entre les Berbères du Maghreb et les Berbères d’Egypte. Cette différenciation semblerait être le résultat de la conjonction de plusieurs facteurs géographiques, préhistoriques et historiques qui ont guidé les contacts (et donc les échanges) entre les populations locales et les groupes migrants. Effectivement, la localisation géographique des populations est très différente : les Berbères du Maghreb sont au bout d’un long couloir de migration, alors que les Berbères de Siouah sont plutôt à un carrefour entre le Proche-Orient, l’Afrique de l’Est, le sub-Sahara et le couloir nord-africain. Ainsi, selon cette localisation géographique, les rencontres et les échanges entre les populations locales et migrantes n’ont donc pas été les mêmes au Nord-Ouest et au Nord-Est de l’Afrique. De plus, la préhistoire et l’histoire des populations du Maghreb sont différentes de celles du groupe égyptien. On a très peu de renseignements sur l’oasis de Siouah à l’époque Néolithique mais on suppose que cette période a été, comme pour le Maghreb, le point de départ de la culture berbère. Puis, au cours de l’histoire, l’oasis a vu se succéder des groupes humains venus de toutes parts, comme des pèlerins se rendant à la Mecque, des commerçants méditerranéens ou des marchands d’esclaves en provenance des régions sahéliennes. Siouah a également été repeuplée par des Berbérophones libyens chassés de leurs terres par les conquérants arabes. Enfin, elle a du faire face, entre les IXème et XIIème siècles, à une réduction démographique drastique de sa population. Le pool génique actuel des Siwi serait donc le résultat de ces différents échanges génétiques survenus dans le Passé.
Finalement, notre étude des populations berbères montre que leurs origines et leur diversité sont très anciennes et que ces communautés ont des traits génétiques particuliers résultant de divers échanges avec les populations migrantes au cours de la préhistoire et/ou de la période historique.
Remerciements. Nous remercions toutes les personnes et infrastructures qui ont participé à la collecte des échantillons et à l’acquisition des résultats. Ces travaux s’inscrivent dans le cadre du projet EUROCORES de l’ESF (European Science Foundation), contrat no. ERAS-CT-2003-980409 et bénéficient de financements du CNRS (Centre National de la Recherche Scientifique) et du Conseil Régional de Midi-Pyrénées (Toulouse, France).
Bibliographie
Brakez, Z., Bosch, E., Izaabel, H., Akhayat, O., Comas, D., Bertranpetit, J., Calafell, F., 2001, Human mitochondrial DNA sequence variation in the Moroccan population of the Souss area. Annals of Human Biology, 28(3), 295-307.
Brandstatter, A., Peterson, C.T., Irwin, J.A., Mpoke, S., Koech, D.K., Parson, W., Parsons, T.J., 2004, Mitochondrial DNA control region sequences from Nairobi (Kenya): inferring phylogenetic parameters for the establishment of a forensic database. International Journal of Legal Medicine, 118(5), 294-306.
Cherni, L., Loueslati, B.Y., Pereira, L., Ennafaa, H., Amorim, A., El Gaaied, A.B., 2005, Female gene pools of Berber and Arab neighboring communities in central Tunisia: microstructure of mtDNA variation in North Africa. Human Biology, 77(1), 61-70.
Coia, V., Destro-Bisol, G., Verginelli, F., Battaggia, C., Boschi, I., Cruciani, F., Spedini, G., Comas, D., Calafell F., 2005, Brief communication: mtDNA variation in North Cameroon: lack of Asian lineages and implications for back migration from Asia to sub-Saharan Africa. American Journal of Physical Anthropology, 128(3), 678-681.
Côrte-Real, H.B., Macaulay, V.A., Richards, M.B., Hariti, G., Issad, M.S., Cambon-Thomsen, A., Papiha, S., Bertranpetit, J., Sykes, B.C., 1996, Genetic diversity in the Iberian Peninsula determined from mitochondrial sequence analysis. Annals of Human Genetics, 60(Pt 4), 331-350.
Coudray, C., Guitard, E., Kandil, M., Harich, N., Melhaoui, M., Baali, A., Sevin, A., Moral, P., Dugoujon, J.M., 2006a, Study of GM immunoglobulin allotypic system in Berbers and Arabs from Morocco. American Journal of Human Biology, 18(1), 23-34.
Coudray, C., 2006b, Histoire génétique et évolution des populations berbérophones nord-africaines. Thèse de Doctorat, Laboratoire CNRS d’Anthropologie biologique, Université Toulouse III.
Coudray, C., Guitard, E., Keyser-Tracqui, C., Melhaoui, M., Cherkaoui, M., Larrouy, G., Dugoujon, J.M., 2007a, Population genetic data of 15 tetrameric short tandem repeats (STRs) in Berbers from Morocco. Forensic Science International, 167(1), 81-86.
Coudray, C., Guitard, E., El-Chennawi, F., Larrouy, G., Dugoujon, J.M., 2007b, Allele frequencies of 15 short tandem repeats (STRs) in three Egyptian populations of different ethnic groups. Forensic Science International, 169(2-3), 260-265.
Crespillo, M., Luque, J.A., Paredes, M., Fernandez, R., Ramirez, E., Valverde, J.L., 2000, Mitochondrial DNA sequences for 118 individuals from northeastern Spain. International Journal of Legal Medicine, 114(1-2), 130-132.
Cruciani, F., La Fratta, R., Santolamazza, P., Sellitto, D., Pascone, R., Moral, P., Watson, E., Guida, V., Coloma, E.B., Zaharova, B., Lavinha, J., Vona, G., Aman, R., Cali, F., Akar, N., Richards, M., Torroni, A., Novelletto, A., Scozzari, R., 2004, Phylogeographic analysis of haplogroup E3b (E-M215) Y chromosomes reveals multiple migratory events within and out of Africa. American Journal of Human Genetics, 74(5), 1014-1022.
Di Benedetto, G., Erguven, A., Stenico, M., Castri, L., Bertorelle, G., Togan, I., Barbujani, G., 2001, DNA diversity and population admixture in Anatolia. American Journal of Physical Anthropology, 115(2), 144-156.
Dugoujon, J.M., Hazout, S., Loirat, F., Mourrieras, B., Crouau-Roy, B., Sanchez-Mazas, A., 2004, GM haplotype diversity of 82 populations over the world suggests a centrifugal model of human migrations. American Journal of Physical Anthropology, 125, 175-192.
Fadhlaoui-Zid, K., Plaza, S., Calafell, F., Ben Amor, M., Comas, D., Bennamar El gaaied, A., 2004, Mitochondrial DNA heterogeneity in Tunisian Berbers. Annals of Human Genetics. 68(Pt3), 222-233.
Gonzalez, A.M., Brehm, A., Perez, J.A., Maca-Meyer, N., Flores, C., Cabrera, V.M., 2003, Mitochondrial DNA affinities at the Atlantic fringe of Europe. American Journal of Physical Anthropology, 120(4), 391-404.
Kéfi, R., Stevanovitch, A., Bouzaid, E., Béraud-Colomb, E., 2005, Diversité mitochondriale de la population de Taforalt (12.000 ans BP – Maroc): une approche génétique à l’étude du peuplement de l’Afrique du Nord. Anthropologie, 43, 1-11.
Kivisild, T., Reidla, M., Metspalu, E., Rosa, A., Brehm, A., Pennarun, E., Parik, J., Geberhiwot, T., Usanga, E., Villems, R., 2004, Ethiopian mitochondrial DNA heritage: tracking gene flow across and around the gate of tears. American Journal of Human Genetics, 75(5), 752-770.
Krings, M., Salem, A.E., Bauer, K., Geisert, H., Malek, A.K., Chaix, L., Simon, C., Welsby, D., Di Rienzo, A., Utermann, G., Sajantila, A., Paabo, S., Stoneking, M., 1999, mtDNA analysis of Nile River Valley populations: A genetic corridor or a barrier to migration? American Journal of Human Genetics, 64(4), 1166-1176.
Larruga, J.M., Diez, F., Pinto, F.M., Flores, C., Gonzalez, A.M., 2001, Mitochondrial DNA characterisation of European isolates: the Maragatos from Spain. European Journal of Human Genetics, 9(9), 708-716.
Loveslati, B.Y., Cherni, L., Khodjet-Elkhil, H., Ennafaa, H., Pereira, L., Amorim, A., Ben Ayed, F., Ben Ammar Elgaaied, A., 2006, Islands inside an island: reproductive isolates on Jerba island. American Journal of Human Biology, 18(1), 149-153.
Macaulay, V., Richards, M., Hickey, E., Vega, E., Cruciani, F., Guida, V., Scozzari, R., Bonne-Tamir, B., Sykes, B., Torroni, A., 1999, The emerging tree of West Eurasian mtDNAs: a synthesis of control-region sequences and RFLPs. American Journal of Human Genetics, 64(1), 232-249.
Olivieri, A., Achilli, A., Pala, M., Battaglia, V., Fornarino, S., Al-Zahery, N., Scozzari, R., Cruciani, F., Behar, D.M., Dugoujon, J.M., Coudray, C., Santachiara-Benerecetti, A.S., Semino, O., Bandelt, H.J., Torroni, A., 2006, The mtDNA legacy of the Levantine Early Upper Palaeolithic in Africa. Science, 314(5806), 1767-1770.
Pereira, L., Prata, M.J., Amorim, A., 2000, Diversity of mtDNA lineages in Portugal: not a genetic edge of European variation. Annals of Human Genetics, 64(Pt 6), 491-506.
Pereira, L., Macaulay, V., Torroni, A., Scozzari, R., Prata, M.J., Amorim, A., 2001, Prehistoric and historic traces in the mtDNA of Mozambique: insights into the Bantu expansions and the slave trade. Annals of Human Genetics, 65(Pt 5), 439-458.
Plaza, S., Calafell, F., Helal, A., Bouzerna, N., Lefranc, G., Bertranpetit, J., Comas, D., 2003, Joining the pillars of Hercules: mtDNA sequences show multidirectional gene flow in the western Mediterranean. Annals of Human Genetics, 67(Pt 4), 312-328.
Plaza, S., Salas, A., Calafell, F., Corte-Real, F., Bertranpetit, J., Carracedo, A., Comas, D., 2004, Insights into the western Bantu dispersal: mtDNA lineage analysis in Angola. Human Genetics, 115(5), 439-447.
Quintana-Murci, L., Chaix, R., Wells, R.S., Behar, D.M., Sayar, H., Scozzari, R., Rengo, C., Al-Zahery, N., Semino, O., Santachiara-Benerecetti, A.S., Coppa, A., Ayub, Q., Mohyuddin, A., Tyler-Smith, C., Qasim Mehdi, S., Torroni, A., McElreavey, K., 2004, Where west meets east: the complex mtDNA landscape of the Southwest and Central Asian corridor. American Journal of Human Genetics, 74(5), 827-845.
Rando, J.C., Pinto, F., Gonzalez, A.M., Hernandez, M., Larruga, J.M., Cabrera, V.M., Bandelt, H.J., 1998, Mitochondrial DNA analysis of Northwest African populations reveals genetic exchanges with European Near-Eastern and sub-Saharan populations. Annals of Human Genetics, 62(Pt 6), 531-550.
Richards, M., Macaulay, V., Hickey, E., Vega, E., Sykes, B., Guida, V., Rengo, C., Sellitto, D., Cruciani, F., Kivisild, T., Villems, R., Thomas, M., Rychkov, S., Rychkov, O., Rychkov, Y., Golge, M., Dimitrov, D., Hill, E., Bradley, D., Romano, V., Cali, F., Vona, G., Demaine, A., Papiha, S., Triantaphyllidis, C. Stefanescu, G., Hatina, J., Belledi, M., Di Rienzo, A., Novelletto, A., Oppenheim, A., Norby, S., Al-Zaheri, N., Santachiara-Benerecetti, A.S., Scozari, R., Torroni, A., Bandelt, H.J., 2000, Tracing European founder lineages in the Near Eastern mtDNA pool. American Journal of Human Genetics, 67(5), 1251-1276.
Salas, A., Richards, M., De la Fe, T., Lareu, M.V., Sobrino, B., Sanchez-Diz, P., Macaulay, V., Carracedo, A., 2002, The making of the African mtDNA landscape. American Journal of Human Genetics, 71(5), 1082-1111.
Stevanovitch, A., Gilles, A., Bouzaid, E., Kefi, R., Paris, F., Gayraud, R.P., Spadoni, J.L., El-Chenawi, F., Beraud-Colomb, E., 2004, Mitochondrial DNA sequence diversity in a sedentary population from Egypt. Annals of Human Genetics, 68(Pt 1), 23-39.
Torroni, A., Huoponen, K., Francalacci, P., Petrozzi, M., Morelli, L., Scozzari, R., Obinu, D., Savontaus, M.L., Wallace, D.C., 1996, Classification of European mtDNAs from an analysis of three European populations. Genetics, 144(4), 1835-1850.
Vernesi, C., Di Benedetto, G., Caramelli, D., Secchieri, E., Simoni, L., Katti, E., Malaspina, P., Novelletto, A., Marin, V.T., Barbujani, G., 2001, Genetic characterization of the body attributed to the evangelist Luke. Proceedings of the National Academy of Sciences of the United States of America, 98(23), 13460-13463.