
El Ossmani, H., Talbi, J., Bouchrif, B., Chafik, A., 2008, Exploitation
de 15 STRs autosomaux pour l’étude phylogénétique de la population Arabophone
de Rabat-Salé-Zemmour-Zaer (Maroc), Antropo,
17, 15-23. www.didac.ehu.es/antropo
Exploitation de 15 STRs
autosomaux pour l’étude phylogénétique de la population Arabophone de
Rabat-Salé-Zemmour-Zaer (Maroc)
Exploitation
of 15 autosomal STRs in the phylogenetic study of the Arabic-speaking
population of Rabat-Salé-Zemmour-Zaer (Morocco)
Hicham El Ossmani1,2, Jalal Talbi1, Brahim
Bouchrif3, Abdelaziz Chafik1
1Laboratoire d’Anthropogénétique et de Physiopathologie,
Département de Biologie, Faculté des
Sciences, Université Chouaïb Doukkali, El Jadida. Maroc
2Laboratoire de Génétique, Gendarmerie Royale, avenue Ibn
Sina 10100, Rabat, Maroc
3Laboratoire de Biologie Moléculaire, Institut Pasteur, 1
place Luis Pasteur, Casablanca
Adresse de
correspondance: Hicham El
Ossmani, Laboratoire de Génétique de la Gendarmerie Royale, Avenue Ibn Sina
10100, Rabat, Maroc. E-mail: helossmani@yahoo.fr
Mots-clés: Rabat-Salé-Zemmour-Zaer; Arabophone; Phylogénétique; STR; Histoire; contexte Afro-méditerranéenne; contexte mondial
Keywords: Rabat-Salé-Zemmour-Zaer; Arabic-speaking; Phylogenetic; STR; History;
Afro-Mediterranean context; worldwide context
Résumé
La volonté de classer les populations humaines actuelles n’est pas récente dans l’histoire de l’anthropologie. Aujourd’hui la réalisation d’une construction phylogénétique associe le critère géographique aux critères anatomiques de l’anthropologie physique et, depuis peu, aux considérations historiques, ethnologiques et linguistiques. Dans ce contexte nous avons réalisé une étude phylogénétique régionale et mondiale de la population arabophone marocaine de Rabat-Salé-Zemmour-Zaer en utilisant les fréquences allèliques des 15 STRs du kit Identifiler (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179,TH01, vWA, D13S317, D16S539, D18S51, D2S1338, D19S433 et D21S11) susceptibles de reconstituer l’histoire des flux géniques qui ont alimenté le substratum génétique dans cette région qui abrite l’un des plus anciens sites archéologiques (Harhoura). La population arabophone de Rabat-Salé-Zemmour-Zaer présente une similitude génétique aux Andalous à l’échelle Afro-méditerranéenne et des rapprochements génétique aux populations du Moyen Orient dans un contexte mondial plus large. Les événements historiques des invasions islamiques au Maroc et de l’expulsion des arabes de la Péninsule ibérique semblent définir de près cette structure.
Abstract
Classification of
the actual human populations is not recent in the history of Anthropology.
Today the realization of a phylogenetic structure links the geographical
criterion to anatomic criteria of physical anthropology and, lately, to
historical, ethnological and linguistic considerations. In this context we
accomplished a regional and worldwide phylogenetic study of the Arabic-speaking
population of Rabat-Salé-Zemmour-Zaer in Morocco, by using allelic frequencies
of 15 STRs of Identifiler kit (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820,
D8S1179, TH01, vWA, D13S317, D16S539, D18S51, D2S1338, D19S433 and D21S11).
These STR are likely to reconstruct the history of gene fluxes that fed the
genetic substratum in this region that shelters one of the most ancient
archeological sites (Harhoura). The Arabic-speaking population of
Rabat-Salé-Zemmour-Zaer presents a genetic similarity with the Andalusia’s
population at the Afro-Mediterranean context and with the Middle East
populations at a larger worldwide context. The historic events of Islamic
plagues in Morocco and of the expulsion of Arabs from the Iberian Peninsula
seem to define, closely, this structure.
Introduction
Le brassage des gènes entre les différentes
populations mondiales a suscité l’intérêt des anthropologues depuis l’émergence
de la théorie de l’effet fondateur et de celle de la dérive génétique.
Plusieurs types d’informations et différentes méthodes permettent d’apprécier
l’hétérogénéité au sein d’une population (Verrier et al, 2005). Les études
portaient et portent encore sur l’origine géographique des individus pour
définir l’état du substratum génétique des populations et apprécier les
affinités génétiques entre les groupes. En effet, les groupes géographiquement
proches sont aussi génétiquement similaires et lorsque la distance géographique
augmente, la différence génétique entre les populations devient plus importante
(Calafell et al. 2000; Rosenberg et al. 2002). Avec le progrès technologique et
scientifique réalisé en biologie moléculaire l’appréciation de l’état du pool
génétique et de la mobilité des gènes s’effectue directement au niveau de l’ADN
en exploitant les séquences marqueurs telles que les STR. Récemment nous avons
exploité 15 STRs autosomaux pour définir la structure génétique de la
population marocaine arabophone de Rabat-Salé-Zemmour-Zaer (El Ossmani et al,
2007).
En faisant toujours appel aux même STR nous
nous proposons dans le présent travail de situer cette population marocaine par
rapport aux populations mondiales. En effet, par rapport au reste des
populations qui définissent la carte phylogénétique mondiale, la population
marocaine occupe plutôt une situation particulière de par sa mosaïque ethnique.
Avec une histoire profonde de métissage alimentée par les flux migratoires
divers et intenses qui ont réaménagé le substratum de la population dite
autochtone (berbère), les barrières génétiques entre les différents groupes
ethniques qui constituent cette population et encore entre celle-ci et les
populations avoisinantes sont devenues très floues. Les facteurs socioculturels
sont, ainsi, devenu encore plus
discriminants que la biologie et, désormais, en parle d’arabophones, de
berbérophones, d’hispaniques…
L’hétérogénéité des flux migratoires se
traduit par l’instabilité de la position phylogénétique de la population
marocaine lors des travaux fragmentaires antérieurement réalisés sur cette
population qui tantôt se présente proche du contexte méditerranéen avec des affinités
aux populations du nord et tantôt rejoigne les populations du proche et moyen
orient. Par rapport à une population qui a tendance à conserver son patrimoine
génétique via un comportement matrimonial endogame et consanguin sous le
témoignage des études récemment effectuées par Talbi et al en 2006 et 2007 à
travers le territoire marocain, serait-ce l’histoire qui gère le plus les
affinités de cette population vis-à-vis les autres populations mondiales. La
confrontation d’une approche historique à l’approche anthropogénétique
entreprise lors de cette étude pourrait confirmer cette hypothèse.
Population et Méthodes
1.
Histoire de la région
Rabat (en Arabe « Ar-Ribat ») est la capitale politique et
administrative du Maroc. Elle est située sur le littoral Atlantique du pays,
sur la rive gauche de l'embouchure du Bouregreg, en face de la ville de Salé.
Elle compte plus de 1,7 million d'habitants.
Des peuplements sont attestés sur le site de Rabat depuis l’Antiquité. La ville, à proprement parler, a été fondée en 1150 par le sultan almohade Abd Al-Mumin. Il y édifia une citadelle (future Kasbah des « Oudaïa »), une mosquée et une résidence. C’est alors ce qu'on appelle un « Ribat », une forteresse. Le nom actuel vient de Ribat Al Fath, « le camp de la victoire ». C’est le petit-fils d’Abd Al-Mumin, Yaequob al-Mansor, qui agrandit et complète la ville, lui donnant notamment des murailles. Par la suite, la ville a servi de base aux expéditions almohades en Andalousie.
A partir de 1610, Rabat reçut une forte population de réfugiés musulmans chassés d’Al-Andalus qui s’établirent dans la Kasbah et à l'intérieur de l'enceinte almohade, dans la partie Nord-Ouest, qu'ils délimitèrent et protégèrent par une nouvelle enceinte, la muraille andalouse.
Pendant quelques dizaines d’années, Rabat, alors
connue de l’Europe sous le nom de « Salé-le-Neuf », fut le siège d'une petite république
maritime, la République du Bouregreg, jusqu’à l’avènement des Alaouites qui s’emparèrent de l’estuaire en 1666. Sa
principale activité était, alors, la course en mer contre les Chrétiens qui lui
procurait la totalité de ses ressources et Salé-le-Neuf devient le premier port du Maroc. Les
descendants de ces Andalous, qui portent souvent des patronymes à consonance
castillane tels que Mouline
(Molina), Bargach (Vargas), Moreno, Balafrej, Ronda, etc., sont toujours considérés comme les
Rbatis dits « de souche ».
2.
Echantillonnage
L’objectif était d’obtenir un nombre suffisant de sujets d’origine de Rabat Salé Zemmour Zaer sans lien de parenté avec un sexe ratio équilibré. Nous avons, ainsi, recueilli des informations sur l’origine de chaque individu, de ses parents, de ses grands-parents et de ses arrières grands parents, son lieu de naissance, sa langue et, bien entendu, son identité. Ensuite, 5 ml de sang ont été prélevés de chaque individu dans des tubes à EDTA. 387 individus ont, ainsi, participé à l’étude.
3.
Analyse génétique
L’ADN a été extrait à partir des échantillons de sang en utilisant la méthode organique du Phénol-chloroforme. La quantification d’ADN est réalisée par la technique de la PCR en temps réel par le kit Quantifiler (Applied Biosystems, Foster City, CA).
Les 15 marqueurs STR à analyser du kit Identifiler (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179, TH01, vWA, D13S317, D16S539, D18S51, D2S1338, D19S433 et D21S11) sont amplifiés par la technique de PCR. Au terme de la réaction d’amplification, l'ADN amplifié est soumis à l'électrophorèse et les allèles des différents microsatellites sont séparés en fonction de leur taille à l’aide d’un analyseur ABI Prism 3130xl (Applied Biosystems, Foster City, CA).
4. Analyse
statistique
La projection bidimensionnelle des fréquences alléliques lors de l’analyse en composantes principales a été effectuée avec la macro Excel XLSTAT V.7.5.2 (Addinsoft, http://www.xlstat.com). Les arbres phylogénétiques ont été établis avec le programme Neighbor-Joining du logiciel Phylip 3.67 (Felsenstein, 2007). La robustesse topologique des arbres a été testée avec 1000 bootstrap en faisant appel au même logiciel. Les fréquences alléliques sont accessibles sur notre travail antérieur publié dans la revue Antropo (El Ossmani et al, 2007). Les fréquences alléliques de 30 populations mondiales ont été introduites dans l’analyse (Tableau 1).
|
Populations |
Effectifs |
Références |
|
|
Afrique du
Nord |
Berbères de
Bouhria Maroc |
104 |
Coudray et
al.2007 |
|
Berbères
d’Asnie Maroc |
105 |
||
|
Berbères de
Siwa Egypte |
98 |
||
|
Musulmans
d’Egypte |
99 |
||
|
Copts
d’Adaima Egypte |
100 |
||
|
Afrique sub-saharienne |
Guinée
Equatorial |
134 |
Cíntia Alves et al. 2005 |
|
Angola
(Cabinda) |
110 |
Beleza et al. 2004 |
|
|
Mozambique |
135-144 |
Alves et al. 2004 |
|
|
Tutsi Rwanda |
108-126 |
Regueiro et al. 2004 |
|
|
Moyen Orient |
EAU (Dubai) |
224 |
Alshamali et al. 2005 |
|
Arabie
Saoudite |
94 |
||
|
Oman |
79 |
||
|
Yémen |
101 |
||
|
Iraq |
103 |
Filippo et
al. 2007 |
|
|
Iran |
150 |
E.M. Shepard
et R.J.Herrera.2006 |
|
|
Asie de
l’Est |
Inde |
317 |
Bindu et
al.2007 |
|
Bangladesh |
127 |
Yuji et al.
2005 |
|
|
Chine |
200 |
Yang et al.
2005 |
|
|
Corée |
231 |
Yoo.Li.Kim et al.2003 |
|
|
Taiwan |
597 |
Chih.Wi.Wang et al. 2003 |
|
|
Thailande |
210 |
Rerkamnuaychoke
et al. 2006 |
|
|
Europe |
Andalousie Espagne |
114 |
Coudray et
al. 2007 |
|
Autochtones
de l’Espagne |
342 |
Camatcho et
al. 2007 |
|
|
Belgique |
100 |
R.Decorte et
al.2003 |
|
|
Belarusse |
176 |
K. Rebała
et al. 2007 |
|
|
Macédonie |
100 |
D.Havas et
al.2007 |
|
|
Amérique
Latine |
Mexique |
180 |
A.Gorostiza
et al. 2007 |
|
Porto Rico |
205 |
J.Zuniga et
al. 2006 |
|
|
Costa Rica |
191-500 |
A.Rodriguez
et al.2007 |
|
|
Venezuela |
203 |
L.P.Bernal
et al. 2006 |
|
Tableau 1. Les populations mondiales introduites dans
l’étude
Table 1. The Worldwide
populations introduced in the study
Résultats et discussion
L’étude a été entreprise dans deux contextes d’ampleurs différentes:
1: Un contexte Afro-Méditerranéen: Pour une analyse régionale visant à situer génétiquement cette population par rapport aux populations voisines avec lesquelles elle partage des traits ou barrières culturels, sociaux et /ou géographiques.
2: Un contexte mondial visant à réaménager la structure génétique établie dans le contexte régional par rapport aux échanges inter-populationnels qui ont eu lieu au cours de l’histoire de l’humanité tout en dépistant d’éventuels apports d’autres populations dans le patrimoine génétique de la population de Rabat-Salé-Zemmour-Zaer traduit par un repositionnement phylogénétique de celle-ci.
Pour l’étude à l’échelle régionale, les fréquences alléliques de quatre populations sub-sahariennes, six populations Nord-Africaines et trois populations du nord du bassin méditerranéen ont été introduites à l’analyse. La figure 1 présente les résultats de l’analyse en composantes principales réalisée à partir de ces fréquences. Les deux premiers axes du graphique représentent 57,53% de la variance totale. La dispersion des nuages de points traduit une structuration claire des populations en deux groupes distincts. Le premier groupe renferme les populations sub-sahariennes reflétant, ainsi, leur homogénéité génétique et à titre parallèle leur situation géographique par rapport aux reste des populations. Les populations Nord-Africaines s’organisent au sein du même groupe à côté des populations Nord-Méditerranéennes témoignant, ainsi, d’une grande affinité génétique traduisant à la fois le rapprochement géographique, socioculturel et historique. Au sein de ce même groupe on assiste à une structuration des populations en deux sous-goupes relativement distincts. Le premier sous-groupe est constitué des populations Nord-Africaines, à l’exception de la population de Rabat-Salé-Zemmour-Zaer qui se positionne à côté des populations Nord-Méditerranéennes au sein du deuxième sous-groupe. Au-delà de la proximité géographique, le contexte historique semble présenter une explication encore plus fiable à cette affinité génétique. En effet, une forte migration des musulmans Andalous vers la région de Rabat-Salé-Zemmour-Zaer a eu lieu en 1610. L’arbre phylogénétique établi à partir des fréquences alléliques (figure 2) explicite fidèlement cette structure avec toujours une population de Rabat-Salé-Zemmour-Zaer plus proche des populations Nord-Méditerranéennes.
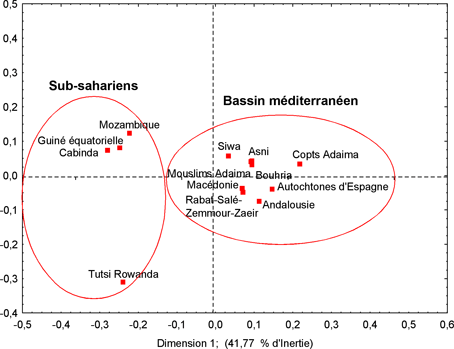
Figure 1. Analyse en composantes principales des 15 STRs chez la population
arabophone de Rabat-Salé-Zemmour-Zaer à l’échelle Afro-Méditerranéenne
Figure 1. Principal
components analysis of the 15 STRs in the Arabic-speaking population of
Rabat-Salé-Zemmour-Zaer in the Afro-Mediterranean context

Figure 2. Arbre phylogénétique des 15 STRs chez la population arabophone de
Rabat-Salé-Zemmour-Zaer à l’échelle Afro-Méditerranéenne
Figure 2. Phylogenetic tree
of the 15 STRs in the Arabic-speaking population of
Rabat-Salé-Zemmour-Zaer in the Afro-Mediterranean context
La figure 3 présente le résultat de l’analyse en composantes principales effectuée après l’introduction des fréquences alléliques de 18 autres populations dans le cadre d’une étude à l’échelle mondiale. Les deux premiers axes du graphique représentent 54,88% de la variance totale. A l’issue de cette analyse on assiste à une structure plus ou moins différente de celle établie à l’échelle régionale. Cette restructuration traduit, en effet, l’impact de l’alternance historique des différentes affinités génétiques qui ont eu lieu entre les différentes populations mondiales. Cette alternance elle-même étant le fruit des remaniements de la carte socioculturelle, économique et géopolitique du monde, ainsi que du progrès technologique que celui-ci a connu. En considérant les grands groupes, les populations du Moyen Orient et du Nord d’Afrique occupent une situation centrale par rapport aux populations de l’Asie de l’Est, celles du Nord de la méditerranée et de l’Amérique latine, et des populations sub-sahariennes. Cette disposition reflète l’importance de la proximité géographique et culturelle (notamment la religion). C’est le cas de l’inde, du Bangladesh, des Andalous, du Porto-Rico et du Mozambique qui témoignent d’une certaine affinité par rapport aux populations du Moyen Orient et du Nord de l’Afrique. Cette structure concorde, en effet, parfaitement avec les résultats de Coudray (2006).
En considérant les populations, la population de Rabat-Salé-Zemmour-Zaer occupe une position centrale et semble retrouver sa position équilibrée après avoir introduit les populations mondiales. En effet, contrairement au contexte régional, la population de Rabat-Salé-Zemmour-Zaer s’est détachée des populations Nord-Méditerranéennes pour rejoindre les populations du Moyen Orient et du Nord de l’Afrique. L’ancienneté de l’effet fondateur joue un rôle important dans ce repositionnement. En effet, les arabes fondateurs de la population Rabat-Salé-Zemmour-Zaer ne sont autres que les musulmans qui ont migré depuis le Moyen Orient pour s’installer dans la région de Rabat-Salé-Zemmour-Zaer avant 1150, soit environ cinq siècle avant l’arrivée des réfugiés musulmans de l’Andalousie. L’arbre phylogénétique établi confirme la structure révélée lors de l’analyse en composantes principales, avec les populations du Moyen Orient et du Nord de l’Afrique toujours en position intermédiaire (Figure 4).
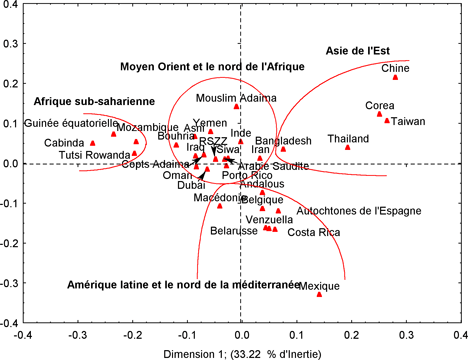
Figure 3. Analyse
en composantes principales des 15 STRs chez la population arabophone de
Rabat-Salé-Zemmour-Zaer à l’échelle mondiale
Figure 3. Principal
components analysis of the 15 STRs in the Arabic-speaking population of
Rabat-Salé-Zemmour-Zaer in the Worldwide context
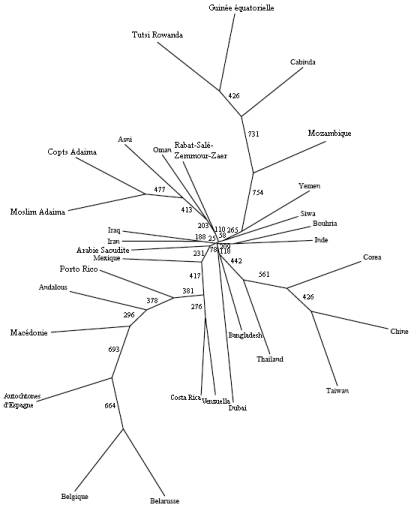
Figure 4. Arbre phylogénétique des 15 STRs chez la population arabophone de
Rabat-Salé-Zemmour-Zaer à l’échelle mondiale
Figure 4.
Phylogenetic tree of the 15 STRs in the Arabic-speaking population of Rabat-Salé-Zemmour-Zaer
in the Worldwide context
Conclusion
Le substratum génétique de la population Rabat-Salé-Zemmour-Zaer présente une continuité par rapport à ceux de la population des Andalous, les populations du Moyen Orient et les populations Nord-Africaines. Ce rapprochement s’inscrit dans un contexte historique et géopolitique pour la population des Andalous (récupération de l’Andalousie par l’Espagne), dans un contexte historique et culturel pour les populations du Moyen Orient (Invasions « Al-Fotouhat » islamiques) et dans un cadre géographique et culturel pour les populations du Nord de l’Afrique (Partage du milieu). Par ailleurs, au-delà de leur usage en criminalistique, les 15 STRs exploités dans la présente étude prouvent, encore une fois leur utilité et fiabilité dans l’établissement des structures génétiques des populations à travers l’étendu important de leur polymorphisme.
Références bibliographiques
Addinsoft, http://www.xlstat.com
Alshamali,
F., Alkhayat, A.Q., Budowle, B. et Watson, N.D., 2005, STR population diversity
in nine ethnic populations living in Dubai. Forensic Sci Int. 152(2-3),
267-279.
Alves, C., Gusmao, L., Damasceno, A., Soares,
B., et Amorim, A., 2004, Contribution for an African autosomic STR database
(AmpF/STR Identifiler and Powerplex 16 System) and a report on genotypic
variations. Forensic Sci Int. 139(2-3), 201-205.
Alves, C., Gusmao, L., Ana Lopez-Parra M.,
Soledad Mesa, M., Antonio Amorim, A. et
Arroyo-Pardob, E., 2005, STR allelic frequencies for an African
population sample (Equatorial Guinea) using AmpFlSTR Identifiler and Powerplex
16 kits Forensic Science International 148, 239–242.
Applied Biosystems, 2001, AmpFl STR®
IdentifilerTM PCR Amplification Kit User's Manual, Foster City, CA, P/N
4323291.
Barni,
F., Berti, A., Pianese, A., Boccellino, A., Miller, M.P., Caperna, A. et Lago,
G., 2007, Allele frequencies of 15 autosomal STR loci in the Iraq population
with comparisons to other populations from the middle-eastern region. Forensic
Sci. Int., 167, 87–92.
Beleza, S., Alves, C., Reis, F., Amorim, A., Carracedo, A. et Gusmao,
L., 2004, 17 STR data (AmpF/STR
Identifiler and Powerplex 16 System) from Cabinda (Angola). Forensic Sci
Int. 141(2-3), 193-196.
Bernal,
LP., Borjas, L., Zabala, W., Portillo, MG., Fernandez, E., Delgado, W., Tovar,
F., Lander, N., Chiurillo, MA., Ramirez, JL. et Garcia, O., Genetic variation
of 15 STR autosomal loci in the Maracaibo population from Venezuela, Forensic
Sci Int. 161(1) 60-63.
Calafell, F., Perez-Lezaun, A. et Bertranpetit, J., Genetic distances and microsatellite diversification in humans. Hum Genet. 2000. 106(1): 133-134.
Coudray C., 2006, Histoire génétique et évolution des populations berbérophones nord-africaines, Thèse de Doctorat, Centre d’Anthropologie, Université Toulouse III-Paul Sabatier.
Coudray, C., Guitard, E., Keyser-Tracqui, C., Melhaoui, M., Cherkaoui, M., Larrouy, G. et
Dugoujon, J.M., 2007, Population genetic data
of 15 tetrameric short tandem repeats (STRs)
in Berbers from Morocco, Forensic Sci. Int.,
167, 81-86.
Coudray, C, Guitard, E., El-Chennawi, F.,
Larrouy, G., Dugoujon, J.M., 2007 Allele frequencies of 15 short tandem repeats
(STRs) in three Egyptian populations of different ethnic groups, Forensic Sci.
Int., 169, 260-265.
Coudray,
C., Calderon, R., Guitard, E., Ambrosio, B., Gonzalez-Martın, A. et
Dugoujon, JM., 2007, Allele frequencies of 15 tetrameric short tandem repeats
(STRs) in Andalusians from Huelva (Spain), Forensic Sci. Int., 168, 21–24.
Decorte,
R., Engelen, M., Larno, L., Nelissen, K., Gilissen, A. et Cassiman, JJ., 2004,
Belgian population data for 15 STR loci (AmpFlSTR SGM Plus and AmpFlSTR
profiler PCR amplification kit), Forensic Sci Int. 139 (2-3), 211-213.
El Ossmani, H., Bouchrif, B., Talbi,
J., El Amri, H. et Chafik, A., 2007, La diversité génétique de 15 STR chez la
population arabophone de Rabat-Salé-Zemmour-Zaer, Antropo, 15, 55-62. www.didac.ehu.es/antropo.
Felsenstein, J., 2007: Phylogeny Inference
Package (PHYLIP) version 3.67, Departement of Genome Sciences and Departement
of Biology, University of Washington, Seattle, WA, USA.
Gorostiza,
A., Gonzalez-Martın, A., Lopez Ramırez, C., Sanchez, C., Barrot, C.,
Ortega, M., Huguet, E., Corbella, J. et Gené, M., 2007, Allele frequencies of
the 15 AmpF/Str Identifiler loci in the population of Metztitla´n (Estado de
Hidalgo), México Forensic Sci. Int., 166, 230–232.
Havas,
D., Jeran, N., Efremovska, L., Ðorpevic, D. et Rudan, P., 2007, Population
genetics of 15 AmpflSTR Identifiler loci in Macedonians and Macedonian Romani
(Gypsy), Forensic Sci. Int., 173 (2-3), 220-224.
Hima
Bindu, G., Trivedi, R. et Kashyap V.K., 2007, Allele frequency distribution
based on 17 STR markers in three major Dravidian linguistic populations of
Andhra Pradesh, India Forensic Sci. Int., 17, 76–85.
Kim,
YL., Hwang, JY., Kim, YJ., Lee, S., Chung, NG., Goh, HG., Kim, CC. et Kim, DW.,
2003, Allele frequencies of 15 STR loci using AmpF/STR Identifiler kit in a
Korean population. Forensic Sci. Int., 136, 92–95.
Manuel,
V., Camacho, Benito, C. et Figueiras, A.M., 2007, Allelic frequencies of the 15
STR loci included in the AmpFlSTR1 IdentifilerTM PCR Amplification Kitin an
autochthonous sample from Spain, Forensic Sci. Int., 173 (2-3), 241-245.
Rebała,
K., Wysocka, J., Kapinska, E., Cybulska, L., Mikulich, A.I., Tsybovsky, I.S.,
et Szczerkowska, Z., 2007, Belarusian population genetic database for 15
autosomal STR loci, Forensic Sci. Int., 173 (2-3), 235-237.
Regueiro, M,. Carril, J.C., Pontes, M.L.,
Pinheiro, M.F., Luis, J.R., et Caeiro, B., 2004, Allele distribution of 15
PCR-based loci in the Rwanda Tutsi population by multiplex amplification and
capillary electrophoresis. Forensic Sci Int. 143(1), 61-63.
Rerkamnuaychoke,
B., Rinthachai, T., Shotivaranon, J., Jomsawat, U., Siriboonpiputtana, T.,
Chaiatchanarat, K., Pasomsub, E. et Chantratita, W., 2006, Thai population data
on 15 tetrameric STR loci-D8S1179 D21S11 D7S820 CSF1PO D3S1358 TH01 D13S317
D16S539 D2S1338 D19S433 vWA TPOX D18S51 D5S818 and FGA. Forensic Sci. Int.,
158(2-3), 234-237.
Rodrıguez,
A., Arrieta, G., Sanou, I., Vargas, M.C., Garcıa, O., Yurrebaso, I.,
Pérez, J.A., Villalta, M. et Espinoza, M., 2007, Population genetic data for 18
STR loci in Costa Rica, Forensic Sci. Int., 168, 85–88.
Rosenberg,
NA., Pritchard, JK., Weber, JL., Cann, HM., Kidd, KK., Zhivotovsky, LA. et
Feldman, MW., 2002, Genetic
structure of human populations. Science., 298 (5602), 2381-2385.
Shepard,
E.M. et Herrara, R.J., 2006, Iranian STR variation at the fringes of
biogeogrophical demarcation, Forensic Sci. Int., 158, 140-148.
Talbi, J., Khadmaoui, A., Soulaymani, A. et Chafik, A. 2006, Caractérisation du comportement matrimonial de la population marocaine. Antropo, 13, 57-67. www.didac.ehu.es/antropo.
Talbi, J., Khadmaoui, A., Soulaymani, A. et Chafik, A. E. A., 2007, Etude de la consanguinité dans la population marocaine. Impact sur le profil de la santé, Antropo, 15, 1-11. www.didac.ehu.es/antropo.
Verrier E., Rognon X., Laloë D. et Rochambeau H., 2005, Ethnozootechnie 76, 67-82.
Wang,
CW., Chen, DP., Chen, CY., Lu, SC. et Sun CF., 2003, STR data for the AmpFlSTR
SGM Plus and Profiler loci from Taiwan, Forensic Sci. Int., 138, 119–122.
Yang,
B., Wang, G., Liu, Y. et Yang, W., 2005, Population data for the AmpFl STR
Identifiler PCR Amplification Kit in China Han in Jilin Province China.
Forensic Sci Int. 151(2-3), 293-297.
Zuniga, J., Ilzarbe, M., Acunha-Alonzo, V., Rosetti, F., Herbert, Z., Romero, V., Almeciga, I., Clavijo, O., Stern, J.N.H., Granados, J., Fridkis-Hareli, M., Morrison, P., Azocar, J. et Yunis E.J., 2006, Allele frequencies for 15 autosomal STR loci and admixture estimates in Puerto Rican Americans. Forensic Sci Int. 164, 266–270.