
El Ossmani, H., Bouchrif, B., Talbi, J., El Amri, H., Chafik, A., 2007, La diversité génétique de 15 STR chez la population arabophone de Rabat-Salé-Zemmour-Zaer, Antropo, 15, 55-62. www.didac.ehu.es/antropo
La diversité génétique de 15 STR chez la population arabophone de Rabat-Salé-Zemmour-Zaer
Gene diversity of 15 STR among the Arab speaking population of Rabat-Salé-Zemmour -Zaer
H. El Ossmani1,2, B. Bouchrif3, J. Talbi1, H. El Amri2, A. Chafik1
1 Laboratoire d’Anthropogénétique et de Physiopathologie, Département de Biologie, Faculté des Sciences, Université Chouaïb Doukkali, El Jadida. Maroc
2 Laboratoire de Génétique, Gendarmerie Royale, avenue Ibn Sina 10100, Rabat, Maroc
3 Laboratoire de Biologie Moléculaire, Institut Pasteur, 1 place Luis Pasteur, Casablanca, Maroc
Adresse de correspondance: Hicham El Ossmani, Laboratoire de Génétique de la Gendarmerie Royale, Avenu Ibn Sina 10100, Rabat, Maroc. E-mail: helossmani@yahoo.fr
Mots-Clés: Anthropogénétique, arabophone, Maroc, polymorphismes génétiques, STR.
Keywords: Anthropogenetics, Arab speaking, Morocco, gene polymorphisms, STR.
Résumé
L´effort fournit pour déchiffrer le génome humain a permis la révélation de nouveaux polymorphismes génétiques dotés de capacités informatives importantes de point de vue anthropogénétique. Les polymorphismes génétiques de l´ADN ont, ainsi, remplacé les marqueurs phénotypiques classiques (ABO, RH, etc...) dans la reconstruction de l´histoire évolutive des populations humaines. Dans ce travail, 15 marqueurs STR (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179, TH01, VWA, D13S317, D16S539, D18S51 et D21S11) ont été exploités pour caractériser la diversité génétique de la population arabophone de Rabat-Salé-Zemmour-Zaer du Maroc. 387 individus originaires de Rabat-Salé-Zemmour-Zaer ont été typés au niveau des 15 marqueurs STR étudiés. L’analyse de la distribution des fréquences alléliques de ces systèmes a montré que la population est en équilibre génétique sauf au niveau des deux STR FGA et D16S539 qui témoignent d’une déviation significative par rapport à la situation d’équilibre. Le pouvoir de discrimination (PD), le pouvoir d’exclusion (PE) et le contenu informatif du polymorphisme (CIP) ont été estimés à partir des fréquences alléliques pour chaque marqueur. Le degré d’hétérozygotie et du polymorphisme au niveau des 15 STR présente un étendu important.
Abstract
Effort provided to characterize the human genome has permitted the revelation of new gene polymorphisms with great informative capacities from anthrpogenetics point of view. Thus, gene polymorphisms of DNA has substituted phenotypic classical markers (ABO, RH,etc..) in the reconstitution of the evolutionary history of human populations. In this work, 15 STR markers (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179, TH01, VWA, D13S317, D16S539, D18S51 and D21S11) were studied to characterize the gene diversity of the Arab speaking population of Rabat Rabat-Salé-Zemmour-Zaer. Genotypes of 387 native individuals of this population have been generated in the 15 loci. The analysis of these systems allelic frequencies showed that the population of Rabat-Salé-Zemmour-Zaer is in equilibrium except for the two loci FGA and D16S539, which are significantly deviated from the Hardy-Weinberg equilibrium. Power of discrimination (PD), power of exclusion (PE) and polymorphic information content (PIC) were estimated from the allelic frequencies of each marker. Heterozygosity and polymorphism level at the 15 STR presents a great range.
Introduction
Plusieurs études ont été réalisées sur des populations arabophones et berbérophones de différentes régions du Maroc, dans le but de leur caractérisation et situation anthropogénétique au sein du bassin Méditerranéen (Perez-Lezaun, A. et al., 2000; Bosch, E. et al., 2001; Dios, S. et al., 2001; Abdin, L. et al., 2003; Jauffrit, A. et al., 2003 et Coudray et al., 2007). Ce genre d’études est censé, en effet, définir les flux migratoires et les affinités génétiques qu’ont eu ces différentes populations au cours du temps.
Néanmoins, en dépit de la multitude des études qui ont été menées sur les populations marocaines, on assiste à un manque flagrant de données sur plusieurs autres populations qui témoignent d’une importance historique dans la constitution du substratum du Maroc. Parmi les populations dont la caractérisation anthropogénétique n’a jamais été rapportée, on cite la population arabophone du plateau de Rabat-Salé-Zemmour-Zaer. Cette population présente la particularité de séparer les populations berbérophones du Moyen Atlas des populations arabophones de la plaine du Zaer et de la Chaouia. En effet, de par son positionnement géopolitique qui en fait une tour pour contrôler tout le pays et de par sa richesse naturelle avec des potentialités particulièrement agricoles, la région de Rabat-Salé-Zemmour-Zaer a été l’objet d’une installation massive des arabophones lors des invasions islamiques au Maroc.
Afin de combler le manque de données sur cette population, nous avons mené la présente étude dans le but de caractériser la distribution des fréquences alléliques de 15 microsatellites.
Population et Méthodes
1. Situation paléoanthropologique et géolinguistique de la région étudiée
Au cours de toute la période préhistorique, l'Homme a laissé de nombreuses traces, sans doute facilitées par un climat plus favorable qu'aujourd'hui, qui marquent un peuplement très ancien. L’Homme de Taforalt (Oujda) en est un exemple très concret (Kéfi et al., 2005).
À l'acheuléen (Paléolithique inférieur), des traces remontant à au moins 700.000 ans montrent une première activité humaine. Ces hommes de type néanderthalien vivaient principalement de la cueillette et de la chasse. Les outils de cette époque sont les galets aménagés, le biface, les hachereaux... découverts principalement dans les régions de Casablanca et de Rabat-Salé-Zemmour-Zaer.
Des sites néolithiques, montrant l'apparition d'une sédentarisation et la naissance de l'agriculture, ont été découverts près de Skhirat (Nécropole de Rouazi-Skhirat) l’une des communes de la région étudiée.
Par ailleurs, en complément des données anthropologiques et archéologiques, la linguistique permet d’apporter des informations sur l’origine des peuples et sur leurs relations. Au Maroc, la langue est aujourd’hui le caractère le plus original qui permet de distinguer les arabes des berbères. La figure 1 illustre la situation de la région de Rabat-Salé-Zemmour-Zaer au sein de la carte géolinguistique du Maroc.
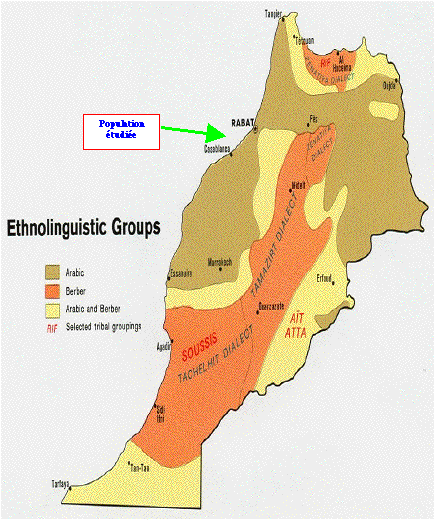
Figure 1. Position géolinguistique de la population Rabat-Sale-Zemmour-Zaer
Figure 1. Geolinguistic position of the population of Rabat-Sale-Zemmour-Zaer
(http://www.souss.com/forum/espace-general/6402-carte-ethnolinguistique-du-maroc-1973-a.html)
2. Echantillonnage
La population arabophone de Rabat-Salé-Zemmour-Zaer a été étudiée à travers un échantillon de prélèvements sanguins de 387 individus arabophones adultes sains, non apparentés et dont les 4 grands-parents sont nés dans la même région. Ces individus ont été recrutés sur la base d’une enquête linguistique réalisée sur le terrain. En effet, un entretien préalable avec les individus nous a permis de confirmer une répartition homogène de leurs origines sur l’ensemble de la région.
3. Choix des marqueurs génétiques: 15 STR
Les critères de sélection des systèmes génétiques doivent prendre en compte l’étendu de leur polymorphisme qui conditionne leur capacité à différencier les populations humaines entre elles, la connaissance validée de leur modalité de transmission génétique, l’action éventuelle de la sélection naturelle sur certains d’entre eux, la possibilité de mettre en œuvre des méthodes d’exploration validées et maîtrisées, et enfin l’existence de données bibliographiques "exploitables" pour de nombreuses populations en vue de comparaison. Il est bien entendu possible d’y associer de nouveaux systèmes génétiques afin d’évaluer leurs possibilités d’utilisation en Anthropologie génétique, c’est le cas des 15 STR du kit Identifiler utilisé dans notre étude (TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179, TH01, VWA, D13S317, D16S539, D18S51 et D21S11).
Ces marqueurs ont été choisis car ils sont particulièrement variables, peu sujets aux mutations, indépendants, assez courts et faciles à amplifier simultanément. Ces marqueurs sont situés sur différents chromosomes autosomaux. Ils sont tous constitués de motifs répétés tétranucléotidiques classés en 3 catégories (Urquhart et al. 1994): simples, composés et complexes. Les répétitions « simples » contiennent des unités de même séquence et longueur (ex: motif [AATG] du locus TPOX). Les répétitions « composées » comprennent au moins 2 unités "simples" adjacentes (ex: [AGAT],[TCTA] du locus D3S1358). Enfin, les répétitions "complexes" se composent d’unités répétitives différentes et de longueur variable avec diverses séquences intercalées entre les blocs (ex: [TTTC]3 TTTTTTCT [CTTT]n CTCC [TTCC]2 pour le locus FGA). La taille des différents allèles STR varie entre 102 et 358 pb.
4. Analyse génétique
Extraction d’ADN
L’ADN a été extrait à partir des échantillons de sang en utilisant la méthode organique du Phénol-chloroforme.
Quantification d’ADN
La quantification d’ADN est réalisée par la technique de la PCR en temps réel par le kit Quantifiler (Applied Biosystems, Foster City, CA).
Amplification et génotypage des STRs
L’ADN est soumis à un marquage par des molécules fluorescentes pour déterminer le profil génétique STR d’un individu: la PCR multiplex. Basée sur le même principe de la PCR classique, cette technique permet l’amplification d’ADN simultanée de plusieurs STRs. Ces colorations sont ensuite détectées lors de la séparation électrophorétique des « produits » de PCR par un Analyseur Génétique ABI Prism 3130xl (Applied Biosystems, Foster City, CA). Ces informations sont ensuite transmises à un ordinateur qui va les collecter et les traiter par un logiciel spécifique GeneMapper ID v3.2 (Applied Biosystems, Foster City, CA). Ce logiciel attribue à chaque allèle STR une taille (en paires de bases) à partir de laquelle le nombre de motifs répétés et le nom de cet allèle sont déduits. Un profil génétique est, ainsi, généré pour chaque individu analysé.
5. Analyse Statistique
Lors de l’analyse des données, nous avons fait appel au logiciel Arlequin version 3.1 (Excoffier et al., 2005) pour estimer les fréquences alléliques, l’hétérozygotie et pour tester l’équilibre de Hardy-Weinberg au niveau de la population. La correction de Bonferroni a été effectuée et seules les valeurs inférieures à 0,003 sont considérées significatives (0,05/15=0,003) (Weir, 1996). Le pouvoir discriminant, le pouvoir d’exclusion et le contenu informatif du polymorphisme ont été estimés par le programme PowerStats version 1.2 (http://www.promega.com/geneticidtools/.).
Résultats
Le tableau 1 présente les fréquences alléliques et les paramètres estimés des 15 STRs étudiés dans la population arabophone de Rabat-Salé-Zemmour-Zaer. La population est génétiquement très diversifiée avec des hétérozygoties allant de 0,672 pour les loci D5S818 et TPOX à 0,879 au niveau du locus D2S1338. La population arabophone de Rabat-Salé-Zemmour-Zaer se présente en équilibre de Hardy-Weinberg pour tous les loci, à l’exception des deux loci D16S539 et FGA dont la divergence constatée par rapport à l’état d’équilibre s’est révélée significative même après l’application de la correction de Bonferroni.
Allele |
D5S818 |
FGA |
D8S1179 |
D21S11 |
D7S820 |
CSF1PO |
D3S1358 |
|
6 |
- |
- |
- |
- |
- |
- |
- |
|
7 |
- |
- |
- |
- |
0,004 |
0,009 |
- |
|
8 |
0,034 |
- |
0,013 |
- |
0,134 |
0,030 |
- |
|
9 |
0,052 |
- |
- |
- |
0,147 |
0,022 |
- |
|
9,3 |
- |
- |
- |
- |
- |
- |
- |
|
10 |
0,060 |
- |
0,121 |
- |
0,315 |
0,263 |
- |
|
11 |
0,263 |
- |
0,129 |
- |
0,220 |
0,384 |
- |
|
12 |
0,419 |
- |
0,086 |
- |
0,155 |
0,237 |
- |
|
13 |
0,168 |
- |
0,194 |
- |
0,026 |
0,047 |
- |
|
13,2 |
- |
- |
- |
- |
- |
- |
- |
|
14 |
- |
- |
0,228 |
- |
- |
0,009 |
0,043 |
|
14,2 |
- |
- |
- |
- |
- |
- |
- |
|
15 |
0,004 |
- |
0,177 |
- |
- |
- |
0,336 |
|
15,2 |
- |
- |
- |
- |
- |
- |
- |
|
16 |
- |
- |
0,052 |
- |
- |
- |
0,233 |
|
16,2 |
- |
- |
- |
- |
- |
- |
- |
|
17 |
- |
- |
- |
- |
- |
- |
0,263 |
|
17,2 |
- |
- |
- |
- |
- |
- |
- |
|
18 |
- |
- |
- |
- |
- |
- |
0,108 |
|
19 |
- |
0,039 |
- |
- |
- |
- |
0,009 |
|
20 |
- |
0,147 |
- |
0,009 |
- |
- |
0,009 |
|
21 |
- |
0,207 |
- |
- |
- |
- |
- |
|
21,2 |
- |
0,004 |
- |
- |
- |
- |
- |
|
22 |
- |
0,220 |
- |
- |
- |
- |
- |
|
23 |
- |
0,121 |
- |
- |
- |
- |
- |
|
24 |
- |
0,121 |
- |
- |
- |
- |
- |
|
25 |
- |
0,095 |
- |
- |
- |
- |
- |
|
26 |
- |
0,035 |
- |
- |
- |
- |
- |
|
27 |
- |
0,013 |
- |
0,026 |
- |
- |
- |
|
28 |
- |
- |
- |
0,116 |
- |
- |
- |
|
29 |
- |
- |
- |
0,241 |
- |
- |
- |
|
30 |
- |
- |
- |
0,190 |
- |
- |
- |
|
30,2 |
- |
- |
- |
0,013 |
- |
- |
- |
|
31 |
- |
- |
- |
0,099 |
- |
- |
- |
|
31,2 |
- |
- |
- |
0,095 |
- |
- |
- |
|
32 |
- |
- |
- |
0,004 |
- |
- |
- |
|
32,2 |
- |
- |
- |
0,142 |
- |
- |
- |
|
33,2 |
- |
- |
- |
0,043 |
- |
- |
- |
|
34,2 |
- |
- |
- |
0,017 |
- |
- |
- |
|
35 |
- |
- |
- |
0,004 |
- |
- |
- |
|
Ho |
0.672 |
0.733 |
0.802 |
0.845 |
0.776 |
0.776 |
0.784 |
|
He |
0.723 |
0.850 |
0.841 |
0.854 |
0.792 |
0.727 |
0.753 |
|
P |
0.254 |
0.001c |
0.031 |
0.030 |
0.013 |
0.398 |
0.371 |
|
PD |
0,884 |
0,948 |
0,947 |
0,949 |
0,916 |
0,861 |
0,883 |
|
PIC |
0,68 |
0,83 |
0,82 |
0,83 |
0,76 |
0,68 |
0,71 |
|
PE |
0,387 |
0,481 |
0,602 |
0,685 |
0,555 |
0,555 |
0,571 |
Tableau 1. les fréquences alléliques des 15 STR de la population Rabat-Salé-Zemmour-Zaer. H0: Hétérozygotie observée; He: Hétérozygotie attendue; P: Test exact de l’équilibre HW; PD: Pouvoir de discrimination; PE: Pouvoir d’exclusion; PIC: Contenu informatif du polymorphisme; c: Correction de Bonferroni (0.05/15=0.0033).
Table 1. Allelic frequencies of the 15 STR of the population of Rabat-Salé-Zemmour-Zaer. H0: Observed heterozyosity; He: Expected heterozygosity; P: Exact test of Hardy-Weinberg equilibrium; PD: Power of discrimination; PE: Power of exclusion; PIC: Polymorphic information content; c: Correction de Bonferroni.
Allele |
THO1 |
D13S317 |
D16S539 |
D2S1338 |
D19S433 |
VWA |
TPOX |
D18S51 |
|
6 |
0,198 |
- |
- |
- |
- |
- |
0,004 |
- |
|
7 |
0,185 |
- |
- |
- |
- |
- |
0,004 |
- |
|
8 |
0,177 |
0,103 |
0,052 |
- |
- |
0,004 |
0,392 |
- |
|
9 |
0,259 |
0,048 |
0,142 |
- |
- |
- |
0,159 |
- |
|
9,3 |
0,142 |
- |
- |
- |
- |
- |
- |
- |
|
10 |
0,030 |
0,035 |
0,073 |
- |
- |
- |
0,082 |
0,004 |
|
11 |
0,009 |
0,280 |
0,237 |
- |
0,009 |
- |
0,319 |
0,030 |
|
12 |
- |
0,392 |
0,276 |
- |
0,134 |
- |
0,034 |
0,147 |
|
13 |
- |
0,082 |
0,185 |
0,004 |
0,190 |
0,013 |
- |
0,078 |
|
13,2 |
- |
- |
- |
- |
0,034 |
- |
- |
- |
|
14 |
- |
0,060 |
0,022 |
- |
0,297 |
0,151 |
- |
0,181 |
|
14,2 |
- |
- |
- |
- |
0,065 |
- |
- |
- |
|
15 |
- |
- |
0,009 |
- |
0,177 |
0,164 |
0,004 |
0,168 |
|
15,2 |
- |
- |
- |
- |
0,039 |
- |
- |
- |
|
16 |
- |
- |
- |
0,039 |
0,022 |
0,190 |
- |
0,151 |
|
16,2 |
- |
- |
- |
- |
0,022 |
- |
- |
- |
|
17 |
- |
- |
- |
0,241 |
0,004 |
0,211 |
- |
0,138 |
|
17,2 |
- |
- |
- |
- |
0,009 |
- |
- |
- |
|
18 |
- |
- |
- |
0,073 |
- |
0,177 |
- |
0,034 |
|
19 |
- |
- |
- |
0,172 |
- |
0,073 |
- |
0,026 |
|
20 |
- |
- |
- |
0,194 |
- |
0,017 |
- |
0,030 |
|
21 |
- |
- |
0,004 |
0,043 |
- |
- |
- |
0,004 |
|
21,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
22 |
- |
- |
- |
0,030 |
- |
- |
- |
- |
|
23 |
- |
- |
- |
0,056 |
- |
- |
- |
0,009 |
|
24 |
- |
- |
- |
0,073 |
- |
- |
- |
- |
|
25 |
- |
- |
- |
0,061 |
- |
- |
- |
- |
|
26 |
- |
- |
- |
0,014 |
- |
- |
- |
- |
|
27 |
- |
- |
- |
- |
- |
- |
- |
- |
|
28 |
- |
- |
- |
- |
- |
- |
- |
- |
|
29 |
- |
- |
- |
- |
- |
- |
- |
- |
|
30 |
- |
- |
- |
- |
- |
- |
- |
- |
|
30,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
31 |
- |
- |
- |
- |
- |
- |
- |
- |
|
31,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
32 |
- |
- |
- |
- |
- |
- |
- |
- |
|
32,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
33,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
34,2 |
- |
- |
- |
- |
- |
- |
- |
- |
|
35 |
- |
- |
- |
- |
- |
- |
- |
- |
|
Ho |
0.759 |
0.750 |
0.828 |
0.879 |
0.810 |
0.828 |
0.672 |
0.784 |
|
He |
0.812 |
0.746 |
0.808 |
0.856 |
0.822 |
0.836 |
0.714 |
0.870 |
|
P |
0.006 |
0.481 |
0.002c |
0.004 |
0.015 |
0.132 |
0.162 |
0.040 |
|
PD |
0,926 |
0,884 |
0,909 |
0,945 |
0,933 |
0,942 |
0,864 |
0,961 |
|
PIC |
0,78 |
0,71 |
0,78 |
0,84 |
0,80 |
0,81 |
0,66 |
0,85 |
|
PE |
0,525 |
0,510 |
0,651 |
0,753 |
0,618 |
0,651 |
0,387 |
0,571 |
Tableau 1. (Suite).
Table 1. (Continued).
Les loci D18S51et D21S11 se présentent les plus polymorphes avec 13 allèles. En effet, ces deux loci présentent les pouvoirs discriminants les plus forts, soient respectivement 0,961 et 0,948. L’allèle qui présente la fréquence la plus élevée (0,419) est rencontré au niveau du locus D5S818 (12 répétitions). Ce locus présente, par ailleurs, le pouvoir d’exclusion le plus faible à côté du locus TPOX (0,387). En effet, les nombres d’allèles au niveau de ces deux loci sont respectivement de 7 et 8.
Discussion
En plus des 13 microsatellites de la base de données américaine CODIS (COmbined DNA Index System): TPOX, D3S1358, FGA, D5S818, CSF1PO, D7S820, D8S1179, TH01, VWA, D13S317, D16S539, D18S51 et D21S11, nous avons ajouté les deux systèmes D2S1338 et D19S433. Ainsi, les fréquences alléliques de 15 STRs autosomaux ont été utilisées pour définir la distribution de leurs fréquences alléliques chez la population arabophone de Rabat-Salé-Zemmour-Zaer.
Les résultats confirment le potentiel discriminant important de ces marqueurs, avec une valeur importante au niveau du système D18S51, ce qui concorde avec les résultats de Shepard et Herrara (2005) lors d'une étude réalisée sur la population Iranienne. Ceci témoigne de la fiabilité de l'usage de ces marqueurs, non pas seulement en matière d’identification des individus, mais surtout pour quantifier les affinités génétiques entre les populations humaines. En effet, la distribution des fréquences alléliques au niveau des différents loci est susceptible d'apporter des informations sur l'état d'équilibre de la population et sur les tendances évolutives de sa structure génétique. Mises dans un contexte démographique, ces informations assurent une approximation très probable de la dynamique socio-comportementale et historique de la population.
13 des 15 loci étudiés témoignent d’une population en équilibre génétique. Cependant, la distribution des fréquences alléliques des loci D16S539 et FGA se présente déviée de l’état d’équilibre. Les sources susceptibles mais très discutables de cette déviation sont, en effet, multiples. On peut en avancer une éventuelle dérive génétique ayant changé la distribution des fréquences alléliques de ces deux marqueurs. Les pratiques matrimoniales dans la région pourraient impliquer une dépression quant à l’équilibre au niveau de ces deux marqueurs. En effet, la proportion des mariages consanguins dans la région de Rabat-Salé-Zemmour-Zaer atteint 20% (Hami et al, 2007). Une des sources du déséquilibre affiché au niveau de ces deux marqueurs pourrait résider au niveau de leur définition moléculaire. En effet, un taux inapproprié de mutations est susceptible de perturber l’état d’équilibre d’un gène donné. Le mode de transmission des allèles au niveau de ces loci pourrait être une éventuelle source de plus à travers un éventuel déséquilibre de liaison.
Conclusion
Hormis le déséquilibre noté au niveau des deux marqueurs (D16S539 et FGA), les 13 autres marqueurs montrent que la population est équilibrée génétiquement. Une recherche dans l’histoire de cette population, associée à une étude moléculaire soigneuse au niveau des loci qui se sont révélés déséquilibrés, permettra d’expliquer ce déséquilibre.
Par ailleurs, l’ensemble des données relevées peuvent être exploitées dans une étude ultérieure pour situer la population de Rabat-Salé-Zemmour-Zaer dans un contexte régional et mondial, notamment par rapport aux populations du moyen orient soupçonnées, historiquement, être l’origine des flux migratoires vers la population étudiée lors de l’installation de l’empire islamique au Maroc. Une comparaison par rapport aux données publiées récemment sur ces populations permettra de ratifier ou rejeter cette hypothèse. Cette comparaison, ainsi qu’une comparaison intrarégionale visant l’estimation du degré de métissage entre cette population arabophone et les berbèrophones du Maroc, feront l’objet de notre prochaine publication.
References
Abdin, L., Shimada, I., Brinkmann, B. et Hohoff, C., 2003, Analysis of 15 short tandem repeats reveals significant differences between the Arabian populations from Morocco and Syria. Legal Medicine (Tokyo), 5(Suppl1), S150-155.
Applied Biosystems, 2001, AmpFl STR® Identifiler™ PCR Amplification Kit User's Manual, Foster City, CA, P/N 4323291.
Bosch, E., Clarimon, J., Perez-Lezaun, A. et Calafell, F., 2001, STR data for 21 loci in north-western Africa, Forensic Science International, 116(1), 41-51.
Coudray, C., Guitard, E., Keyser-Tracqui, C., Melhaoui, M., Cherkaoui, M., Larrouy, G. et Dugoujon, J.M., 2007, Population genetic data of 15 tetrameric short tandem repeats (STRs) in Berbers from Morocco. Forensic Science International, 167, 81-86.
Dios, S., Luis, J.R., Carril, J.C. et Caeiro, B., 2001, Sub-Saharan genetic contribution in Morocco: microsatellite DNA analysis, Human Biology, 73(5), 675-688.
Excoffier L., Laval, G. and Schneider S., 2005, Arlequin ver. 3.1: An integrated software package for population genetics data analysis, Evolutionary Bioinformatics Online, 1, 47-50.
Hami, H., Soulaymani, A. et Mokhtari, A., 2007, Traditions matrimoniales dans la région de Rabat-Salé-Zemmour-Zaer au Maroc, Bulletins et Mémoires de la Société d'Anthropologie de Paris, 19, 1-2.
Jauffrit, A., El Amri, H., Airaud, F., Andre, M.T., Herbert, O., Landeau-Trottier, G., Giraudet, S., Richard, C., Chaventre, A. et Moisan, J.P., 2003, DNA short tandem repeat profiling of Morocco, Journal of Forensic Science, 48(2), 458-459.
Kéfi, R., Stevanovitch, A., Bouzaid, E. et Béraud-Colomb, E.,2005, Diversité mitochondriale de la population de Taforalt (12.000 ans BP – Maroc): une approche génétique à l’étude du peuplement de l’Afrique du Nord, Anthropologie, 43, 1-11.
Pérez-Lezaun, A., Calafell, F., Clarimón, J., Bosch, E., Mateu, E., Gusmao, L., Amorim, A., Benchemsi, N. et Bertranpetit, J., 2000, Allele frequencies of 13 short tandem repeats in population samples from the Iberian Peninsula and Northern Africa. International Journal of Legal Medicine, 113(4), 208-214.
PowerStats ver. 1.2, 1999, http://www.promega.com/geneticidtools/.
Shepard, E.M. et Herrara, R.J., 2006, Iranian STR variation at the fringes of biogeogrophical demarcation, Forensic Science International, 158, 140-148.
Urquhart, A., Kimpton, C.P., Downes, T.J. et Gill, P., 1994, Variation in short tandem repeat sequences: a survey of twelve microsatellite loci for use as forensic identification markers. Internationa Journal of Legal Medicine, 107(1), 13-20.
Weir, B.S., 1996, Multiple tests. Dans: Genetic data analysis II, Sinauer Associates, USA, 134, 109-110.