
Gómez-Pérez, L., Alfonso-Sánchez, M.A.,
Pérez-Miranda, A.M., de Pancorbo, M.M., Peña, J.A., 2007, Utilidad
de las inserciones Alu en los estudios de mestizaje, Antropo, 14,
29-36. www.didac.ehu.es/antropo
Utilidad de las inserciones Alu en los estudios de
mestizaje
The usefulness of Alu insertions in admixture studies
Luis Gómez-Pérez1, Miguel A.
Alfonso-Sánchez1, Ana M.
Pérez-Miranda1, M. M.
de Pancorbo2, Jose A.
Peña1
1 Departamento
de Genética, Antropología Física y Fisiología Animal, Facultad de Ciencia y
Tecnología, Universidad del País Vasco, 48080 Bilbao, Spain.
2
Departamento de Zoología y Dinámica Celular Animal, Facultad de Farmacia,
Universidad del Pais Vasco, 48080 Bilbao, Spain
Dirección para
correspondencia: Luis Gómez-Pérez. Departamento de Genética,
Antropología Física y Fisiología Animal, Facultad de Ciencia y Tecnología,
Universidad del País Vasco, Aptdo. 644, 48080 Bilbao, Spain. E-mail:
luis.gomez@ehu.es
Palabras
clave:
Inserciones Alu, análisis discriminante, mestizaje.
Keywords:
Alu insertions, discriminant
analysis, admixture.
Resumen
Los polimorfismos de inserción Alu son unos marcadores muy interesantes para los
estudios de la evolución humana, porque se producen por un único evento
mutacional y se conoce su estado ancestral.
Sobre una base de datos de frecuencias génicas de 8
inserciones Alu en 47 poblaciones de diferentes continentes, se ha
realizado un análisis discriminante, con el objetivo de evaluar la capacidad de
estos marcadores para clasificar poblaciones y en consecuencia para estimar el
grado de mestizaje de poblaciones híbridas. Para las 6 regiones establecidas,
se ha obtenido un porcentaje de correcta clasificación del 91,5%, permitiendo
distinguir razonablemente bien entre poblaciones del Africa subsahariana,
Africa del Norte y Próximo Oriente, Europa, Medio Oriente, Asia y América. Se
confirma por tanto la utilidad de las inserciones Alu para los
estudios de mestizaje.
Abstract
Alu insertion polymorphisms are robust markers for human evolutionary
studies because they
have a unique mutational mechanism, with a known ancestral state.
On a data base of gene
frequencies for 8 Alu insertions in 47 populations of different continents, a
discriminant analysis has been performed, with the aim of evaluating the
capacity of these markers to classify populations and consequently to estimate
the degree of admixture of hybrid populations. For the 6 established regions, a
percentage of correct classification of 91.5% has been obtained, allowing to
distinguish reasonably well between populations of Sub-Saharan Africa, North
Africa and Middle East, Europe, India and Central Asia, East Asia and America.
The utility of the Alu insertions is confirmed therefore for the admixture
studies.
Introducción
Los elementos móviles pueden constituir
aproximadamente el 45% del genoma del hombre. Entre ellos, el grupo más
abundante lo compone la familia de elementos Alu, que supone
más del 10% de nuestro ADN con más de un millón de copias. Las inserciones Alu son marcadores
bialélicos que se definen por la presencia o ausencia de unas secuencias de
aproximadamente 300 pares de bases, que con variaciones más o menos importantes
derivan del gen 7SL RNA (el cual forma parte del complejo ribosomal) por
retrotransposición. Son especialmente interesantes en Antropogenética por
aparecer hace unos 65 millones de años y encontrarse asociadas con el origen y
expansión de los primates (Batzer y Deininger, 2002).
La mayor parte de las inserciones Alu son
relativamente recientes, por lo que en general son polimórficas, pudiendo tomar
diferentes valores de frecuencias en diferentes poblaciones humanas (Stoneking et
al.,
1997).
Es altamente improbable que la misma
inserción Alu ocurra dos
veces independientemente en el mismo lugar, por lo que se considera que son
eventos mutacionales únicos (Batzer et al., 1994). Sin embargo, se han observado excepciones, como es el caso
de la inserción PV92, en la que al menos algunos individuos del País Vasco y
del Norte de Africa muestran una inserción dentro de una inserción (Comas et
al., 2001; García-Obregón et al, 2007).
Por otra parte, salvo algunas excepciones, la
selección no debiera tener una influencia considerable sobre las secuencias Alu, ya que no son codificantes. No obstante, en
algunos casos pueden encontrarse fuertemente ligadas a un gen y verse afectadas
por las presiones selectivas que afecten al mismo o pueden incluso determinar
alteraciones en un gen próximo facilitando la aparición de enfermedades, como
la neurofibromatosis tipo 1, el síndrome de Apert y varios tipos de cáncer
(Wallace et al. 1991;
Miki et al. 1996;
Deininger y Batzer 1999; Oldridge et al. 1999).
En general, puede afirmarse que una de las
principales características de las inserciones Alu es su estabilidad, debido a la escasa
influencia de la mutación y de la selección sobre estos polimorfismos
(García-Obregón et al,
2007). Por ello, cabe esperar que a partir de un número limitado de inserciones
Alu pueda obtenerse una
buena discriminación entre poblaciones de diferentes continentes, lo que
sustentaría el argumento de que estos marcadores son interesantes para los
estudios de mestizaje.
En este trabajo se han recopilado las
frecuencias de 8 inserciones Alu (A25, ACE, APO, B65, D1, FXIIIB, PV92 y TPA25) en muestras
representativas de diferentes continentes, con el objetivo de contrastar la
capacidad de discriminación de estos marcadores y en consecuencia su interés
para evaluar el grado de mestizaje de una población metamórfica.
Materiales y métodos
A partir de trabajos publicados previamente
se han recopilado las frecuencias alélicas de 8 inserciones Alu (A25, ACE, APO, B65, D1, FXIIIB, PV92 y
TPA25) en 47 popblaciones de 4 continentes, que se distribuyeron en 6 regiones
definidas con una cierta coherencia desde un punto de vista antropogenético: 1) Africa subsahariana, 2) Norte de
Africa y Próximo Oriente, 3) Europa, 4) Medio Oriente, 5) Asia y 6) América
(Tabla 1). La ausencia de poblaciones de Oceanía se explica por la inexistencia
de muestras analizadas para las 8 inserciones en un número representativo. Por
otra parte, América se encuentra representada por tan sólo 3 poblaciones, pero
se ha considerado conveniente incluirlas, por ser el continente donde mayor
número de poblaciones mestizas existen. La localización geográfica, las referencias de origen de los datos y el
código en las figuras de las 47 muestras poblacionales se muestran en la
tabla 1 y la figura 1.
Considerando las 6 regiones geográficas mencionadas, se analizó la heterogeneidad genética intra e interpoblacional mediante un análisis molecular de la varianza (AMOVA) con el programa Arlequin versión 3.0 (Excoffier et al., 2005). Las distancias genéticas entre poblaciones se establecieron a partir del coeficiente R de Harpending y Jenkins (1973). Sobre la matriz de distancias se realizó un análisis de escalamiento multidimensional (MDS) (Kruskal 1964), con el paquete estadístico SPSS versión 13.0 (SPSS, Chicago, Ill.). Se obtuvieron 6 dimensiones, para cuyos vectores propios se contrastó la hipótesis de normalidad de su distribución, mediante el test de Kolmogorov-Smirnov. Finalmente, se realizó sobre dichos vectores un análisis discriminante, también con el programa SPSS.
|
Muestra |
Región |
Referencia |
Código |
|
!Kung |
Africa subsahariana |
Stoneking et al, 1997 |
1 |
|
Kenia (Bantús) |
Africa
subsahariana |
Terreros et
al, 2005 |
2 |
|
Nguni |
Africa
subsahariana |
Stoneking et
al, 1997 |
3 |
|
Nigeria |
Africa
subsahariana |
Stoneking et al, 1997 |
4 |
|
Pigmeos Biaka |
Africa
subsahariana |
Bowcock et al, 1987 |
5 |
|
Pigmeos Mbuti |
Africa
subsahariana |
Bowcock et al, 1987 |
6 |
|
Sotho-Tswana |
Africa
subsahariana |
Stoneking et al, 1997 |
7 |
|
Argelia |
N. Africa y
Próximo Oriente |
Comas et al, 2000 |
8 |
|
Marruecos
(Norte) |
N.
Africa y Próximo Oriente |
Comas et al, 2000 |
9 |
|
Marruecos
(Oeste) |
N.
Africa y Próximo Oriente |
Comas et al, 2000 |
10 |
|
Marruecos
(Sudeste) |
N.
Africa y Próximo Oriente |
Comas et al, 2000 |
11 |
|
Sáhara
Occidental |
N.
Africa y Próximo Oriente |
Comas et al, 2000 |
12 |
|
Túnez |
N.
Africa y Próximo Oriente |
Comas et al, 2000 |
13 |
|
Armenia |
N.
Africa y Próximo Oriente |
Nasidze et al, 2001 |
14 |
|
Azerbayán |
N.
Africa y Próximo Oriente |
Nasidze et
al, 2001 |
15 |
|
Emiratos
Arabes |
N.
Africa y Próximo Oriente |
Stoneking et al, 1997 |
16 |
|
Siria |
N.
Africa y Próximo Oriente |
Nasidze et
al, 2002 |
17 |
|
Turquía |
N.
Africa y Próximo Oriente |
Romualdi et
al, 2004 |
18 |
|
Albania |
Europa |
Comas et al, 2004 |
19 |
|
España (AndAlucía) |
Europa |
Comas et al, 2000 |
20 |
|
España (CatAluña) |
Europa |
Comas et al, 2000 |
21 |
|
España (País
Vasco) |
Europa |
García-Obregón et al, 2007 |
22 |
|
España
(Valencia) |
Europa |
García-Obregón et
al, 2006 |
23 |
|
Francia |
Europa |
Stneking et
al, 1997 |
24 |
|
Grecia |
Europa |
Comas et al, 2004 |
25 |
|
Macedonia |
Europa |
Comas et al, 2004 |
26 |
|
Moldavia |
Europa |
Varzari et
al, 2007 |
27 |
|
Rumanía |
Europa |
Comas et al, 2004 |
28 |
|
Suiza |
Europa |
Stoneking et
al, 1997 |
29 |
|
La
India (Cristianos) |
Medio Oriente |
Stoneking et
al, 1997 |
30 |
|
La
India (Hindús) |
Medio Oriente |
Stoneking et
al, 1997 |
31 |
|
La
India (Musulmanes) |
Medio Oriente |
Stoneking et
al, 1997 |
32 |
|
Paquistán |
Medio Oriente |
Stoneking et
al, 1997 |
33 |
|
Pastunes |
Medio Oriente |
Stoneking et
al, 1997 |
34 |
|
Tamiles |
Medio Oriente |
Stoneking et al, 1997 |
35 |
|
China
(Han) |
Asia |
Xiao et al, 2002 |
36 |
|
China (Sibo) |
Asia |
Xiao et
al, 2002 |
37 |
|
China (Uygures) |
Asia |
Xiao et al, 2002 |
38 |
|
Filipinas |
Asia |
Stoneking et al, 1997 |
39 |
|
Indonesia
(Nusa Tenggara) |
Asia |
Stoneking
et al, 1997 |
40 |
|
Java |
Asia |
Stoneking et al, 1997 |
41 |
|
Malasia |
Asia |
Stoneking et al, 1997 |
42 |
|
Molucas |
Asia |
Stoneking et al, 1997 |
43 |
|
Taiwan |
Asia |
Stoneking et al, 1997 |
44 |
|
Mayas |
América |
Stoneking et
al, 1997 |
45 |
|
Muskokes |
América |
Stoneking et al, 1997 |
46 |
|
Yanomamos |
América |
Romualdi et
al, 2004 |
47 |
Tabla 1. Procedencia de las
muestras consideradas. El código numérico se corresponde con la Figura 1.
Table 1. Location of the samples. The
numerical code is the same in the Figure 1.
Figura 1. Origen de las muestras. El
código numérico se corresponde con
la Tabla 1.
Figure 1. Location of the samples. The
numerical code is the same in the Table 1.
Resultados
En la tabla 2 se muestran los valores medios
y las varianzas estandarizadas de las frecuencias de las 8 inserciones,
calculados para el conjunto de poblaciones consideradas.
El valor más alto de frecuencia alélica media corresponde a APO y el
más bajo a A25; el resto de
inserciones muestran unos valores no muy dispares, dentro de un rango
medio de frecuencias. Los valores más altos de FST, por el
contrario, corresponden a FXIIIB y PV92 y los más bajos a TPA25, D1 y B65. Esta
disparidad entre ambos estadísticos
se pone de manifiesto al calcular el cociente entre la media y la varianza
estandarizada, en el que se observan dos grupos de inserciones claramente
diferenciados. Por un lado, A25, PV92 y FXIIIB, con unos valores del índice
próximos a 1 (1,1, 1,6 y 1,7 respectivamente). Por otro, el resto de inserciones con valores claramente más altos (ACE: 4,4; D1:
4,6; B65: 6,7; APO: 7,8 y TPA25: 8,1).
|
Inserción |
Posición |
Media |
FST
|
|
A25 |
8q21.3 |
0,146 |
0,132 |
|
ACE |
17q23 |
0,453 |
0,104 |
|
APO |
11q23-q24 |
0,870 |
0,112 |
|
B65 |
11q14.2 |
0,520 |
0,078 |
|
D1 |
3q26.32 |
0,297 |
0,064 |
|
FXIIIB |
1q31-q32.1 |
0,473 |
0,284 |
|
PV92 |
16q24.2 |
0,396 |
0,244 |
|
TPA25 |
8p11.2 |
0,488 |
0,060 |
Tabla 2. Posición en el genoma,
valor medio y varianza estandarizada de las frecuencias de las 8 inserciones Alu para las 47 poblaciones
consideradas.
Table 2. Chromosome location, average
frequencies and standardized variance of the 8 Alu insertions for the 47 populations.
Los resultados del análisis molecular de la
varianza se muestran en la Tabla 3. Las 3 componentes del AMOVA resultaron
significativas para las 8 inserciones. En relación a las diferencias entre las
6 regiones en que se agruparon las 47 poblaciones, el valor medio del
porcentaje de la varianza entre grupos fue del 10,08%, con un valor mínimo del
1,33% para D1 y un valor máximo del 22,53% para PV92. Obviamente, los valores
del porcentaje de la varianza entre grupos y de FST se encuentran
claramente relacionados. Por otra parte, si se considera la varianza dentro de
poblaciones, aquellas que presentan unos porcentajes más bajos son precisamente
las que mostraron una relación más baja entre los valores medios y la varianza
estandarizada de las frecuencias alélicas, aunque dicho resultado entra
asimismo dentro de lo esperado.
Puesto que las frecuencias génicas no son
variables adecuadas para realizar un análisis discriminante, se obtuvieron previamente
unos vectores propios sobre los que aplicarlo. Con este fin, se calculó un
coeficiente de distancia (distancia R de Harpending y Jenkins, 1973) entre cada
par de poblaciones. Se añadió una población hipotética ancestral, en la que las
frecuencias de todas las inserciones eran 0. Sobre la matriz de distancias se
hizo un análisis de escalamiento multidimensional (MDS), obteniéndose 6
dimensiones, puesto que eran 6 los grupos poblacionales que se pretendía
discriminar. Consecuentemente el estrés fue muy bajo, de tan sólo un 2,1% para
una varianza explicada del 99,8%. Se realizó un test de Kolmogorov-Smirnov para
comprobar la normalidad de los 6 vectores propios obtenidos, encontrandose una
probabilidad no significativa en todos los casos (con unos valores del
coeficiente Z de 0,923, 0,767, 0,595, 0,602, 0,668 y 0,886 respectivamente).
|
|
Dentro de poblaciones |
Entre
poblaciones dentro de
grupos |
Entre grupos |
|||
|
Inserción |
Varianza (%) |
|
Varianza (%) |
|
Varianza (%) |
|
|
A25 |
85,74 |
0,14** |
6,37 |
0,07** |
7,89 |
0,08** |
|
ACE |
88,36 |
0,12** |
2,89 |
0,03** |
8,75 |
0,09** |
|
APO |
87,71 |
0,12** |
4,71 |
0,05** |
7,58 |
0,08** |
|
B65 |
91,78 |
0,08** |
5,04 |
0,05** |
3,18 |
0,03** |
|
D1 |
93,53 |
0,06** |
5,14 |
0,05** |
1,33 |
0,01* |
|
FXIIIB |
68,72 |
0,31** |
6,21 |
0,08** |
25,07 |
0,25** |
|
PV92 |
72,93 |
0,27** |
4,54 |
0,06** |
22,53 |
0,23** |
|
TPA25 |
93,41 |
0,07** |
2,27 |
0,02** |
4,32 |
0,04** |
Tabla 3. Análisis molecular de la varianza (AMOVA). ** p<0,001; * p<0,05
Table 3. Analysis of molecular variance (AMOVA). ** p<0,001; * p<0,05
Sobre los vectores propios se realizó un
análisis discriminante. Siendo 6 los vectores, se obtuvieron 5 funciones, de
las cuales las 2 primeras explicaron el 94,7% de la varianza.
En la Figura 2 se representan los
coeficientes de las dos primeras funciones discriminantes en las 47 poblaciones
analizadas y la población ancestral hipotética, que se utilizó como testigo. De
una forma perfectamente coherente, las más cercanas a la población ancestral
son las africanas. Entre éstas y las europeas se encuentran las muestras
representativas del Norte de Africa y Próximo Oriente. Asimismo, en una zona
intermedia entre las poblaciones africanas y asiáticas aparecen dispuestas las
muestras del Medio Oriente. Por último, las poblaciones americanas se localizan
en la proximidad de las asiáticas.
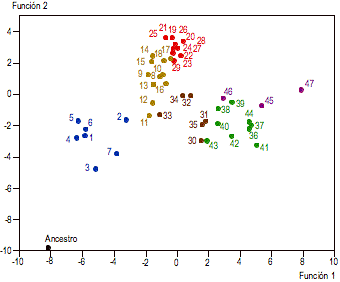
Figura 2. Representación de las 47
poblaciones comparadas, más la población ancestral hipotética, respecto a las 2
primeras funciones discriminantes.
Figure 2. Chart showing the 47
populations plus the hypothetical ancestral population, with respect to the 2
first discriminant functions.
El porcentaje de clasificación correcta
considerando las 5 funciones discriminantes fue del 91,5%, quedando
incorrectamente asignada una población asiática (Nusa Tenggara, de Indonesia),
que se incluyó dentro del grupo de
Medio Oriente y tres poblaciones del Próximo Oriente (Armenia, Siria y Turquía), que se incluyeron dentro
del grupo europeo.
Discusión
Sobre una base de datos de 47 poblaciones de
todo el mundo analizadas, para 8 inserciones Alu, se ha comparado la variabilidad genética
observada entre inserciones y entre regiones continentales.
Teniendo en cuenta que, como se ha dicho, las
inserciones Alu pueden
ser consideradas como eventos mutacionales únicos y que no han podido ocurrir procesos
de flujo génico y deriva diferentes entre las 8 inserciones, pues las
poblaciones utilizadas han sido las mismas, es verosímil la hipótesis de que
las diferencias observadas en la relación entre el valor medio y la varianza
estandarizada de las frecuencias génicas puedan deberse a los efectos de
presiones selectivas. Así, un valor medio más alto debiera implicar en general
una mayor antigüedad de la inserción, por lo que debiera ir acompañado de un
elevado valor de FST. El mayor valor de dicho índice se observa en TPA25, un marcador que se
encuentra en las proximidades del gen que codifica para el activador tisular
del plasminógeno (t-PA, del inglés tissue-type
Plasminogen Activator),
una proteasa que se produce especialmente en en el cerebro y tiene que ver con
la degeneración neuronal. También presenta un valor elevado APO, que se
encuentra a 4,3 kb del gen que codifica para la Apolipoproteina A-I, implicada en el metabolismo del
colesterol. En general, pueden
encontrarse genes más o menos próximos a todas las inserciones Alu estudiadas y sin embargo, salvo casos muy
concretos, no se han publicado evidencias de efectos de la selección sobre las frecuencias de inserciones
Alu. Lamentablemente,
desde un punto de vista metodológico el test de Ewens y Watterson no es
informativo, por tratarse de marcadores bialélicos.
Por otra parte, la clasificación realizada
por el análisis discriminante ha sido realmente efectiva, no sólo por obtenerse
un porcentaje de asignación errónea muy bajo, sino porque la representación
respecto de las 2 primeras funciones ha resultado ser una excelente descripción
del proceso de dispersión del hombre moderno por los diferentes continentes.
Todo ello obtenido tan sólo con 8 marcadores bialélicos.
En cuanto a las poblaciones incorrectamente
clasificadas, la población de Nusa Tenggara, situada en el Extremo Sudeste de
Indonesia aparece incluída dentro del grupo de Medio Oriente, junto a
poblaciones de La India, Paquistán y Afganistán. Es una posición difícil de
explicar, dado que es precisamente la población asiática más alejada
geográficamente del Medio Oriente. Sin embargo, en el trabajo original
realizado sobre esta población (Stoneking et al, 1997),
los resultados obtenidos en forma de dendrograma, son similares a los descritos aquí. Cabe pensar, por tanto, en un
efecto de la deriva genética por sus características históricas y geográficas.
Un posible efecto fundador durante el proceso de colonización de la zona, junto
con la dispersión y el consiguiente aislamiento de las subpoblaciones, todo
ello condicionado por un entorno salpicado de islas, podría haber determinado
cambios al azar en el patrimonio genético de esta población.
La posición de Armenia, Siria y Turquía,
clasificadas como poblaciones europeas, tiene su razón de ser si se tiene en
cuenta que, como se observa en la Figura 2 (puntos 14, 17 y 18
respectivamente), su posición no se encuentra en el interior del grupo europeo,
sino en sus proximidades, reflejando más bien una variación gradual en las
frecuencias alélicas sin solución de continuidad entre Próximo Oriente y
Europa.
Sea como fuere, se ha constatado la gran
eficacia de las 8 inserciones Alu consideradas para la clasificación de poblaciones de diferentes
continentes y por tanto para la estima de tasas de mestizaje en poblaciones
mestizas.
Agradecimientos.
Este trabajo ha sido financiado por el programa de subvenciones a Grupos de
Investigación de la Universidad del País Vasco (GIU 05/51). El trabajo de Luis
Gómez-Pérez ha sido financiado por una beca predoctoral de la Universidad del País Vasco.
Bibliografía
Batzer, M.A., y Deininger, P.L., 2002, Alu repeats and human genomic diversity. Nature Reviews Genetics, 3,370-379
Batzer, M.A., Stoneking, M., Alegria-Hartman, M., Bazan, H., Kass,
D., Shaikh, T., Novick, G., Ioannou, P., Scheer, D., Herrera, R., Deininger,
P., 1994, African origin of human-specific polymorphic Alu insertions. Proc. Natl. Acad. Sci. USA, 91,12288–12292.
Bowcock, A.M., Bucci, C., Hebert, J.M., Kidd, J.R., Kidd, K.K.,
Friedlaender, J.S., Cavalli-Sforza, L.L., 1987, Study of 47 DNA markers in five
populations from four continents. Gene Geogr., 1, 47-64.
Comas, D., Calafell, F., Benchemsi, N., Helal, A., Lefranc, G.,
Stoneking, M., Batzer, M.A., Bertranpetit, J., Sajantila, A., 2000, Alu insertion polymorphisms in NW Africa and the Iberian Peninsula:
evidence for a strong genetic boundary through the Gibraltar straits. Hum,
Genet., 107, 312-319
Comas, D., Plaza, S., Calafell, F., Sajantila, A., Bertranpetit, J.,
2001, Recent insertion of an Alu element within
a polymorphic human-specific Alu insertion. Mol. Biol. Evol., 18,85–88
Comas, D., Schmid, H., Braeuer, S., Flaiz, C., Busquets, A.,
Calafell, F., Bertranpetit, J., Scheil, H.G., Huckenbeck, W., Efremovska, L.,
Schmidt, H., 2004, Alu insertion polymorphisms
in the Balkans and the origins of the Aromuns. Ann. Hum. Genet., 68,120–127.
Deininger, P.L. and Batzer, M.A., 1999, Alu repeats and human disease. Mol. Genet. Metab., 67,183–193
Excoffier, L., Laval, G., Schneider, S.,
2005, ARLEQUIN ver. 3.0: an integrated software package for population genetics
data analysis, Evol Bioinf Online, 1,47–50
García-Obregón, S., Alfonso-Sánchez, M.A., Pérez-Miranda, A.M., de
Pancorbo, M.M., Peña, J.A., 2007, Polymorphic Alu insertions and the genetic structure of Iberian Basques. J. Hum.
Genet., 52,317-327
García-Obregón, S., Alfonso-Sánchez, M.A., Pérez-Miranda, A.M., Vidales, C., Arroyo, D., Peña, J.A., 2006, Genetic position of Valencia (Spain) in the Mediterranean basin according to Alu insertions. Am. J. Hum. Biol., 18,187–195
Harpending,
H., Jenkins, T., 1973, Genetic distance among southern African populations. En
Methods and Theories of Anthropogical Genetics, editado por M. Crawford y P.
Workman (Alburquerque: University
of México Press) pp. 177–99.
Kruskal, J.B., 1964, Multidimensional scaling by optimizing goodness of fit to a nonmetric hypothesis, Psychometrika, 29,1–27
Maca-Meyer, N., Villar, J., Pérez-Méndez, L., de León, A. Cabrera, Flores, C., 2004, A Tale of Aborigines, Conquerors and Slaves:
Alu
Insertion Polymorphisms and the Peopling of Canary Islands. Annals of Human Genetics, 68, 600-605
Miki, Y., Katagiri, T., Kasumi, F., Yoshimoto, T., and Nakamura, Y.,
1996, Mutation analysis in the BRCA2 gene in primary breast cancers. Nat. Genet.,
13,245–247
Nasidze, I., Risch, G.M., Robichaux, M., Sherry, S.T., Batzer, M.A.,
Stoneking, M., 2001, Alu insertion polymorphisms
and the genetic structure of human populations from the Caucasus, Eur. J. Hum.
Genet., 9,267-72
Oldridge, M., Zackai, E.H., McDonald-McGinn, D.M., Iseki, S.,
Morriss-Kay, G.M., Twigg, S.R., Johnson, D., Wall, S.A., Jiang, W., Theda, C., et
al., 1999, De novo Alu-element insertions in FGFR2 identify a distinct pathological basis
for Apert yndrome. Am. J. Hum. Genet., 64,446–461
Romualdi, C., Balding, D., Nasidze, I.S., Risch, G., Robichaux, M.,
Sherry, S.T., Stoneking, M., Batzer, M.A., Barbujani, G., 2002, Patterns of
human diversity, within and among continents, inferred from biallelic DNA
polymorphisms, Genome Research, 12,602-612
Stoneking,
M., Fontius, J., Clifford, S., Soodyall, H., Arcot, S., Saha, N., Jenkins, T.,
Tahir, M., Deininger, P., Batzer, M.A., 1997, Alu insertion polymorphisms and human
evolution: evidence for a larger population size in Africa. Genome Res., 7,1061–1071
Terreros,
M.C., Martinez, L., Herrera, R.J., 2005, Polymorphic Alu insertions and genetic
diversity among African populations, 77, 675-704.
Varzari,
A., Stephan, W., Stepanov, V., Raicu, F., Cojocaru, R., Roschin, Y., Glavce,
C., Dergachev, V., Spiridonova , M., Schmidt, H.D., Weiss, E., 2007, Population
history of the Dniester–Carpathians: evidence from Alu markers. J. Hum.
Genet., 52, 308-316
Xiao,
F.X., Yang, J.F., Cassiman, J.J., Decorte, R., 2002, Diversity at eight
polymorphic Alu insertion loci in Chinese populations shows evidence for European
admixture in an ethnic minority population from northwest China, Hum.
Biol., 74,555-68
Wallace, M.R., Andersen, L.B., Saulino, A.M., Gregory, P.E., Glover, T.W., y Collins, F.S., 1991, A de novo Alu insertion results in neurofibromatosis type 1. Nature, 353, 864–866