Colloques du Groupement des
Anthropologistes de Langue Française (GALF)
Giovannoni, L., Falchi, A., Piras, I.S., Amoros, J.P, Moral, P., Ghiani M.E., Calo’, C.M., Paoli, G., Varesi, L., Vona, G., 2006, Structure génétique de la population Corse. Antropo, 11, 37-50. www.didac.ehu.es/antropo
Structure génétique de la population Corse
Genetic structure of Corsican population
Giovannoni L1., Falchi A1., Piras I.S2.,
Amoros J.P1, Moral P3., Ghiani M.E2., Calo’
C.M2., Paoli G4., Varesi L1., Vona G2
1Université de Corse, laboratoire de Génétique Humaine, Faculté des Sciences 20250 Corte (F)
2Département de Biologie Expérimentale, section Anthropologie, Citadella Universitaria 09042 Monserrato (I)
3Département de Biologie Animale-Anthropologie, Faculté de Biologie Av. Diagonal 645 08028 Barcelone (E)
4Université de Pise, Département Ecologie et Evolution, Unité d’Anthropologie, Via S..Maria 55, 56126 PISA – (I)
Auteur chargé de la correspondance: Varesi Laurent, Université de Corse, laboratoire de génétique humaine,
Faculté des Sciences, 20250 Corte (F). Tél: 0495450027. adresse électronique:
varesi@univ-corse.fr.
Mots clés: Corse, génétique, ADNmt, isoenzymes, ß
Thalassémie
Key-words: Corsica, genetic, mtDNA, isoenzymes, ß Thalassaemias
Résumé
Les fréquences de 18 marqueurs érythrocytaires et plasmatiques ont été analysées dans des populations originaires de quatre micro régions de l’île de Corse: le Nord-est, le Nord-ouest, le centre et l’extrême sud.
L’analyse effectuée dans le contexte méditerranéen,
à partir de l’ensemble de l’échantillonnage insulaire, montre, en premier lieu
une faible influence des populations continentales sur la structure génétique
de la population Corse et en second lieu, une similitude génétique entre les
populations corse et sarde.
Parallèlement, l’analyse du polymorphisme de la
zone hypervariable I de l’ADN mitochondrial, montre les mêmes similitudes,
renforçant ainsi l’hypothèse de la communauté de peuplement des deux îles.
Cette similitude se reflète également dans
le déterminisme génétique d’une pathologie très fréquente en Méditerranée: la ß
Thalassémie (3,1% en Corse). Les populations des deux îles se caractérisent par
une forte fréquence de la mutation ß°-39. L’étude de la répartition des
mutations ß°-39 et ß+IVS1-110 présentes en Corse montre la prédominance du
codon non-sens (88,40%) et la rareté de la mutation ß+IVS1-110 (2,33%) pourtant
majoritaire dans l’est méditerranéen. L’analyse des haplotypes associés à cette
mutation révèle une association préférentielle avec l’haplotype II, en Corse et
en Sardaigne, contrairement aux populations continentales. Ceci suggère
l’origine commune et la sélection par effet fondateur de cette mutation, dans
le bloc corso-sarde.
Abstract
The frequencies of 18 erythrocytary and
plasmatic marker were studied in sample of individuals born and residing in
four micro areas of Corsica island: Eastern-North, Western-North, Centre and
extreme South. Compared to the other Mediterranean populations, Corsica appears
to be greatly differentiated from the populations of regions such as France and
Italy, which have had great political and cultural influence. The Mediterranean
population most comparable to Corsica is Sardinia. Despite their common origin,
they do not prove to be absolutely identical. The genetic characteristics of
Corsica and their relationship with the Mediterranean populations are
interpreted in terms of demographic structure, isolation, and genetic drift.
The mtDNA sequence variation of the
hypervariable segment I of the control region was studied in unrelated
individuals of Corte (Corsica, France). The study confirms results obtained
with classic genetic markers.
In the Corsican population, the incidence of ß Thalassemia traits is reported to be 3.1%. We
have investigated the 2 more important ß Thalassemia mutations present in the Corsican population: ߺ-39 and ß+IVS1-110. Seven polymorphic sites in the b-globin gene cluster were analyzed. Among the Corsican
patients analyzed, the nonsense codon is predominant (88.40%), whereas the ß+IVS1-110 mutation, the most common in the
eastern part of the Mediterranean basin, is underrepresented (2.33%). The other
individuals were not positive for the two tested mutations (9.27%). The ߺ-39 mutation in the studied population shows a
strong association with haplotype II. This strong association was also found in
Sardinia, suggesting that the mutation on the two islands have the same origin.
These data support the hypothesis of a western Mediterranean origin of the ߺ-39 mutation.
Introduction
Par sa superficie la Corse est la troisième île de la
Méditerranée occidentale, après la Sicile et la Sardaigne. Face au golfe de
Gênes, elle se situe à environ 90 km de la Toscane et à 12 km de la Sardaigne.
La Corse possède une altitude moyenne de 568m, avec un point culminant, le
Monte Cinto, à 2710m au-dessus du niveau de la mer.
Les données archéologiques estiment les premières traces
humaines à 6570 avant JC (Camps 1988). Cependant, des traces du Paléolithique
en Sardaigne suggèrent l’existence de communautés ayant peuplé le bloc
Corso-Sarde à différentes périodes, du Pléistocène moyen au Pléistocène récent.
Le passage le plus probable, d’un point de vue géographique, pourrait être
l’archipel toscan (Melillo 1971).
A l’age du bronze, la culture "Torreana",
caractérisée par de nombreux monuments Mégalithiques: dolmens, menhirs,
"Castelli" a laissé de nombreuses traces dans l’ensemble de l’île. Ce
type de culture montre de grandes similitudes avec la culture des
"Nuraghe" de Sardaigne.
L’Histoire commence officiellement en 565 avant JC avec la
fondation de Alalia, l’actuelle Aléria, par une colonie grecque. L’occupation
romaine débuta en 269 avant JC. Après les invasions Vandales et la domination
Byzantine, la Corse fut sous le contrôle du Vatican pendant un certain temps,
avant les dominations Pisane (1077-1284) et du roi d’Aragon (1284-1346). Gênes
conquit ensuite l’île et la contrôla jusqu’au milieu du 18ème siècle
où la Corse connut une brève période d’indépendance avant d’être annexée à la
France par le traité de Versailles de 1768.
La Corse a donc été habitée par différentes populations
comme des Ligures, Phéniciens, Grecs, Romains, Byzantins, Pisans, Génois et
Français.
Elle appartient ainsi au système linguistique italien et
est sous-divisée en quatre zones selon l’affinité au dialecte Toscan. Ce
système montre un certain degré de conservation du Latin et du langage pré-romain
(Rolhfs 1972).
Durant les deux derniers siècles, la Corse a subi une
profonde évolution démographique. De 1800 à 1884, on observa une croissance
démographique constante avec un point culminant en 1884: 276000 habitants
comparés à 125000 en 1729 et 162000 en 1801. Après 1884, une sévère crise
démographique débuta pour atteindre 170000 habitants au début du 19éme siècle.
Une nouvelle croissance démographique commença après 1955 pour atteindre 250371
habitants en 1990.
L’évolution démographique a aussi affecté le mode
d’occupation du territoire. Dans beaucoup de centres urbains, et en particulier
pour Ajaccio et Bastia, la population a augmenté de façon significative, alors
que beaucoup de villages de l’intérieur se sont dépeuplés: en 1990, quatre
communautés rurales sur dix comptaient moins de 100 habitants.
D’un point de vue environnemental la Corse, comme la
Sardaigne, subit durant des millénaires les influences néfastes de la Malaria.
Le parasite ne fut éradiqué qu’à la fin de la seconde guerre mondiale.
La population d’aujourd’hui est d’environ 250000 habitants
avec un passé de forte endogamie, ce qui offre de nombreux avantages pour une
étude génétique. Ainsi, la présence d’allèles, avec des fréquences plus élevées
que dans des populations ouvertes, rend plus facile l’étude du déterminisme
génétique de pathologies ou d’un effet environnemental. Elle est aussi de ce
fait propice à des études d’anthropologie moléculaire comme la recherche de
similitudes inter ou intra régionales, en mettant en lumière des flux géniques
corrélés à des flux migratoires.
Afin d’essayer de comprendre l’histoire du peuplement de
cette île, différents marqueurs génétiques ont été étudiés sur la
population actuelle:
- des marqueurs neutres pour tenter de mettre en lumière
des flux migratoires: autosomiques (protéines érythrocytaires et plasmatiques)
et à transmission maternelle (ADN mitochondrial).
- des marqueurs sélectifs, liés à la pression de la
Malaria, représentés par les mutations responsables des ß-Thalassémies, ainsi
que les haplotypes associés à ces mutations.
Matériel
et méthodes
Marqueurs autosomiques
990 échantillons sanguins originaires de quatre
micro régions ont été examinés: Nord-est, Nord-ouest, Centre et Sud-ouest. Les
individus des deux sexes étaient tous non apparentés, sains, originaires de la
micro région étudiée depuis au moins trois générations (lignées paternelle et
maternelle).
Les génotypes de 18 marqueurs génétiques ont été déterminés
par des techniques d’enzymoélectrophorèse pour les enzymes érythrocytaires
(ACP, ADA, AK, DIA, ESD, GLO, PGD, PGM) , d’immunoélectrophorèse pour les
protéines plasmatiques (Gc, C3, HP, PLG, ORM et PI) et d’agglutination pour les
marqueurs de groupes sanguins (ABO, Kell et Rhésus). Ces marqueurs sont
répartis sur 12 chromosomes différents et représentent un total de 55 allèles.
ADNmt
L’ADN a été extrait de 47 échantillons sanguins de Corse du
Centre examinés pour les marqueurs génétiques classiques. L’extraction a été
réalisée à partir de sang total selon la méthode phénol-chloroforme.
La zone hypervariable I de ADNmt a été amplifiée par PCR
avec les amorces: L15996 (5’-CTCCACCATTAGCACCCAAAGC-3’) et H16401
(5’-TGATTTCACGGAGGATGGTG-3’), produisant un fragment de 445 bases.
Les produits de PCR ont été purifiés et 10 µl de ADNmt ont
été séquencés (ABI 377, Perkin Elmer) avec les amorces H et L utilisées pour la
PCR Les séquences ainsi obtenues couvraient un total de 360 bases, de L15981 à
L16340 et ont été comparées à la séquence de référence d’Anderson (1981).
Etude des ß Thalassémies
Les échantillons étaient composés de 43 hétérozygotes
ß-Thalassémiques non apparentés originaires du centre de l’île. Les patients
ont été sélectionnés à l’Hôpital de Bastia sur la base des paramètres
hématologiques suivants: MCV < 84 fL, MCH < 27 pg, HbA2 >
3.5%. Chaque donneur a été informé et a donné son accord pour participer à ce
programme de recherche.
A partir de l’ADN extrait, les mutations les plus
fréquemment rencontrées en Méditerranée ont été recherchées par dot-blot avec
des sondes oligonucléotidiques (ASO) (Ristaldi et al. 1989), par l’utilisation
d’enzymes de restriction et par la
méthode amplification refractory mutation system (ARMS) (Old 1990).
Les haplotypes ont été déterminés par l’analyse des sites de restriction polymorphes,
situés dans le domaine des gènes ß-Globine: (1) HincII 5’ de ![]() ; (2) HindIII 5’ de
; (2) HindIII 5’ de ![]() ; (3) HindIII dans la région 5’ de IVSII de
; (3) HindIII dans la région 5’ de IVSII de ![]() ; (4) HincII de
; (4) HincII de ![]() ; (5) HincII 3’ de
; (5) HincII 3’ de ![]() ; (6) AvaII de ß; et (7) HinfI 5’ de ß (Orkin et al. 1982). Les fragments contenant
chacun de ces sites ont été amplifiés puis digérés avec les enzymes appropriés.
; (6) AvaII de ß; et (7) HinfI 5’ de ß (Orkin et al. 1982). Les fragments contenant
chacun de ces sites ont été amplifiés puis digérés avec les enzymes appropriés.
Analyses statistiques
Marqueurs classiques
La différentiation entre la Corse et les autres populations Européennes et Méditerranéennes a été analysée avec la méthode R-Matrix (Harpending and Jenkins 1973) et les distances génétiques de Reynolds (Reynolds et al. 1983). L’ensemble des micro régions corses a été comparé avec des populations d’Afrique du Nord, d’Espagne, de France, de Grèce, d’Italie, du Pays Basque, du Portugal, de Sardaigne et de Turquie. Les distances génétiques ont été utilisées pour construire un arbre Neighbor Joining. La robustesse de ce dernier a été testée avec la méthode du Bootstrap en utilisant le programme PHYLIP 3.5 (Felsenstein 1989).
ADNmt
Les analyses statistiques ont été menées avec les
programmes PHYLIP 3.5c et ARLEQUIN 1.1 (Schneider et al. 1997). Le nombre
attendu de fréquences et la taille effective de la population ont été estimés
par le modèle des allèles infinis avec les moyennes des nombres d’individus et
de séquences (Hartl and Clark 1989). La variabilité interne des échantillons a
été analysée par l’indice de diversité de Shannon dans sa forme standardisée H’
(Magurran 1988).
La distribution du nombre moyen de différences et les
erreurs standards ont été calculées après 1000 itérations avec le programme
ARLEQUIN 1.1. Les paramètres ![]() et
et ![]() ont été calculés
en accord avec le modèle de Harpending et al. (1993). Les temps d’expansion ont
été estimés avec le paramètre
ont été calculés
en accord avec le modèle de Harpending et al. (1993). Les temps d’expansion ont
été estimés avec le paramètre ![]() en utilisant les
fréquences de mutation citées dans la littérature (Vigilant et al. 1991 ;
Ward et al. 1991).
en utilisant les
fréquences de mutation citées dans la littérature (Vigilant et al. 1991 ;
Ward et al. 1991).
Les séquences d’ADNmt relatives à la Corse ont été comparées avec celles des populations suivantes: Pays Basque (Bertranpetit et al. 1995), Sardaigne (Di Rienzo and Wilson 1991), Toscane (Francalacci et al. 1996), Turquie (Comas et al. 1996), Caucase (Stoneking 1993), Moyen Orient (Di Rienzo and Wilson 1991), Afrique (Vigilant et al. 1989), et Asie (Stoneking 1993). La comparaison a été effectuée par la méthode des distances basée sur le nombre moyen de substitution (Horai et Hayasaka 1990) et un arbre Neighbor Joining a été construit avec la matrice de distance obtenue (Saitou et Nei 1987).
ß Thalassémies
La fréquence de chaque mutation est déterminée par simple
comptage après analyse moléculaire.
Les fréquences haplotypiques à partir des 7 sites
polymorphes ont été déterminées par la méthode du maximum de vraisemblance
(Excoffier et Slatkin 1995). Cette analyse a été effectuée sur un
échantillonnage homogène caractérisé par la mutation majoritaire ß¼-39 et
comparée à un échantillonnage sain de même origine.
Résultats
Marqueurs autosomiques
Sur les 18 marqueurs étudiés seulement 5 se trouvent être en
déséquilibre selon la loi de Hardy-Weinberg: ESD et Tf (P<0,05), PGM1, PI et
Gc (P<0,01).
Les fréquences géniques (http://fst.univ-corse.fr/,
rubrique "La recherche", sous-rubrique "Lab génétique
humaine") obtenues à partir de l’ensemble de nos échantillons sont
comparées aux fréquences géniques relatives à l’Afrique du Nord, à l’Espagne, à
la France, à la Grèce, à l’Italie, au Pays Basque, au Portugal, à la Sardaigne
et à la Turquie.
L’analyse R-MATRIX
avec les populations citées montre que 54,25% de la variabilité est expliquée
par les deux premières composantes (38,99% pour la première et 15,26% pour la
seconde)
La Figure 1
visualise la projection des populations par rapport à ces deux premiers axes.
Hormis l’Afrique du Nord, toutes les autres populations sont rassemblées par la
première composante, par contre la seconde différencie les populations corse et
sarde des autres populations.
Les distances génétiques (Tableau 1, valeurs en haut de la diagonale) pour
la seule population corse, montrent que la plus faible distance est décelée
avec la Sardaigne, et la plus élevée avec le Portugal. De plus, les distances
moyennes les plus élevées sont trouvées pour l’Afrique du Nord (0,0347), la
Sardaigne (0,0282) et la Corse (0,0252), traduisant une dérive génétique plus
importante, comparativement aux autres populations, pour lesquelles la distance
moyenne s’échelonne entre 0,0150 et 0,0180.
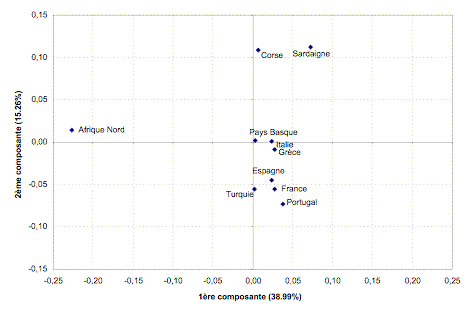
Figure 1. Projection des populations par rapport aux deux premières composantes de l’analyse R-Matrix.
|
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
|
1. Corse |
0,0156 |
0,0116 |
0,0205 |
0,0307 |
0,0242 |
0,0233 |
0,0317 |
0,0327 |
0,0182 |
0,0340 |
|
2. Sardaigne |
0,0104 |
0,0196 |
0,0177 |
0,0350 |
0,0296 |
0,0137 |
0,0492 |
0,0350 |
0,0259 |
0,0362 |
|
3. Grèce |
-0,0021 |
0,0015 |
0,0064 |
0,0156 |
0,0091 |
0,0051 |
0,0341 |
0,0088 |
0,0155 |
0,0099 |
|
4. France |
-0,0048 |
-0,0050 |
-0,0015 |
0,0051 |
0,0030 |
0,0162 |
0,0348 |
0,0166 |
0,0080 |
0,0083 |
|
5. Espagne |
-0,0029 |
-0,0046 |
-0,0005 |
0,0049 |
0,0064 |
0,0125 |
0,0316 |
0,0112 |
0,0087 |
0,0062 |
|
6. Italie |
-0,0028 |
0,0035 |
0,0026 |
-0,0015 |
-0,0012 |
0,0065 |
0,0344 |
0,0093 |
0,0177 |
0,0170 |
|
7; Afrique Nord |
-0,0007 |
-0,0144 |
-0,0060 |
-0,0072 |
-0,0062 |
-0,0048 |
0,0519 |
0,0316 |
0,0275 |
0,0378 |
|
8. Turquie |
-0,0065 |
-0,0053 |
0,0016 |
-0,0017 |
-0,0009 |
0,0026 |
-0,0023 |
0,0067 |
0,0195 |
0,0156 |
|
9. Pays Basque |
0,0007 |
-0,0005 |
-0,0040 |
0,0030 |
0,0011 |
-0,0042 |
-0,0015 |
-0,0040 |
0,0099 |
0,0132 |
|
10. Portugal |
-0,0070 |
-0,0053 |
0,0020 |
0,0037 |
0,0039 |
-0,0007 |
-0,0088 |
-0,0004 |
-0,0004 |
0,0110 |
Tableau
1. Distances de Reynolds (au dessus de la
diagonale) ; contribution à l’hétérogénéité de la représentation R-MATRIX
pour chaque population (en italique sur la diagonale). Valeurs de rij en
dessous de la diagonale.
Ces distances génétiques sont utilisées pour construire un arbre de
type Neighbor Joining après cent itérations (Figure 2). Cette représentation
confirme en partie la configuration de l’analyse R-MATRIX. Le groupe
Corse-Sardaigne éloigné des populations continentales et présentant une
communauté génétique d’importance croissante avec principalement l’Italie, le
Pays Basque et l’Afrique du Nord. Les valeurs de "bootstrap" aux
noeuds de l’arbre traduisent une robustesse suffisante de la représentation.
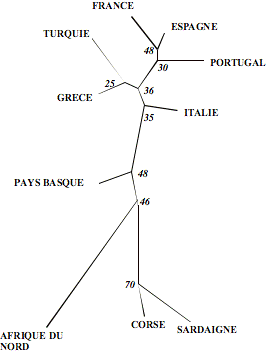
Figure 2. Arbre de type Neighbor Joining pour les populations comparées.
ADNmt
Les 47 individus étudiés présentent 31 séquences différentes avec en totalité 40 sites variables (http://fst.univ-corse.fr/, rubrique "La recherche", sous-rubrique "Lab génétique humaine").
Deux sites semblent spécifiques à la population corse et n’ont pas été
trouvés dans les populations envisagées pour cette étude: site 16179 (C®T) et 16252 (A®G).
Certaines mutations spécifiques des caucasoïdes montrent un gradient
d’est en ouest:
16069: Basques 4,4%, Sardes 5,8%, Corses 6,4%, Toscans 13,5%, Turcs
15,6%, Moyen Orient 19,0%.
16126: Basques 6,7%, Corses 8,5%, Sardes 13,0%, Toscans 23,1%, Turcs
26,7%, Moyen Orient 47,6%.
La mutation 16223 définit elle aussi un gradient significatif depuis
l’occident jusqu’à l’Afrique: Basques 4,4%, Corses 6,4%, Sardes 7,2%, Toscans
13,5%, Moyen Orient 23,8%, Turcs 26,7%,Asiatiques 47,8%, Africains 86,4%.
La variabilité des séquences est mesurée par l’indice de Shannon H’
(Magurran 1988) et est égal à 0,903 (Tableau 2), valeur intermédiaire comparée
aux autres populations caucasoïdes (0,899 pour la Toscane, 0,991 pour le Moyen
Orient).
La population corse présente un nombre moyen de substitution de 3,71
avec une variance de 6,13.
|
|
N |
K |
a |
b |
H' |
|
Corse |
47 |
31 |
40 |
3,18 |
0,903 |
|
Toscane |
52 |
40 |
54 |
5,03 |
0,899 |
|
Sardaigne |
69 |
46 |
52 |
4,22 |
0,930 |
|
Pays Basque |
45 |
27 |
32 |
3,15 |
0,912 |
|
Moyen Orient |
42 |
36 |
59 |
7,08 |
0,991 |
|
Asie |
23 |
23 |
59 |
8,28 |
1,000 |
|
Caucasoides |
18 |
16 |
29 |
5,15 |
0,976 |
|
Afrique |
22 |
12 |
34 |
8,66 |
0,946 |
|
Turquie |
45 |
38 |
56 |
5,38 |
0,987 |
Tableau 2. Paramètres de divergence nucléotidique. N= nombre d’individus ; k = nombre de séquences différentes ; a= nombre de sites polymorphes ; b= nombre moyen de substitutions. ; H’ = indice de Shannon.
La période d’expansion est déterminée en utilisant les trois taux de
mutations cités dans la littérature et avec q0 = 1.556, q1 = 14.869 et ![]() = 2.15 (Tableau 3), donnant ainsi une date moyenne de
27643 ans avant le présent, correspondant au paléolithique supérieur.
= 2.15 (Tableau 3), donnant ainsi une date moyenne de
27643 ans avant le présent, correspondant au paléolithique supérieur.
Les différences de distribution sont utilisées pour définir des
distances génétiques (Horai et Hayasaka 1990) entre les populations définies
dans Matériel et Méthodes. Ces distances (Tableau 4) sont utilisées pour
construire l’arbre de type Neighbor Joining (Saitou et Nei 1987) représenté à
la Figure 3.
|
|
|
µ= 4.14 x 10-6 |
µ= 2.16 x 10-6 |
µ= 1.44 x 10-6 |
|
Corse |
2.152 |
14443 |
27743 |
41584 |
|
Sardaigne |
4.052 |
27174 |
52083 |
78234 |
|
Toscane |
4.800 |
32206 |
61728 |
92593 |
|
Pays Basque |
2.138 |
14467 |
28074 |
42619 |
|
Caucasoides |
5.045 |
33859 |
65039 |
97587 |
|
Turquie |
5.385 |
38556 |
74566 |
102960 |
|
Moyen Orient |
7.019 |
47101 |
90278 |
135605 |
|
Asie |
7.555 |
50190 |
97172 |
145960 |
|
Afrique |
3.065 |
20361 |
39422 |
59215 |
Tableau 3. Temps de divergence calculés à partir de t, avec différents taux de mutation µ.
Les populations africaine et asiatique se détachent nettement des
populations européennes. Ces dernières plus homogènes, se positionnent selon un
gradient Est-Ouest. La population corse se trouvant étroitement liée à la
population basque et sarde, suivie ensuite par la Toscane.
|
|
Corse |
Sardaigne |
Toscane |
Pays Basque |
Caucasoïdes |
Turquie |
Moyen Orient |
Asie |
Afrique |
Corse
|
3.71 |
3.993 |
4.419 |
3.450 |
4.267 |
4.603 |
5.745 |
6.305 |
9.271 |
|
Sardaigne |
0.028 |
4.22 |
4.633 |
3.694 |
4.695 |
4.845 |
5.850 |
6.500 |
9.475 |
|
Toscane |
0.043 |
0.018 |
5.03 |
4.100 |
5.120 |
5.245 |
6.155 |
6.885 |
10.044 |
|
Pays basque |
0.020 |
0.009 |
0.010 |
3.15 |
4.280 |
4.334 |
5.553 |
6.055 |
9.610 |
|
Caucasoïdes |
0.117 |
0.020 |
0.030 |
0.130 |
5.15 |
5.306 |
5.980 |
6.275 |
9.740 |
|
Turquie |
0.058 |
0.045 |
0.040 |
0.069 |
0.041 |
5.38 |
6.321 |
6.995 |
9.832 |
|
Moyen Orient |
0.350 |
0.200 |
0.100 |
0.438 |
0.160 |
0.091 |
7.08 |
7.900 |
10.765 |
|
Asie |
0.310 |
0.250 |
0.230 |
0.340 |
0.140 |
0.165 |
0.220 |
8.28 |
10.655 |
|
Afrique |
5.821 |
5.770 |
5.930 |
6.340 |
5.570 |
5.547 |
5.630 |
4.920 |
3.19 |
Tableau
4. Distances génétiques basées sur le nombre moyen de
substitutions. Sur la diagonale (en italique) nombre moyen de substitution
intrapopulation. Au dessus de la diagonale: nombre moyen de substitution
interpopulation. Au dessous de la diagonale: distance Dij = dij - ((di+dj)/2).
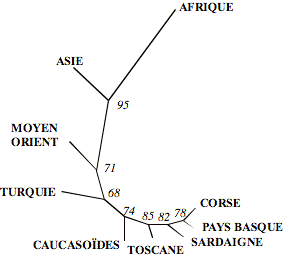
Figure 3. Arbre de type Neighbor Joining construit à partir des distances génétiques Dij.
Etude des ß Thalassémies
Sur les 43 individus testés, 38 présentent une mutation en position
codon 39 (88,40 %), 1 individu seulement est caractérisé par la mutation IVS I-
110 (2,33%) et 4 individus n’ont pu être typés (9,30 %).
L’homogénéité mutationnelle dans cette micro-région centrale, est
légèrement supérieure à celle établie pour l’ensemble de la Corse du Nord
(Varesi et al. 2000).
Ceci peut être expliqué par l’origine géographiquement limitée des
individus.
Ces deux mutations se retrouvent dans toutes les populations
méditerranéennes avec des fréquences variables.
Les populations insulaires présentent en général une homogénéité très
importante: la Corse et la Sardaigne pour la mutation ߺ-39, et Chypre pour la
mutation IVS I-110. Dans ces régions une ou deux mutations représentent 90 % environ
des ß Thalassémies, ce qui facilite le dépistage prénatal éventuel.
Les fréquences haplotypiques liées à la mutation ߺ-39, sont établies à
partir des 38 thalassémiques ߺ-39 et de 47 individus sains, originaires de la
même région (Falchi et al. 2005).
La mutation ߺ-39 se retrouve dans le centre de la Corse avec une
prévalence de 88,40 %, en seconde position, par ordre d’importance, après la
Sardaigne voisine (95,40 %).
Cette mutation prépondérante, dans la partie méditerranéenne occidentale, semble avoir dans les deux îles subi l’influence d’un effet fondateur.
La répartition des deux principales mutations, ߺ-39 et IVS I- 110,
dans des pays principalement méditerranéens à partir des données issues de la
littérature est illustrée par la Figure 4 (Cao et al. 1989 (a; b); Giambona et
al. 1995; Zahed et al. 1997; Perrin et al. 1998; Cabeda et al. 1999).
Ces deux mutations se répartissent différemment d’Est en Ouest, la
partie occidentale caractérisée par la mutation ߺ-39 et la partie orientale
par la mutation IVS I-110.
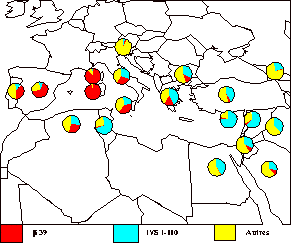
Figure 4. Répartition géographique des deux principales mutations du gène de la b globine dans la zone euroméditerranéenne.
La plupart des régions et pays étudiés ci dessus ont, durant des
siècles, voire des millénaires subis la pression sélective de la malaria. Or,
les ß Thalassémies sont connues pour donner un effet protecteur vis-à-vis du
Plasmodium (Cavalli Sforza et al. 1994).
La question est de savoir si ces mêmes mutations sont apparues, sous
l’effet de la même pression, d’une façon indépendante dans des populations
isolées les unes des autres (origine mutationnelle multicentrique). Ou bien
ont-elles pour origine, un ou quelques foyers humains, et se sont ensuite
répandues grâce à des évènements migratoires.
L’étude des haplotypes associés à la mutation majoritaire ߺ-39
pourrait apporter quelques éléments de réponse. Les associations haplotypiques
ont été définies en utilisant la méthode du maximum de vraisemblance. Les
résultats obtenus sont reportés sur le Tableau 5
La mutation ߺ-39 se trouve être associée majoritairement avec
l’haplotype II (fréquence: 0,187).
Des associations minoritaires sont détectées avec l’ haplotypes I
(fréquence: 0,023) et l’haplotype VII (fréquence: 0,021).
Des études de corrélation entre ß Thalassémies et haplotypes ont permis
d’établir que la mutation ߺ-39 est, dans de nombreuses populations
méditerranéennes préférentiellement associée à l’haplotype II, et à l’haplotype
I.
La mutation IVS I-110 a très souvent été décrite comme étant associée
presque exclusivement à l’haplotype I (Thein et al. 1987).
Les associations minoritaires semblent plutôt être issues de
recombinaisons méiotiques (Fullerton et al. 1994).
|
Haplotypes |
Fréquences |
Hinc-II
|
Hind-III |
Hind-III |
Hinc-II |
Hinc-II |
Ava-II |
Hinf-I |
ߺ39 |
||
|
I |
0,236 |
±0,012 |
+ |
- |
- |
- |
- |
+ |
+ |
- |
|
|
I |
0,023 |
±0,001 |
+ |
- |
- |
- |
- |
+ |
+ |
+ |
|
|
II |
0,109 |
±0,008 |
- |
+ |
+ |
- |
+ |
+ |
+ |
- |
|
II
|
0,187 |
±0,014 |
- |
+ |
+ |
- |
+ |
+ |
+ |
+ |
|
|
III |
0,123 |
±0,010 |
- |
+ |
- |
+ |
+ |
+ |
- |
- |
|
|
V |
0,066 |
±0,004 |
+ |
- |
- |
- |
- |
+ |
- |
- |
|
|
VII |
0,048 |
±0,005 |
+ |
- |
- |
- |
- |
- |
+ |
- |
|
|
VII |
0,021 |
±0,002 |
+ |
- |
- |
- |
- |
- |
+ |
+ |
|
|
VIII |
0,026 |
±0,004 |
- |
+ |
- |
+ |
- |
+ |
- |
- |
|
|
IX |
0,009 |
±0,001 |
- |
+ |
- |
+ |
+ |
+ |
+ |
- |
|
|
1 |
0,092 |
±0,006 |
+ |
- |
- |
- |
+ |
+ |
+ |
- |
|
|
2 |
0,028 |
±0,004 |
- |
+ |
- |
- |
+ |
+ |
- |
- |
|
|
3 |
0,032 |
±0,006 |
+ |
- |
+ |
+ |
- |
+ |
+ |
- |
|
Tableau
5. Fréquences haplotypiques des échantillons étudiés
avec la nomenclature de ORKIN et al. (1982).
Discussion et conclusions
L’étude des marqueurs classiques montre un certain nombre de
caractéristiques génétiques:
- Une forte similitude relie les populations corse et sarde (Figure 2).
- Une hétérogénéité est décelée au sein de ces mêmes populations
(Tableau 1), probablement liée à l’isolement et à leur structure démographique.
- La répartition des autres populations sur l’arbre (Figure2), du fait
des valeurs de Boostrap peu significatives, est à considérer avec précaution.
La population d’Afrique du Nord avec une valeur de Boostrap à la limite de la
significativité (46%) occupe une position qui semble en désaccord avec
l’analyse R matrix (Figure 1). Si l’on considère la forte distance génétique
entre cette population et le groupe corso sarde, il serait logique de ne pas
considérer ce rapprochement. Le cas de la population du pays Basque est différent, malgré la valeur de
Bootstrap, également proche de la significativité (48%), elle présente une
faible distance génétique avec le groupe insulaire (Tableau 1). De plus ce
rapprochement est également visualisé avec l’ analyse R matrix et surtout avec
l’étude de l’ADNmt (Figure 3).
- Une influence modérée est visualisée avec les populations de la
péninsule italienne.
- L’influence française demeure pratiquement inexistante, sans doute
trop récente. De plus l’enquête généalogique permet, à travers les noms,
d’éliminer les migrants ou leurs descendants.
Si les généticiens s’accordent sur l’origine ancestrale de la structure
génétique actuelle des populations méditerranéennes, ils restent par contre
partagés entre une origine paléolithique et néolithique.
Cavalli-Sforza et al. (1994) en utilisant les fréquences géniques de
nombreux marqueurs protéiques, ont démontré que leurs variations étaient corrélées
géographiquement aux sites néolithiques. Leur modèle assure que notre structure
génétique actuelle provient de la lente expansion des fermiers néolithiques à
partir du Moyen Orient. Le nouveau mode économique a permis une croissance
démographique qui déplaça (ou absorba) le faible nombre de chasseurs cueilleurs
mésolithiques.
Un modèle opposé (Richard et al. 1996) propose, au contraire, une
simple diffusion culturelle avec une intrusion minimale des peuples du Moyen
Orient.
L’analyse de l’ADNmt de la population corse semble confirmer les
observations publiées par un certain nombre d’auteurs, montrant, en Europe, un
gradient Est-Ouest à partir d’une population ancestrale unique (Francalacci et
al. 1996 ; Comas et al. 1997). Les
valeurs de l’indice de Shannon H’ et du nombre moyen de substitution vont
également dans ce sens (Tableau 2).
La date d’expansion démographique de la population corse, une des plus
récentes, se situerait entre 14443 et 41584 ans avant présent (AP), durant le
paléolithique, sensiblement durant la même période que l’expansion de la
population basque: 14467 à 42619 (Bertranpetit et al. 1995).
L’expansion sarde, plus ancienne, se situerait entre 27174 et 78234 ans
AP (Comas et al. 1997).
La structure génétique des populations étudiées semble quasiment
établie au paléolithique supérieur, ceci est donc en accord avec le second
modèle décrit par Richards et al. (1996).
Le peuplement se serait donc effectué, depuis le Moyen Orient, à partir de 35000 ans environ avant notre ère, qui correspond à la première phase du retrait des glaciers en Europe. L’occupation des territoires par nos ancêtres a dû certainement n’être que sporadique. En Europe, le véritable essor démographique a dû intervenir à la fin de la dernière glaciation, il y a 15000 ans environ. Les expansions des fermiers du néolithique, à partir de 10000 ans avant notre ère, auraient eu, en Méditerranée, une influence technologique plus que génétique.
En considérant
l’unité du bloc corso-sarde et les facteurs climatiques au paléolithique, ainsi
que les caractéristiques géographiques, il serait logique de penser que le
peuplement le plus ancien concerna la partie la plus propice à une implantation
humaine: la Sardaigne actuelle. Ces conditions sont attestées par les traces de
glaciers quaternaires en Corse, indiquant la présence de trois épisodes
glaciaires durant le paléolithique. Ces traces ne se retrouvent pas en
Sardaigne, qui à la même époque, connu des épisodes de forte pluviométrie
(Vardabasso-Bonicelli 1968). Ce climat, plus favorable, aurait attiré les
groupes humains vers la partie méridionale du bloc corso-sarde, par la suite,
le réchauffement progressif, à partir de 15000 ans environ AP, en supprimant
les "ponts" avec l’Italie continentale, aurait favorisé une expansion
vers le Nord. Cette hypothèse semble être confortée par les données obtenues à
partir de marqueurs protéiques (Varesi et al. 1996, Vona et al. 1995; Moral et
al. 1996; Memmi et al. 1998), et nucléaires (Calo et al. 1998, Vona et al.
1996).
Ce scénario semble
conforté par le gradient des temps de divergence, obtenus avec l’étude de
l’ADNmt, depuis la Sardaigne centrale jusqu’au centre de la Corse.
La similitude génétique entre Corses et Sardes, révélée avec
différentes approches, peut s’expliquer par les échanges constants qui ont eu
lieu entre les deux populations depuis la Préhistoire jusqu’au Moyen Age.
L’influence des mêmes envahisseurs aux même périodes n’a été que très limitée,
comme le montre nos résultats. En complétant notre raisonnement, nous pouvons avancer
l’hypothèse que les deux populations insulaires ont eu la même origine de
peuplement, et que cette similitude génétique actuelle n’est autre que le
reflet d’un patrimoine génétique ancestral commun. Ceci semble confirmé par les
nombreuses ressemblances archéologiques sur plusieurs millénaires (Tanda 1995;
Lilliu 1988; Mazzia 1975). Par la suite, les visées politiques de la République
de Gènes d’une part, et des Aragonais d’autre part, initièrent le
ralentissement des échanges entre les deux îles. Le facteur démographique
accentua l’effet de dérive pour les deux populations insulaires, ce qui se
traduit par la différenciation mise en évidence dans notre étude.
Cette similitude est également décelée avec des marqueurs sélectifs
tels que les mutations du gène de la ß globine.
Dans les régions méditerranéennes l’apparition de la malaria a
probablement eu lieu lorsque les conditions géoclimatiques étaient favorables
au Plasmodium, c’est-à-dire lors de la montée des eaux (formation de zones
lacustres) associée à un réchauffement terrestre. Cette période eut lieu il y a
3 000 ans environ avant notre ère, à la fin du néolithique (Conchon 1987).
Cette période correspond également à un échange de cultures, de
techniques et d’idées dans tout le bassin méditerranéen: agriculture, industrie
du cuivre, culture mégalithique, etc. (Camps 1988).
Ces contacts humains étaient très fréquents entre peuples de la
Méditerranée occidentale, comme le démontrent les découvertes archéologiques.
La mutation ߺ-39 dans la population du centre de la Corse montre donc
une forte association avec l’haplotype II, les valeurs de fréquences obtenues,
soulignent l’importante homogénéité génétique de cette micro-région, tout au
moins au niveau du complexe bêta globine. Cette association quasi exclusive se
retrouve également en Sardaigne, laissant supposer que les habitants des deux
îles ont été en contact avec un seul foyer humain thalassémique ߺ-39 et que le
maintient de cette mutation a été favorisé par un effet fondateur du à
l’insularité et à la présence endémique de la malaria.
Remerciements. Ces recherches ont été financées par les programmes
de l’Union Européenne INTERREG 1, 2 et 3.
Références bibliographiques
Anderson, S., Bankier, A.T., Barrell, B.G., De Bruijn,
M.H.L., Coulson, A.P., Drouin, J., Eperon, I.C., Nierlich, D.P., Roe, B.A.,
Sanger, F., Schreier, P.H., Smith, A.J.H., Staden, R., Young, I.G., 1981,
Sequence and Organisation of the Human Mitochondrial Genome. Nature, 290,
457-465.
Bertranpetit, J., Sala, J., Calafell, F., Underhill,
P.A., Moral, P., Comas, D., 1995, Human Mitochondrial DNA Variation and the
Origin of Basques. Ann.Hum.Genet, 59, 63-81.
Cabeda, J.M., Correia, C., Estevinho, A., Simoes, C.,
Amorim, M.l., Pinho, L., Justica, B., 1999, Unexpected pattern of beta globin
mutations in beta thalassemia patients from northern Portugal. Br. J.
Haematology, 105(1), 68-74.
Calo, C.M., Autori, L., Di Gaetano, C., Latini, V.,
Mameli, G.E., Memmi, M., Varesi, L. , Vona, G., 1998, The polymorphism of the
Apo B 3’ VNTR in the Western Mediterranean. Anthrop Anz , 56(3 ), 1-11.
Camps, G. 1988, Préhistoire d’une île. Editions
Errance. Paris.
Cao, A., Pirastu, M., Ristaldi, M.S., Leoni, G.B.,
Rosatelli, M., 1989a, ß thalassemias: Mediterranean aspects. Molecular probes,
Ed. Raven Press NY, pp257.
Cao, A., Rosatelli, C., Galanello, R., Monni, G.,
Olla, G., Cossu, P., Ristaldi, S., 1989b, The prevention of thalassemia in
Sardinia. Clinical Genetics, 36, 277-285.
Cavalli-Sforza, L.L., Menozzi, P., Piazza A., 1994,
History and Geography of Human Genes. Princeton, NJ: Princeton Univ.Press,
518p.
Comas, D,. Calafell, F., Mateu, E., Perez-Lezun, A.,
Bertranpetit, J., 1996, Geographic Variation in Human Mt DNA Control Region
Sequence: the Population History of Turquey and its Relationship to the
European Populations. Mol. Biol. Evol., 13, 1067-1077.
Comas, D., Calafell, F., Mateu, E., Perez-Lezaun, A.,
Bosch, E., Bertranpetit, J., 1997, Mitochondrial DNA variation and the origin
of the Europeans. Hum. Genet., 99(1), 443-449.
Conchon, O., 1987, Corrélations entre les formations
glaciaires, fluviatiles et marines de Corse et les sédiments sous marins de
Méditerranée occidentale au Pléistocène supérieur. Revue Géol. Dyn. Géogr.
Phys., 27(2), 85-93.
Di Rienzo, A., Wilson, A.C., 1991, Branching Pattern
in the Evolutionary Tree for Human Mitochondrial DNA. Proc.Natl.Acad.Sci.USA.,
88, 1597-1601.
Excoffier, L., Slatkin, M., 1995, Maximum likelihood
estimation of molecular haplotype frequencies in a diploid population. Mol.
Biol. Evol., 12(5), 921-927.
Falchi, A., Giovannoni, L., Vacca, L., Latini, V.,
Vona, G., Varesi, L., 2005, Beta-globin gene cluster haplotypes associated with
beta-thalassemia on Corsica island. Am J Haematol, 78(1), 27-32.
Felsentein, J., 1989, PHYLIP, Physiology influence
package (version 3.2). Clastic 5, 164-166.
Francalacci, P., Bertranpetit, J., Calafell, F.,
Underi11, P.A., 1996), Sequence Diversity of the Control Region of
Mitochondrial DNA in Tuscany and its Implications for the Peopling of Europe.
Am.J.Phys Anthropol, 100, 443-460.
Fullerton, S.M., Harding R.M., Boyce A.J., Clegg J.B.,
1994, Molecular and population genetic analysis of allelic sequence diversity
at the human beta-globin locus. Proc. Natl. Acad. Sci. USA, 91(5), 1805-1809.
Giambona, A., Lo Gioco, P., Marino, M., 1995, The
great heterogeneity of thalassemia molecular defects in Sicily. Hum. Genet.,
95, 526-530.
Harpending, H.C., Jenkins, T., 1973, Genetic distance
among Southern African populations. Methods and theories in Anthropological
Genetics, University of New Mexico Press, Albuquerque, 177-199.
Harpending, H.C., Sherry, S.T., Rogers, A.R.,
Stoneking, M., 1993, The genetic structure of ancient human populations.
Current Anthropology, 34, 483-496.
Hartl, D.L., Clark, A.G., 1989, Principles of
population genetics. Sunderland, MA: Sinauer Associates.
Horai, S., Hayasaka, K., 1990, Intraspecific
Nucleotide Sequence Differences in the Major Noncoding Region of Human
Mitochondrial DNA. Am. J. Hum. Genet., 46, 828-842.
Lilliu, G., 1988, La civilità dei Sardi. TORINO NUOVA
RI 3n ed., 320p.
Magurran, A.E., 1988, Ecological diversity and its
measure. Princeton: Princeton University Press.
Mazzia, C., 1975, Uomo e ambiante in Corsica dalla
preistoria ai tempi attuali. Rendiconti del seminario della Facoltà di Scienze
dell’ Univerisità di CAGLIARI, XLV, 253-323.
Melillo, A.M., 1971, Profilo dei dialetti italiani:
Corsica. Pisa Ed Pacini.
Memmi, M., Moral, P., Calo, C.M., Autori, L., Mameli,
G.E., Succa, V., Varesi, L., Vona, G., 1998, Genetic structure of southwestern
Corsica (France). American Journal of Human Biology, 10, 567-577.
Moral, P., Varesi, L., Memmi, M., Mameli, G.E., Succa,
V., Guttierrez, B., Lutken, N., Vona, G., 1996, Study on the variability of
seven serum protein genetic markers in Corsica (France). Anthropologisher
Anzeiger, 54(2), 97-107.
Old, J.M., Varawalla, N.Y., Weatherall, D.J., 1990,
Rapid detection and prenatal diagnosis of ß thalassemia: studies in Indian and
Cypriot population in the UK. The Lancet, 336, 834-837.
Orkin, S.H., Kazazian H.H., Antonarikis S.E., 1982,
Linkage of ß-thalassemia mutations and ß-globin gene polymorphism in human
globin gene cluster. Nature, 296, 627-631.
Perrin, P., Bouhassa, R., Mselli, L., Garguier, N.,
Nigon, V.M., Bennani, C., Labie, D., Trabuchet, G., 1998, Diversity of sequence
haplotypes associated with beta thalassemia mutations in Algeria: implication
for their origin. Gene, 213(1-2), 169-177.
Reynolds, J.B., Weir, B.S., Cokerham, C.C, 1983,
Estimation of coancestry coefficient:basis of a short-term genetic distance.
Genetics, 105, 767-779.
Richards, M., Corte-Real, H., Forster, P., Maccaulay,
V., Wilkinson-Herbots, H., Demaine, A., Papiha S., Hedges, R., Bandelt, H.J.,
Skyes, B., 1996, Paleolithic and Neolithic Lineages in the European
mitochondrial gene pool. Am. J. Hum. Genet., 59, 185-203.
Ristaldi, M.S., Pirastu, M., Rosatelli C., Monni, G.,
Erlich, H., Saiki, R., Cao, A., 1989, Prenatal diagnosis of beta-thalassemia in
Mediterranean populations by dot blot analysis with DNA amplification and
allele specific oligonucleotide probes. Prenat. Diagn., 9(9), 629-638.
Rohlfs, G., 1972, Studi e Ricerche su Lingua e
Dialetti d’Italia. Firenze Ed Sensoni.
Saitou, N., Nei, M., 1987, The Neighbor-joining
Method: a New Method for Reconstructing Phylogenetic Trees. Mol. Biol. Evol.,
4(4), 406-425.
Schneider S., Kueffer J.M., Roessli D., Excoffier L.,
1997, Arlequin 1.1: A software for population genetic data analysis. User
manual. Genetics and Biometry Lab, University of Geneva.
Stoneking, M., 1993, DNA and Recent Human Evolution.
Evol. Anthropol., 2, 60-73.
Tanda, G., 1995, I siti del neolitico antico e
l’ambiante in Sardegna. Anthropologia Contemporanea, 18(3-4), 5-17.
Thein, S.L., Wainscoast, J.S., Sampietro, M., Old,
J.M., Cappellini, D., Fiorelli, G., Modell, B., Weatherall, D.J., 1987,
Association of thalassemia intermedia with a beta-globin gene haplotype. Br. J.
Haematol., 65(3), 367-373.
Vardabasso-Bonicelli, S., 1968, Il quartenario lungo
la facscia costiera della Sardegna. Atti del X Congresso di Studi Sardi Ed.
Sarda Fossataro Cagliari.(I).
Varesi, L., Memmi, M., Moral, P., Mameli, G.E., Succa,
V., Vona, G., 1996, La Distribution de Quatorze Marqueurs Génétiques dans la
Population de l'Ile de Corse. Bulletin et Mémoires de la Société
d'Anthropologie de PARIS, 1(2), 5-14.
Varesi, L., Vona, G., Memmi, M., Marongiu, M.F.,
Ristaldi, M.S., 2000, Beta thalassemia mutations in Corsica. Hemoglobin, 24(3),
239-244.
Vigilant, L., Pennington, R., Harpending, H., Kocher,
T.D., Wilson, A.C., 1989, Mitochondrial DNA sequences in single hairs from a
Southern African population. Evolution, 86, 9350-9354.
Vigilant, L., Stoneking, M., Harpending, H., Hawkes,
K., Wilson, A.C., 1991, African Populations and the Evolution of Mitochondrial
DNA. Science, 253, 1503-1507.
Vona, G., Memmi, M., Varesi, L., Mameli, G.E., Succa,
V., 1995, Study of several red cell enzyme markers in Corsica. Antropologisher
Anzeiger, 53(2), 125-132.
Vona, G., Varesi, L., Memmi, M., Mameli, G.E., Calo,
C.M., Esteban, E., Moral, P., 1996, Genetic structure of Corsica (FRANCE). 10th
Congress of the European Anthropological Association, Brussels (B).
Ward, R.H., Frazier, B.L., Dew-Jager, K., Pääbo, S.,
1991, Extensive mitochondrial diversity within a single Amerindian tribe. Proc.
Natl. Acad. Sci. USA, 88, 8720-8724.
Zahed, L., Talhouk, R., Saleh, M., Abou-Jaoudeh, R., Fisher, C., Old, J., 1997, The spectrum of ß thalassemia in the Lebanon. Hum. Hered., 47, 241-249.
