
Colloques du
Groupement des Anthropologistes de Langue Française (GALF)
Buhler, S., Sanchez-Mazas, A., 2006, De l’Europe à l’Inde: structure génétique et diversité des populations de part et d’autre de leurs frontières géographiques et culturelles. Antropo, 11, 249-259. www.didac.ehu.es/antropo
De l’Europe à l’Inde: structure génétique et diversité des
populations de part et d’autre de leurs frontières géographiques et culturelles
From Europe to India: genetic structure
and diversity of populations across their geographic and cultural boundaries
Stéphane Buhler1 et
Alicia Sanchez-Mazas2
1Laboratoire d’Anthropologie, Génétique et Peuplements,
Département d’Anthropologie et d’Ecologie, Université de Genève, 12 rue
Gustave-Revilliod, 1227 Genève, Suisse. E-mail: stephane.buhler@anthro.unige.ch
2Laboratoire d’Anthropologie, Génétique et Peuplements,
Département d’Anthropologie et d’Ecologie, Université de Genève, 12 rue
Gustave-Revilliod, 1227 Genève, Suisse. E-mail: alicia.sanchez-mazas@anthro.unige.ch
Mots-clés:
Anthropologie, polymorphisme
génétique, HLA-C, populations humaines, Europe, Proche-Orient, Inde-Pakistan
Keywords:
Anthropology, genetic
polymorphism, HLA-C, human populations, Europe, Middle East, India-Pakistan
Résumé
Dans cette étude, nous nous sommes intéressés au polymorphisme
moléculaire du gène HLA-C au sein de 39 populations humaines localisées entre
l’Europe et le sous-continent indien. Cette aire géographique correspond plus
ou moins à la zone de locution des langues du phylum indo-européen, mais des
populations de langues afro-asiatique et dravidienne sont aussi intégrées dans
les comparaisons.
Nos résultats nous indiquent que la géographie est l’un des facteurs déterminant les différenciations des populations situées en Europe. De plus, une frontière génétique est repérée le long d’un axe nord-est / sud-ouest à hauteur des Alpes et à travers les Pyrénées, qui met grosso modo en évidence une séparation entre les populations du pourtour méditerranéen et celle d’Europe de l’ouest. En revanche, on ne retrouve pas de corrélation entre géographie et génétique au Proche-Orient ni dans le sous-continent indien. Si la géographie ne semble pas jouer de rôle prépondérant au sein de ces deux groupes, les différenciations génétiques s’expliquent de manière très différente dans ces deux cas. En effet, les populations proche-orientales constituent un groupe génétiquement homogène, ce qui s’expliquerait par un taux élevé de flux génique dû aux migrations dont cette région, située à un carrefour continental, a été le théâtre à travers l’histoire. Au contraire, les populations du groupe indo-pakistanais sont très hétérogènes. La différenciation de ces dernières, dont plusieurs parlent une langue dravidienne, semble avoir été gouvernée par une dérive génétique rapide sous l’influence complexe de barrières culturelles, sociales, linguistiques et/ou religieuses.
Abstract
In this study, we compared HLA-C molecular polymorphism in 39 human
populations located from Europe to the Indian subcontinent. This geographic
area encompasses more or less the spoken area of the Indo-European linguistic
phylum, but some Afro-asiatic and Dravidian speakers were also considered in
the comparisons.
Our results indicate that geography is one of the main factors acting on
population differentiations in Europe. Moreover, we detect a genetic boundary following a north-east / south-west axis cutting through the Alps
and the Pyrenees and which separates the western European populations from
those located around the Mediterranean basin. On the other hand, we do not find
any significant correlation between geography and genetics in the Middle East
and the Indian subcontinent. Although geography does not play a major role in
these two regions, the factors that could explain the observed genetic
diversity are not the same. Indeed, the Middle Eastern populations constitute a
genetically homogeneous group, which could reflect a high level of gene flow
due to the numerous migrations that took place in that region throughout
history. Conversely, the Indian-Pakistani populations are very heterogeneous
genetically, particularly the Dravidian speakers. The differentiation of these
populations seems to result from rapid genetic drift under the complex
influence of social, cultural, linguistic and religious boundaries.
Introduction
Le Complexe Majeur d’Histocompatibilité (MHC), l’un des systèmes génétiques les plus polymorphes chez l’Homme, est un important marqueur pour étudier les relations génétiques entre populations. En effet, outre son rôle crucial en médecine clinique et en transplantation d’organes et de cellules souches hématopoïétiques, ce système est très informatif pour comparer les profils génétiques des populations humaines en relation avec des événements historiques et des migrations passées. Jusqu’à récemment, le gène HLA-C faisait figure d’exception en raison de limitations méthodologiques liées à la caractérisation sérologique peu fiable des différentes spécificités génériques répertoriées à ce locus (Dard, 1991; Bunce et al., 1997a; Mytilineos et al., 1997). Toutefois, la mise en place de techniques moléculaires, telles que le séquençage, ou les protocoles basés sur la PCR (Kennedy et al., 1995; Bunce et al., 1997b; Turner et al., 1998), ont permis de caractériser plus de 200 variants alléliques à ce locus, ouvrant la voie à de nombreuses études anthropologiques (Sanchez-Mazas et al., 2000; Buhler, 2001; Buhler et al., 2002; Sanchez-Mazas, sous presse).
La zone de répartition des locuteurs du phylum indo-européen, qui s’étend sur une aire géographique chevauchant l’Europe et l’Asie du sud-ouest, a été le théâtre d’importants mouvements de populations à travers l’histoire, mais également d’événements plus localisés qui ont pu avoir une influence importante sur les différenciations génétiques de ces populations. Dans cette étude, nous nous sommes donc intéressés à voir dans quelle mesure interviennent des facteurs géographiques et/ou culturels dans ces différenciations, d’une part à l’intérieur du groupe indo-européen dans son ensemble, d’autre part entre les trois régions géographiques principales définies a priori - Europe, Proche-Orient, et Inde / Pakistan -, et, enfin, à l’intérieur de chaque région, entre entités linguistiques différentes.
Matériel
et méthodes
Nous disposons d’un ensemble de 39 populations réparties comme suit entre les trois régions géographiques considérées: 16 en Europe, 8 au Proche-Orient, et 15 dans le sous-continent indien. Ces populations consistent en des données publiées qui ont été intégrées à la base de données Gene[VA] maintenue par A. S.-M. La majorité de celles-ci, soit un total de 26, est composée de locuteurs du phylum indo-européen, le reste des populations se répartissant en locuteurs afro-asiatiques (3 populations), dravidiens (3 populations), altaïques-turkiques (1 population), ouraliques-youkhagir (1 population), sud-caucasiens (1 population), ainsi que 4 isolats linguistiques.
|
Population |
Pays, Localité |
N |
Références |
|
Suisses |
Suisse, Genève |
99 |
(Sanchez-Mazas et al., 2000; Buhler et al., soumis) |
|
Irlandais |
Irlande du Nord |
1000 |
(Williams et Middleton, 2004; Mack et al., sous presse) |
|
Anglais |
Royaume-Uni |
604 |
(Bunce et al., 1997a) |
|
Tchèques |
Tchéquie, Prague |
105 |
(Zahlavova et al., 2004; Mack et al., sous presse) |
|
Finnois |
Finlande |
90 |
(Mack et al., sous presse) |
|
Corses |
France, Corse |
100 |
(Grimaldi et al., 2001) |
|
Basques |
France |
67 |
(Grimaldi et al., 2001) |
|
Grecs |
Grèce |
246 |
(Papassavas et al., 2000) |
|
Bulgares |
Bulgarie |
128 |
(Ivanova et al., 2001) |
|
Catalans |
Espagne, Gérone |
87 |
(Comas et al., 1998) |
|
Basques |
Espagne, province de Gipuzkoa |
99 |
(Comas et al., 1998) |
|
Baléares |
Espagne, îles Baléares |
90 |
(Grimaldi et al., 2001) |
|
Sardes |
Italie, Sardaigne |
100 |
(Grimaldi et al., 2001) |
|
Italiens |
Italie, Bergame |
101 |
(Piazza et Lonjou, 1997; Ferrara et al., 1998a; Ferrara et al., 2004a) |
|
Italiens |
Italie du Nord |
97 |
(Ferrara et al., 1998b; Ferrara et al., 2004b) |
|
Italiens |
Italie, Rome |
100 |
(Testi et Mariani, 1998; Testi et Mariani, 2004) |
|
Libanais |
Liban |
97 |
(Buhler et al., soumis) |
|
Ashkénazes |
Nord du Caucase |
412 |
(Cox et al., 1999) |
|
Iraniens |
Iran, Téhéran |
120 |
(Buhler et al., 2002) |
|
Géorgiens |
Géorgie, Tbilisi |
107 |
(Mack et al., sous presse) |
|
Kurdes |
Géorgie, Tbilisi |
29 |
(Mack et al., sous presse) |
|
Juifs |
Israël |
94 |
(Mack et al., sous presse) |
|
Druzes |
Israël |
100 |
(Mack et al., sous presse) |
|
Turcs |
Turquie, Istanbul |
142 |
(Uyar et al., 2004) |
|
Hunzas-Burushaski |
Pakistan, Gilgit |
46 |
(Qasim Medhi et al., 1997) |
|
Burusho |
Pakistan, montagnes du Karakoram |
98 |
(Mohyuddin et al., 2002) |
|
Baloutches |
Pakistan, Baloutchistan |
66 |
(Mohyuddin et al., 2002) |
|
Sindhi |
Pakistan, Sindh |
39 |
(Qasim Medhi et al., 1997) |
|
Brahui |
Pakistan, Baloutchistan |
104 |
(Mohyuddin et al., 2002) |
|
Kalash |
Pakistan, montagnes Hindu Kush |
69 |
(Mohyuddin et al., 2002) |
|
Pathans |
Pakistan, province du Nord-Ouest, et Balouchistan |
100 |
(Mohyuddin et al., 2002) |
|
Nadars |
Inde, Virudhunagar, Tamil Nadu |
61 |
(Shankarkumar et al., 2003) |
|
Indiens |
Inde, New Delhi |
106 |
(Mack et al., sous presse) |
|
Tamils |
Inde, Tamil Nadu |
48 |
(Mack et al., sous presse) |
|
Pawra |
Inde, Kandesh |
50 |
(Shankarkumar et al., 1999; Shankarkumar,
2004a) |
|
Marathas |
Inde, Bombay |
91 |
(Shankarkumar et al., 2001;
Shankarkumar, 2004b) |
|
Bhils |
Inde de l’Ouest |
50 |
(Shankarkumar et al., 1999;
Shankarkumar, 2004c) |
|
Parsi |
Inde de l’Ouest |
50 |
(Shankarkumar, 2002; Shankarkumar, 2004d) |
|
Baloutches |
Iran, Baloutchistan |
100 |
(Farjadian et al., 2004) |
Tableau 1. Populations considérées dans cette étude. N: taille de
l’échantillon.
Table 1. Populations compared in this study. N: sample size
Les différentes analyses décrites ci-après se font à partir des distributions de fréquences alléliques dans chacune des populations. Le niveau de résolution du polymorphisme nous permet de discriminer les différentes spécificités génériques répertoriées pour le gène HLA-C (14 au total: Cw*01 à Cw*08, Cw*12, et Cw*14 à Cw*18). A noter que, pour un nombre restreint de populations, nous disposons d’un typage plus précis permettant de travailler au niveau allélique. Néanmoins, les résultats étant concordants pour ces deux niveaux de résolution, nous allons uniquement nous baser sur la résolution générique qui donne une meilleure représentativité géographique au niveau du nombre de populations comparées.
Nous ferons
parfois mention de la résolution allélique lorsqu’elle apporte une information
supplémentaire utile à l’interprétation des résultats.
La diversité génétique du gène HLA-C est étudiée à différents niveaux: entre populations, par l’indice de fixation FST, entre populations au sein des groupes géographiques, par l’indice FSC, et entre les groupes géographiques, par l’indice FCT. Ces divers indices sont testés par une procédure non-paramétrique de permutation de gènes entre populations, pour le FSC et le FST, ou permutation de populations entre groupes, pour le FCT, comme décrit dans le logiciel Arlequin (Schneider et al., 2000). La structure linguistique est testée selon le même schéma. Des distances génétiques de Reynolds (Reynolds et al., 1983), directement liées aux FSTs, sont calculées et utilisées pour représenter graphiquement, sur deux dimensions, les relations entre populations par la méthode d’analyse d’échelonnement multidimensionnel (ou MDS) (Kruskal, 1964). Des distances géographiques sont également calculées entre chaque paire de populations en se basant sur la longueur de l’arc à une sphère, et transformées en logarithmes naturels (Ray, 2003), afin de tester la corrélation entre génétique et géographie. Finalement, des frontières génétiques potentielles entre groupes de populations sont recherchées à l’aide de la méthode SAMOVA (Dupanloup et al., 2002).
Résultats
En étudiant la zone de locution du phylum indo-européen dans son ensemble, et comme le montre l’analyse d’échelonnement multidimensionnel (Figure 1), les populations européennes, proches-orientales et indo-pakistanaises sont clairement différenciées génétiquement les unes par rapport aux autres. Cette observation est corroborée par le faible pourcentage de FSTs non-significatifs recensé entre paires de populations appartenant à des régions différentes (1% entre l’Europe et le Proche-Orient, 1,8% entre l’Europe et l’Inde-Pakistan, 3.6% entre le Proche-Orient et l’Inde-Pakistan), et par la structure significative observée entre les trois grands groupes géographiques: l’indice FCT (diversité génétique entre groupes) est supérieur à l’indice FSC (diversité génétique entre populations à l’intérieur des groupes) sur l’ensemble des trois régions (FCT = 1.91%, FSC = 1.82%, Table 2). Une corrélation significative (rgen,géo) est aussi observée entre distances génétiques et géographiques si l’on considère l’ensemble des populations (Table 3).
Néanmoins, plusieurs chevauchements entre groupes sont décelables sur la MDS, et la corrélation entre distances génétiques et géographiques est très différente selon les régions (Table 3). En conséquence, les résultats ci-après sont présentés par région géographique (d’autant plus que nous n’observons aucune structure significative entre les grands groupes et familles linguistiques représentées dans cette étude).
En Europe, les différenciations génétiques sont majoritairement influencées par la géographie comme l’atteste la projection des populations sur la MDS et le coefficient de corrélation élevé mesuré entre ces deux variables (r = 0.562, Table 3). De plus, nous détectons une barrière génétique significative suivant un axe nord-est / sud-ouest qui longe approximativement les Alpes et coupe à travers les Pyrénées, séparant les populations du pourtour méditerranéen de celle d’Europe du nord-ouest (frontière 1, Figure 1). La valeur élevée de l’indice de fixation FST en Europe (FST = 1.98 %, Table 3) semble résulter de la forte différenciation génétique des populations du nord-ouest vis-à-vis des populations du sud-est. Le groupe du nord-ouest est en fait très homogène du point de vue génétique: le FST entre populations (Irlandais, Anglais, Finnois, Suisses) est seulement de 0.39% lorsque la diversité génétique est estimée avec une résolution de typage maximale (niveau allélique, résultat non montrés).
|
Analyses inter-régionales |
n |
FCT (%) |
FSC (%) |
|
Europe / Proche-Orient / Inde-Pakistan |
39 |
1.91 *** |
1.82 *** |
|
Europe versus Proche-Orient |
24 |
2.66 *** |
1.76 *** |
|
Europe versus Inde-Pakistan |
31 |
1.52 *** |
1.96 *** |
|
Proche-Orient versus Inde-Pakistan |
23 |
0.87 *** |
1.67 *** |
Tableau 2. Comparaisons des groupes de populations à l’échelle inter-régionale. ***: P<0.001, ns: non significatif, n: nombre de populations.
Table 2. Population comparisons between geographic groups. ***: P<0.001, ns: non significant, n: population number.
Les populations du Proche-Orient
forment un groupe génétiquement plus homogène (FST = 1.31 %, Table
3) et exhibent la plus forte proportion de distances génétiques deux à deux
non-significatives (21.4%, Table 3). Sur la MDS, ces populations sont projetées
entre l’Europe et le sous-continent indien, ce qui correspond à leur
localisation géographique intermédiaire à ce carrefour continental. Toutefois,
bien qu’élevé, le coefficient de corrélation entre distances génétiques et
géographiques n’est pas significatif (r=0.213, Table 3).
|
Analyses intra-régionales |
n |
FST par paire non significatifs (%) |
FST global (%) |
r |
|
Toutes les populations |
39 |
8.0 |
3.01 *** |
0.286 *** |
|
Europe |
16 |
10.8 |
1.98 *** |
0.562 *** |
|
Proche-Orient |
8 |
21.4 |
1.31 *** |
0.213 ns |
|
Inde-Pakistan |
15 |
17.1 |
2.02 *** |
0.09 ns |
Tableau 3. Comparaisons des populations à l’échelle
régionale. Le pourcentage de FSTs non significatifs entre paires de
populations nous indique la proportion des distances génétiques qui ne sont pas
significativement différentes de zéro au sein de chacune des régions
géographiques. Le FST
global représente quant à lui la diversité génétique estimée sur l’ensemble des
populations d’une région donnée.
La dernière colonne donne la valeur du coefficient de corrélation calculé
entre distances génétiques et
géographiques. ***: P<0.001, ns: non significatif, n: nombre de
populations.
Table 3. Population comparisons within geographic groups. The
percentage of non significant
pairwise FSTs gives us the proportion of genetic distances that do
not differ significantly from zero within each geographic area. Global FST
values are a measure of the genetic diversity among populations within a
group. Correlation coefficients between geographic and genetic distances are
given in the last column. ***: P<0.001, ns: non significant, n: population
number.
De plus, bien que l’analyse ANOVA confirme une différenciation
intergroupe significative (FCT = 2.66%,
compare à FSC = 1.76%, Table 2), ce groupe se superpose partiellement au
groupe européen, notamment les Turcs, qui sont génétiquement plus proches des
populations méditerranéennes d’Europe (Grecs, Baléares, Italiens et Catalans)
que des populations proche-orientales.
Au contraire de celles du Proche-Orient, les populations indo-pakistanaises
ne forment pas un groupe homogène. Les Nadars et les Parsi sont séparés du
groupe principal sur la MDS et sont projetés à proximité des Catalans et des
Italiens du Nord (Figure 1). Cette proximité est partiellement causée par le
type de représentation et le nombre de dimensions choisies, d’après des
analyses complémentaires intégrant une troisième dimension ou excluant le
groupe indo-pakistanais (résultats non montrés ici). En réalité, ces deux
populations divergent fortement des autres populations du sous-continent
indien. Ceci est particulièrement marqué pour les Nadars, locuteurs dravidiens
du Tamil-Nadu, isolés génétiquement malgré leur proximité géographique avec les
Tamils. Mais cette divergence se généralise à l’ensemble des locuteurs
dravidiens inclus dans ces analyses, comme le montrent leur emplacement
excentré sur la MDS (Figure 1) et leur hétérogénéité génétique (FST
= 2.4%), plus élevée que celle des populations européennes (FST =
1.98%, Table 3). Bien que les Dravidiens représentent un cas extrême de
diversité, les populations indo-pakistanaises sont tout particulièrement
hétérogènes les unes par rapport aux autres (FST=2.02%, Table 3; et
FSC > FCT lorsque l’on compare l’Europe ou le
Proche-Orient au groupe Inde-Pakistan, Table 2). En effet, une estimation de
leur diversité génétique dans un sous-ensemble de 9 populations typées à haute
résolution (niveau allélique) révèle un indice de fixation de 3.45% dans le
sous-continent indien, soit une valeur 1.5 fois plus élevée que celle obtenue
en considérant le niveau de résolution générique (résultats non montrés). Ce
résultat est causé principalement par le fait que les populations
indo-pakistanaises sont très différenciées pour divers allèles des mêmes
spécificités génériques (notamment Cw*03, Cw*07, Cw*12,
Cw*14, et Cw*16).
Finalement, la recherche de frontières génétiques nous suggère aussi une
grande hétérogénéité génétique au sein du groupe indo-pakistanais. En effet,
comme illustré dans la Figure 1, une barrière génétique significative (frontière
2) est décelée entre les populations du Baloutchistan (Baloutches et Brahui) et
les autres populations du sous-continent indien.
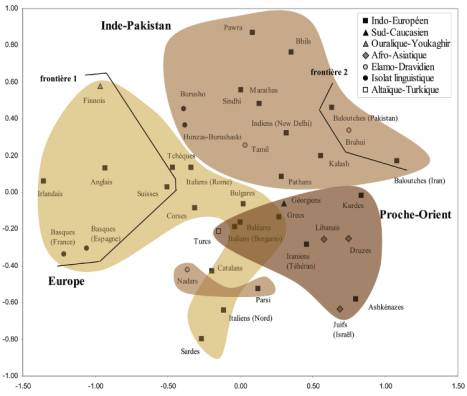
Figure
1. Analyse d’échelonnement
multidimensionnel (MDS) à partir des distances génétiques de Reynolds calculées
entre paires de populations. L’indice de stress (0.2018) indique une adéquation
moyenne de notre projection vis-à-vis des données d’après l’échelle qualitative
donnée pour cet indice (Rohlf, 2000). Une explication possible réside dans le
nombre important de populations projetées sur le graphique, mais aussi du fait
que de telles valeurs sont souvent observées lorsque le système génétique
étudié est très polymorphe comme ici. L’appartenance géographique et
linguistique des 39 populations y est indiquée, de même que les frontières
génétiques détectées par l’analyse SAMOVA. Frontière 1
(détectée en Europe): FCT(%) = 2.76, FSC(%) = 1.59;
Frontières 1&2 (détectées en Europe et en Inde-Pakistan): FCT(%)
= 2.75, FSC(%) = 1.43.
Tous les indices sont significatifs (P<0.001).
Figure 1. Multidimensional scaling analysis (MDS) based on
Reynolds genetic distances calculated between pairs of populations. The stress
index (0.2018) indicates an average goodness-of-fit of the 2-dimensional
projection to the original data (Rohlf, 2000). Geographic and linguistic
affiliations are ploted, as well as genetic boundaries detected by the SAMOVA
analysis. Frontier 1 (detected in Europe): FCT(%) = 2.76, FSC(%) = 1.59;
Frontiers 1&2 (detected in Europe and in India-Pakistan): FCT(%)
= 2.75, FSC(%) = 1.43.
All indexes are significant (P<0.001).
Discussion
et conclusion
Les analyses réalisées sur un ensemble de 39 populations localisées entre l’Europe et la région Inde-Pakistan montrent une corrélation significative entre distances géographiques et génétiques lorsque l’ensemble des populations est considéré. Ce n’est toutefois plus le cas à l’échelle régionale, où des schémas bien plus complexes de différenciation génétique semblent être en vigueur. Contrairement à de nombreuses études portant sur le système HLA et d’autres marqueurs qui observent une correspondance significative entre génétique et linguistique pour plusieurs grands phylums (Chen et al., 1995; Poloni et al., 2005; Sanchez-Mazas et al., 2005), dont l’Indo-Européen (Sokal et al., 1992), la linguistique ne semble pas être un facteur prépondérant dans les différenciations génétiques des populations au locus HLA-Cw.
En Europe, et à l’instar de ce que nous venons de mentionner, la géographie a une influence prépondérante sur les profils génétiques observés pour le gène HLA-Cw. Ce résultat est en concordance avec des observations obtenues sur le chromosome Y et l’ADN mitochondrial (Rosser et al., 2000; Simoni et al., 2000). Nous remarquons également une forte différenciation des populations du nord-ouest de celles du pourtour méditerranéen, mis en évidence par la détection d’une frontière génétique séparant ces deux groupes. Celle-ci semble correspondre à une barrière géographique du fait qu’elle traverse la chaîne des Alpes et les Pyrénées. Néanmoins, nous ne pouvons exclure ici l’effet d’une barrière culturelle entre les populations de l’ouest de l’Europe et celles du bassin méditerranéen. En effet, la majeure partie des populations du bassin méditerranéen sont des locuteurs balto-slaviques et grecs, deux groupes clairement différenciés des populations d’Europe de l’ouest lorsque le polymorphisme du gène HLA-DRB1 est étudié (Sanchez-Mazas, 2-4 Oct 2004 (sous presse); Abdennaji Guenounou et al., soumis). Ces mêmes études montrent toutefois que les locuteurs italiques du sud-est (Italiens) sont plus proches des Balto-Slaviques et Grecs que des Italiques du sud-ouest (Espagnols et Français), suggérant plutôt l’influence d’une barrière géographique au niveau des Alpes. Une telle hypothèse a également été proposée dans une étude s’intéressant à la diversité génétique du chromosome Y en Europe (Rosser et al., 2000), qui détectait une série de barrières génétiques allant de l’Italie à la mer Baltique.
Le Proche-Orient constitue un groupe génétiquement plus homogène au sein duquel les différenciations ne semblent pas avoir été très fortement influencées par la géographie. Cela pourrait s’expliquer par les nombreux et importants mouvements de populations dont cette région, située à un carrefour continental entre l’Europe, l’Afrique et l’Asie, a été le théâtre à travers l’histoire. De plus, nous ne détectons aucune frontière génétique entre les populations européennes du sud-est et les populations proches-orientales. La Méditerranée semble donc avoir constitué une zone d’échanges génétiques et non une barrière géographique. Des résultats semblables ont été obtenus pour le polymorphisme HLA-DRB1 (Sanchez-Mazas, 2-4 Oct 2004 (sous presse); Abdennaji Guenounou et al., soumis), alors que des études menées sur le chromosome Y et différents marqueurs classiques (Simoni et al., 1999; Rosser et al., 2000) suggèrent que la Méditerranée a constitué une frontière génétique importante. Ces discordances pourraient refléter des pressions sélectives différentes agissant sur le polymorphisme de ces systèmes génétiques, mais aussi être la conséquence de modes de transmission génétique distincts, autosomique pour les gènes HLA, et sexe-spécifique pour le chromosome Y, impliquant diverses histoires évolutives.
Concernant le sous-continent indien, nous ne détectons pas de corrélation significative entre distances géographiques et génétiques. L’extrême diversité génétique observée dans cette région nous porte à croire que la majorité des populations ont subi une dérive génétique rapide sous l’effet complexe de barrières culturelles, sociales, linguistiques et religieuses. Notamment, les populations dravidiennes sont très divergentes les unes des autres d’après leurs profils génétiques malgré la proximité géographique de certaines d’entre elles. Les Parsi, une communauté immigrante en provenance d’Iran, isolée de par sa religion (zoroastrisme) et le système de castes prévalant en Inde, est également très divergente génétiquement. Le système de castes divise d’ailleurs les Indiens et Pakistanais en une multitude de sous-groupes et régit strictement le choix des conjoints et les tâches quotidiennes. Il a sans doute eu une forte influence sur les différenciations génétiques de ces populations (Cordaux et al., 2004). Plusieurs études portant sur la diversité du chromosome Y et de l’ADN mitochondrial proposent également une forte dérive génétique au sein des populations indo-pakistanaises (Cordaux et al., 2003; Vishwanathan et al., 2004), ainsi qu’une forte influence socioculturelle sur leurs profils génétiques (Bamshad et al., 1998; Bhattacharyya et al., 1999; Das et al., 2004), ce qui s’accorde avec nos résultats.
Pour conclure, cette étude confirme que le gène HLA-Cw est un marqueur très informatif pour étudier l’histoire du peuplement humain, non seulement à l’échelle mondiale, comme cela a déjà été proposé (Buhler et al., 2002; Sanchez-Mazas, sous presse), mais aussi à un niveau régional.
Bibliographie
Abdennaji Guenounou, B., Yacoubi Loueslati, B., Buhler, S., Hmida, S.,
Ennafaa, H., Khodjet-Elkhil, H., Moojat, N., Dridi, A., Boukef, K., Ben Ammar
Elgaaied, A. et Sanchez-Mazas, A., soumis, HLA class II genetic diversity in
Southern Tunisia and the Mediterranean area.
Bamshad, M. J., Watkins, W. S.,
Dixon, M. E., Jorde, L. B., Rao, B. B., Naidu, J. M., Prasad, B. V.,
Rasanayagam, A. et Hammer, M. F., 1998, Female gene flow stratifies Hindu
castes. Nature, 395, 651-2.
Bhattacharyya, N. P., Basu, P., Das,
M., Pramanik, S., Banerjee, R., Roy, B., Roychoudhury, S. et Majumder, P. P.,
1999, Negligible male gene flow across ethnic boundaries in India, revealed by
analysis of Y-chromosomal DNA polymorphisms. Genome Res, 9, 711-9.
Buhler, S., 2001, Etude moléculaire
du polymorphisme HLA-Cw en Iran. Contributions à l'analyse de la variabilité
génétique humaine, Diploma thesis, University of Geneva, Switzerland, pp. 186.
Buhler, S., Sanchez-Mazas, A.,
Zanone, R., Djavad, N. et Tiercy, J. M., 2002, PCR-SSOP molecular typing of
HLA-C alleles in an Iranian population. Tissue Antigens, 59, 525-30.
Buhler, S., Mégarbané, A., Lefranc,
G., Tiercy, J.-M. et Sanchez-Mazas, A., soumis, The HLA-C molecular
characterization of a Lebanese population and an analysis of HLA-C genetic
structure of 39 populations from Europe to India-Pakistan.
Bunce, M., Barnardo, M., Procter,
J., Marsh, S., Vilches, C. et Welsh, K., 1997a, High resolution HLA-C typing by
PCR-SSP: identification of allelic frequencies and linkage disequilibria in 604
unrelated random UK Caucasoïds and a comparison with serology. Tissue Antigens,
50, 100-111.
Bunce, M., Young, N. et Welsh, K.,
1997b, Molecular HLA typing-the brave new world. Transplantation, 64,
1505-1513.
Chen, J., Sokal, R. R. et Ruhlen,
M., 1995, Worldwide analysis of genetic and linguistic relationships of human
populations. Hum Biol, 67, 595-612.
Comas, D., Mateu, E., Calafell, F.,
Pérez-Lezaun, A., Bosch, E., Martínez-Arias, R. et Bertranpetit, J., 1998, HLA
class I and class II DNA typing and the origin of Basques. Tissue Antigens, 51,
30-40.
Cordaux, R., Saha, N., Bentley, G.
R., Aunger, R., Sirajuddin, S. M. et Stoneking, M., 2003, Mitochondrial DNA
analysis reveals diverse histories of tribal populations from India. Eur J Hum
Genet, 11, 253-64.
Cordaux, R., Aunger, R., Bentley,
G., Nasidze, I., Sirajuddin, S. M. et Stoneking, M., 2004, Independent origins
of Indian caste and tribal paternal lineages. Curr Biol, 14, 231-5.
Cox, S., Marsh, S., Scott, I.,
Clayton, J., Argüello, J., McWhinnie, A., Prokupek, B., Holman, R., Madrigal,
J. et Little, A., 1999, HLA-A, -B, -C polymorphism in a UK Ashkenazi Jewish
potential bone marrow donor population. Tissue Antigens, 53, 41-50.
Dard, P., 1991, Analyse sérologique
du polymorphisme HLA de classe I de la population Mandenka du Sénégal oriental
et intégration des données dans une étude comparative de la variabilité
génétique humaine, Diploma thesis, University of Geneva, Switzerland, pp. 70.
Das, B., Chauhan, P. S. et Seshadri,
M., 2004, Minimal sharing of Y-chromosome STR haplotypes among five endogamous
population groups from western and southwestern India. Hum Biol, 76, 743-63.
Dupanloup, I., Schneider, S. et
Excoffier, L., 2002, A simulated annealing approach to define the genetic
structure of populations. Mol Ecol, 11, 2571-81.
Farjadian, S., Naruse, T., Kawata,
H., Ghaderi, A., Bahram, S. et Inoko, H., 2004, Molecular analysis of HLA
allele frequencies and haplotypes in Baloch of Iran compared with related
populations of Pakistan. Tissue Antigens, 64, 581-7.
Ferrara, G., Delfino, L., Longo, A.,
Morabito, A. et Parodi, A., 1998a, Northern Italian (Bergamo) normal. Dans HLA
1998, édité par P. Terasaki and D. Gjertson (Lenexa: American Society for
Histocompatibility and Immunogenetics) p.168.
Ferrara, G., Delfino, L., Longo, A.,
Morabito, A. et Parodi, A., 1998b, Caucasian Italian normal. Dans HLA 1998,
édité par P. Terasaki and D. Gjertson (Lenexa: American Society for
Histocompatibility and Immunogenetics) p.166-167.
Ferrara, G., Delfino, L., Longo, A.,
Morabito, A. et Parodi, A., 2004a, HLA-A, -B, -Cw, -DPB1, -DQA1, -DQB1 and
-DRB1 Allele Frequencies in a Population from Bergamo, Italy. Hum Immunol, 65,
974-980.
Ferrara, G., Delfino, L., Longo, A.,
Morabito, A. et Parodi, A., 2004b, HLA-A, -B, -Cw, -DPB1, -DQA1, -DQB1 and
-DRB1 Alleles in a Population from North Italy. Hum Immunol, 65, 982-987.
Grimaldi, M. C., Crouau-Roy, B.,
Amoros, J. P., Cambon-Thomsen, A., Carcassi, C., Orru, S., Viader, C. et Contu,
L., 2001, West Mediterranean islands (Corsica, Balearic islands, Sardinia) and
the Basque population: contribution of HLA class I molecular markers to their
evolutionary history. Tissue Antigens, 58, 281-92.
Ivanova, M., Spassova, P.,
Michailova, A. et Naumova, E., 2001, Distributions of HLA class I alleles and
haplotypes in Bulgarians--contribution to understanding the origin of the
population. Tissue Antigens, 57, 208-15.
Kennedy, L., Poulton, K., Dyer, P.,
Ollier, W. et Thomson, W., 1995, Definition of HLA-C alleles using
sequence-specific oligonucleotide probes (PCR-SSOP). Tissue Antigens, 46, 187-195.
Kruskal, J., 1964, Multidimensional
scaling by optimizing goodness of fit to a nonmetric hypothesis. Psychometrika,
29, 1-27.
Mack, S., Tsai, Y., Sanchez-Mazas,
A. et Erlich, H., sous presse, Anthropology / human genetic diversity
population reports. Dans HLA 2004: Immunobiology of the Human MHC. Proceedings
of the 13th International Histocompatibility Workshop and Congress, édité par
J. Hansen and B. Dupont (IHWG Press).
Mohyuddin, A., Ayub, Q., Khaliq, S.,
Mansoor, A., Mazhar, K., Rehman, S. et Mehdi, S. Q., 2002, HLA polymorphism in
six ethnic groups from Pakistan. Tissue Antigens, 59, 492-501.
Mytilineos, J., Christ, U., Lempert,
M. et Opelz, G., 1997, Comparison of typing results by serology and polymerase
chain reaction with sequence-specific primers for HLA-Cw in 650 individuals.
Tissue Antigens, 50, 395-400.
Papassavas, E., Spyropoulou-Vlachou,
M., Papassavas, A., Schipper, R., Doxiadis, I. et Stavropoulos-Giokas, C.,
2000, MHC Class I and Class II Phenotype, Gene, and Haplotype Frequencies in Greeks
Using Molecular Typing Data. Hum Immunol, 61, 615-623.
Piazza, A. et Lonjou, C., 1997, HLA
in Europe and in the Mediterranean contries. Dans Proc 12th Int
Histocompatibility Workshop and Conference, édité par D. Charron (Paris:EDK)
p.374-384.
Poloni, E., Sanchez-Mazas, A.,
Guillaume, J. et Sagart, L., 2005, Comparing linguistic and genetic
relationships among East Asian populations: A study of the RH and GM
polymorphisms. Dans The Peopling of East Asia: Putting together archeology,
linguistics and genetics, édité par L. Sagart, R. Blench and A. Sanchez-Mazas
(London and NY: RoutledgeCurzon).
Qasim Medhi, S., Mohyuddin, A.,
Qamar, R. et Khaliq, S., 1997, INPA (India and Pakistan) regional report: HLA
Class I polymorphism in different Pakistani ethnic groups. Dans Proc 12th Int
Histocompatibility Workshop and Conference, édité par D. Charron (Paris:EDK)
p.321-322.
Ray, N., 2003 communication
personnelle.
Reynolds, J., Weir, B. et Cockerham,
C., 1983, Estimation for the coancestry coefficient: basis for a short-term
genetic distance. Genetics, 105, 767-779.
Rosser, Z. H., Zerjal, T., Hurles,
M. E., Adojaan, M., Alavantic, D., Amorim, A., Amos, W., Armenteros, M.,
Arroyo, E., Barbujani, G., Beckman, G., Beckman, L., Bertranpetit, J., Bosch,
E., Bradley, D. G., Brede, G., Cooper, G., Corte-Real, H. B., de Knijff, P.,
Decorte, R., Dubrova, Y. E., Evgrafov, O., Gilissen, A., Glisic, S., Golge, M.,
Hill, E. W., Jeziorowska, A., Kalaydjieva, L., Kayser, M., Kivisild, T.,
Kravchenko, S. A., Krumina, A., Kucinskas, V., Lavinha, J., Livshits, L. A.,
Malaspina, P., Maria, S., McElreavey, K., Meitinger, T. A., Mikelsaar, A. V.,
Mitchell, R. J., Nafa, K., Nicholson, J., Norby, S., Pandya, A., Parik, J.,
Patsalis, P. C., Pereira, L., Peterlin, B., Pielberg, G., Prata, M. J.,
Previdere, C. et Roewer, L., 2000, Y-chromosomal diversity in Europe is clinal
and influenced primarily by geography, rather than by language. Am J Hum Genet,
67, 1526-43.
Sanchez-Mazas, A., 2-4 Oct 2004
(sous presse), Genetic diversity around the Mediterranean according to GM and
HLA molecular markers. Dans Proceedings of the international symposium:
"Le Peuplement de la Méditerranée: synthèse et question d'avenir",
édité par (Bibliotheca Alexandrina).
Sanchez-Mazas, A., Steiner, Q.-G.,
Grundschober, C. et Tiercy, J.-M., 2000, The molecular determination of HLA-Cw
alleles in the Mandenka (West Africa) reveals a close genetic relationship
between Africans and Europeans. Tissue Antigens, 56, 303-12.
Sanchez-Mazas, A., Poloni, E.,
Guillaume, J. et Sagart, L., 2005, HLA diversity and linguistic variation in
East Asia. Dans The Peopling of East Asia: Putting together archeology,
linguistics and genetics, édité par L. Sagart, R. Blench and A. Sanchez-Mazas
(London and NY: RoutledgeCurzon).
Sanchez-Mazas, A., sous presse, HLA
genetic diversity of the 13th IHWC population data relative to worldwide
linguistic families. Dans HLA 2004: Immunobiology of the Human MHC. Proceedings
of the 13th International Histocompatibility Workshop and Congress, édité par
J. Hansen and B. Dupont (IHWG Press)
Shankarkumar, U., Ghosh, K., Gupte,
S., Mukerjee, M. et Mohanty, D., 1999, Distribution of HLA antigens in Bhils
and Pawars of Dhadgaon, Maharashtra, India. J. Human Ecology, 10, 173-178.
Shankarkumar, U., Ghosh, K. et Mohanty,
D., 2001, HLA antigen distribution in Maratha community from Mumbai. Inter. J.
Human Genetics, 1, 173-177.
Shankarkumar, U., 2002, HLA A19 and
B14 frequencies among Parsis from Mumbai. Dans MHC, édité par p.71-77.
Shankarkumar, U., Sridharan, B. et
Pitchappan, R. M., 2003, HLA diversity among Nadars, a primitive Dravidian
caste of South India. Tissue Antigens, 62, 542-7.
Shankarkumar, U., 2004a, HLA-A, -B
and -Cw Allele Frequencies in a Pawra Population from Khandesh, India. Hum
Immunol, 65, 932-933.
Shankarkumar, U., 2004b, HLA-A, -B
and -Cw Allele Frequencies in a Maratha Population from Mumbai, India. Hum
Immunol, 65, 954-956.
Shankarkumar, U., 2004c, HLA-A, -B
and -Cw Allele Frequencies in a Bhil Population from Western India. Hum
Immunol, 65, 960-961.
Shankarkumar, U., 2004d, HLA-A, -B
and -Cw Allele Frequencies in a Parsi Population from Western India. Hum
Immunol, 65, 992-993.
Simoni, L., Gueresi, P., Pettener,
D. et Barbujani, G., 1999, Patterns of gene flow inferred from genetic
distances in the Mediterranean region. Hum Biol, 71, 399-415.
Simoni, L., Calafell, F., Pettener,
D., Bertranpetit, J. et Barbujani, G., 2000, Geographic patterns of mtDNA
diversity in Europe. Am J Hum Genet, 66, 262-78.
Sokal, R. R., Oden, N. L. et
Thomson, B. A., 1992, Origins of the Indo-Europeans: genetic evidence. Proc
Natl Acad Sci U S A, 89, 7669-73.
Testi, M. et Mariani, M., 1998, Dans
HLA 1998, édité par P. Terasaki and D. Gjertson (Lenexa: American Society for
Histocompatibility and Immunogenetics)
Testi, M. et Mariani, M., 2004,
HLA-A, -B, -Cw, -DPB1, -DQA1, -DQB1 and -DRB1 Allele Frequencies in a
Population from Rome, Italy. Hum Immunol, 65, 987-991.
Turner, S., Ellexson, M., Hickman,
H., Sidebottom, D., Fernández-Viña, M., Confer, D. et Hildebrand, W., 1998, Sequence-Based
Typing Provides a New Look at HLA-C Diversity. The Journal of Immunology, 161,
1406-1413.
Uyar, F. A., Dorak, M. T. et
Saruhan-Direskeneli, G., 2004, Human leukocyte antigen-A, -B and -C alleles and
human leukocyte antigen haplotypes in Turkey: relationship to other
populations. Tissue Antigens, 64, 180-7.
Vishwanathan, H., Deepa, E.,
Cordaux, R., Stoneking, M., Usha Rani, M. V. et Majumder, P. P., 2004, Genetic
structure and affinities among tribal populations of southern India: a study of
24 autosomal DNA markers. Ann Hum Genet, 68, 128-38.
Williams, F. et Middleton, D., 2004,
HLA-A, -B, -Cw and -DRB1 Alleles, KIR Alleles and Cytokine Polymorphisms in a
Population from Northern Ireland. Hum Immunol, 65, 964-971.
Zahlavova, L., Bendukidze, N. et
Ivaskova, E., 2004, HLA-A, -B, -Cw, -DPB1, -DQA1, -DQB1 and -DRB1 Allele
Frequencies in a Population from the Czech Republic. Hum Immunol, 65, 925-930.