Colloques
du Groupement des Anthropologistes de Langue Française (GALF)
Coudray, C., Guitard, E., Gibert, M., Sevin, A., Larrouy, G., Dugoujon, J.M., 2006, Diversité génétique (allotypie GM et STRs) des populations Berbères et peuplement du nord de l’Afrique. Antropo, 11, 75-84. www.didac.ehu.es/antropo
Diversité génétique (allotypie GM et STRs) des populations Berbères et peuplement du nord de l’Afrique
Genetic diversity (GM
allotypes and STRs) of the Berber populations and the peopling of Northern Africa
Clotilde Coudray, Evelyne Guitard, Morgane Gibert, André Sevin, Georges Larrouy, Jean-Michel Dugoujon
Centre d’Anthropologie UMR 8555 CNRS, 37, allées Jules Guesde, 31073 Toulouse Cedex 4, France. E-mail : clotilde.coudray@wanadoo.fr
Mots Clés: Berbères, peuplement nord-africain, marqueurs génétiques, allotypes GM, STRs.
Key
Words: Berbers,
North-African settlement, genetic polymorphisms, GM allotypes, STRs.
Résumé
L’origine des populations berbères n’est pas encore clairement établie mais on considère aujourd’hui leurs ancêtres comme les plus anciens habitants du nord de l’Afrique, probablement depuis le Paléolithique supérieur ou le Néolithique. Ces hommes ont ensuite connu un passé historique marqué par de nombreuses invasions et conquêtes qui ont induit de profonds changements sur les autochtones nord-africains. Ces impacts, à la fois culturels et génétiques, peuvent être appréhendés par l’étude des populations berbères actuelles permettant ainsi d’apporter des éléments de réponse sur la question du peuplement du nord de l’Afrique.
Dans le cadre d’un projet EUROCORES « Origin of Man, Language and Languages » (OMLL), le présent travail décrit la structure génétique de différentes populations berbérophones à l’aide de 2 polymorphismes autosomaux : le système allotypique GM des immunoglobulines et 13 microsatellites ou Short Tandem Repeats (STR). Notre objectif est de décrire les relations génétiques et culturelles entre les populations berbérophones et de retracer les degrés de parenté entre celles-ci et des populations voisines de culture et de langue différentes.
Nos analyses révèlent une concordance entre les résultats obtenus d’après l’étude du système GM et ceux apportés par les polymorphismes STR. Nous constatons ainsi (1) une nette différenciation génétique entre les populations berbères nord-ouest et nord-est africains ; (2) une proximité entre les Berbères nord-ouest africains et les populations sud-ouest européennes ; (3) une absence de distinction génétique entre arabophones et berbérophones marocains.
Il apparaît ainsi que les Berbères actuels portent les traces d’un peuplement nord-africain ancien qui aurait réussi à préserver dans les générations suivantes ses originalités culturelles (coutumes et langage), malgré les impacts des évènements historiques.
Abstract
The origin of the Berber populations is not clearly established today
but their ancestors are considered as the oldest inhabitants of North Africa
since the Upper Palaeolithic or the Neolithic.
Then, the historical period is marked by successive invasions and
conquests that caused important changes on North-African autochthonous people.
By studying present-day Berber populations it may be possible to describe both
cultural and religious changes, in the context of the North-African settlement.
Within the EUROCORES framework "The Origin of Man, Language and
Languages" (OMLL), we describe here the genetic structure of several
Berber-speakers. We study 2 genetic polymorphisms: the GM immunoglobulin
allotypic system and 13 autosomal microsatellites (STR). The aim of the work is
to characterize both the cultural and genetic relationships between
North-African Berbers and between the Berbers and geographically related
populations.
The analyses reveal the correspondence between GM and STR results. We
see (1) a clear genetic differentiation between North-West and North-East
African Berbers; (2) a relationship between North-West African Berbers and
South-West European populations; (3) in Morocco, a genetic distinction between
Arabic- and Berber-speakers.
Finally, it seems that the present-day Berbers bear some old traces of
the North-African settlement. Moreover, they have preserved their cultural
identity in spite of the successive past events.
Introduction
En Afrique du Nord, les grandes questions de l’histoire du peuplement passent nécessairement par l’étude des populations berbères. En effet, leurs ancêtres sont considérés comme les plus anciens habitants du nord de l’Afrique. Ils seraient les descendants d’individus Capsiens ayant vécu au Mésolithique autour de 10 000 à 4 700 ans BP (Desanges, 1990). Ces Capsiens auraient ensuite persisté jusqu’au Néolithique (à partir de –5 000 ans av. J.-C.) et auraient introduit ou diffusé au cours de leur sédentarisation les premiers langages Afro-Asiatiques (dont la question du foyer linguistique originel est encore débattue) (Renfrew et al., 1991 ; Cavalli-Sforza et al., 1994 ; Ben Hamed et Darlu, 2003). Ces anciens Berbères ont par la suite connu un passé historique très mouvementé avec de nombreuses conquêtes, invasions et tentatives d’assimilation. La plus récente et importante d’entre elles est la conquête arabe amorcée au VIIème siècle qui a conduit à de profonds changements culturels, linguistiques et religieux. Le refus d’islamisation et d’arabisation de certains groupes berbères a contraint ces derniers à se retirer dans des régions isolées. Au milieu d’un ensemble de populations majoritairement arabophones, on compte aujourd’hui sur le continent africain plus de 20 millions de berbérophones répartis en petites communautés disséminées de la Mauritanie à l’Egypte (oasis de Siouah) et du désert du Sahara aux régions montagneuses de l’Atlas marocain. La diversité linguistique des Berbères est grande. Au sein du phylum Afro-Asiatique (Hayward, 2000), la famille linguistique du berbère regroupe plus d’une trentaine de langages. L’arabe appartient à la famille linguistique sémitique de ce même phylum Afro-Asiatique.
Nos travaux s’inscrivent dans le cadre d’un projet EUROCORES « Origin of Man, Language and Languages » (OMLL) ayant pour but de décrire la diversité génétique et linguistique des Berbères et de retracer leurs degrés de parenté avec des populations géographiquement proches, de culture et de langue différentes.
La caractérisation anthropogénétique des populations berbères a été récemment établie grâce à différents marqueurs : les allotypes des immunoglobulines (Dugoujon et al., 2004; Coudray et al., 2004 ; Coudray et al., sous presse), l’ADN mitochondrial (Fadhlaoui-Zid et al., 2004), le chromosome Y (Arredi et al., 2004), les microsatellites autosomaux (Bosch et al., 2000), les séquences Alu (Gonzalez-Pérez et al., 2003). Dans le but d’actualiser la base de données de notre projet OMLL, la présente analyse décrit la structure génétique des populations berbérophones à l’aide de deux polymorphismes génétiques : les allotypes des immunoglobulines et 13 microsatellites autosomaux.
Le polymorphisme allotypique des immunoglobulines a été défini par Oudin en 1956 (présence de déterminants antigéniques sur les chaînes lourdes et légères des immunoglobulines chez certains individus d’une même espèce). On décrit à ce jour 4 systèmes allotypiques selon la classe d’immunoglobuline considérée mais, en Génétique des Populations, seul le système GM le plus polymorphe (et donc le plus informatif) est analysé. Ce système est porté par 3 des 4 sous-classes d’IgG (IgG1, IgG2 et IgG3) et se compose de 18 allotypes (Lefranc et Lefranc, 1990). Les allotypes GM sont codés par des gènes proches les uns des autres sur le chromosome 14 et dont les allèles sont transmis dans des combinaisons fixes appelées haplotypes. On reconnaît aujourd’hui 15 haplotypes GM communs dont la distribution qualitative et quantitative varie fortement d’une population à l’autre, permettant de définir trois grands groupes : les haplotypes GM fréquemment rencontrés dans les populations européennes, ceux fréquents en Asie et ceux communs aux groupes sub-sahariens.
Les microsatellites ou Short Tandem Repeats (STR) sont de courtes séquences d’ADN répétées en tandem (100 à 400 pb) et dispersées dans la partie non codante du génome humain. Ils sont très polymorphes avec un polymorphisme caractérisé par la variation du nombre de motifs répétés. Notre travail porte sur 13 STRs autosomaux situés sur différents chromosomes : D8S1179, D21S11, D7S820, CSF1PO, D3S1338, TH01, D13S317, D16S539, D2S1338, D19S433, VWA, TPOX, D18S51, D5S818 et FGA.
Grâce à l’étude de ces différents polymorphismes génétiques, notre but est de décrire les relations génétiques et culturelles entre les groupes berbères et de retracer les degrés de parenté entre ces Berbères et les populations voisines de culture et de langue différentes. Les résultats obtenus permettront de discuter les points suivants : les populations berbères sont-elles homogènes, ou existe-t-il des variations entre les groupes Nord-Ouest et Nord-Est africains ? La distinction linguistique entre les populations arabophones et berbérophones se superpose-t-elle à une différenciation génétique ? Quels impacts génétiques et culturels ont eu les populations migrantes (en particulier lors de la conquête arabe amorcée au VIIème siècle) sur les populations autochtones nord-africaines ?
Populations et Méthodes
Les données génétiques (distributions des fréquences haplotypiques et alléliques) figurent dans les différentes publications citées en bibliographie ou sont disponibles auprès de J.M. Dugoujon (dugoujon@cict.fr).
Les populations berbérophones ont été sélectionnées à partir d’une enquête linguistique réalisée sur le terrain. Les individus sont des adultes sains, non apparentés et dont les 4 grands-parents sont nés dans la même région.
Pour le système GM, les analyses portent sur les allotypes G1M(1,2,3,17), G2M(23) et G3M(5,6,10,11,13,14,15,16,21,24,28) mis en évidence selon la méthode classique de l’inhibition de l’hémagglutination (Field et Dugoujon, 1989). Les fréquences haplotypiques sont calculées à partir des phénotypes observés, selon la méthode du maximum de vraisemblance (Edwards, 1992).
Les analyses des fréquences alléliques STRs portent sur 13 microsatellites autosomaux : D8S1179, D21S11, D7S820, CSF1PO, D3S1338, TH01, D13S317, D16S539, D2S1338, D19S433, VWA, TPOX, D18S51, D5S818 et FGA. Les amplifications ADN sont réalisées par PCR multiplex et la séparation des fragments fluorescents est faite par électrophorèse capillaire dans un analyseur génétique. Les fréquences alléliques de chaque microsatellite sont calculées d’après les profils électrophorétiques observés.
Pour les comparaisons entre populations, nous avons réalisé différentes Analyses en Composantes Principales (ACP) (Ward, 1963). Ces analyses sont basées sur la projection graphique d’un dendrogramme construit à partir de la variabilité des fréquences haplotypiques (ou alléliques) rencontrées dans l’ensemble des populations considérées.
Résultats et Discussion
Nous avons tout d’abord considéré les populations nord-africaines berbérophones et arabophones (Fig. 1 et 2).
L’analyse comparative des populations berbères pour le système GM révèle une nette différenciation entre la population berbère de l’oasis égyptienne de Siouah et toutes les autres populations berbérophones ou arabophones du Nord-Ouest de l’Afrique (Fig. 1). Cette distinction entre Siouah et les autres populations berbères traduit la différenciation entre les haplotypes GM fréquemment rencontrés dans les populations sub-sahariennes (présents à plus de 50% à Siouah) et les haplotypes GM fréquemment rencontrés dans les populations ouest européennes (présents autour de 80% dans les groupes nord-ouest africains). L’analyse comparative des populations berbères portant sur les microsatellites autosomaux révèle aussi cette différenciation entre les Berbères de Siouah et les autres populations du Maroc, de Tunisie et même d’Egypte (Fig. 2).
Les ACP des figures 3 et 4 concernent l’ensemble des populations composant les bases de données GM et STR.
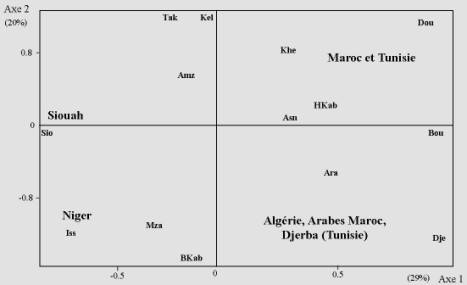
Figure 1. Analyse en Composantes Principales : les Berbères et le système GM.
Références: Ara, Amz, Asn, Khe et Bou : Maroc
(Dugoujon et al., sous presse ; Coudray et al., sous presse) ; BKab,
HKab, Mza et Kel : Algérie (Dugoujon et al., 2004 ; Dugoujon,
résultats non publiés ; Lefèvre-Witier, 1982) ; Tak, Dou et
Dje : Tunisie (Chaabani et al., 1984 ; Loveslati et al., 2001) ;
Iss : Niger (Lefèvre-Witier, 1982) ; Sio : Egypte (Dugoujon,
résultats non publiés).
Figure 1. Principal
Coordinate Analysis: the Berbers and the GM system.
References: Ara, Amz, Asn, Khe and Bou: Morocco
(Dugoujon et al., In press; Coudray et al., In press); BKab, HKab, Mza and Kel:
Algeria (Dugoujon et al., 2004; Dugoujon, unpublished results; Lefèvre-Witier,
1982); Tak, Dou, Dje: Tunisia (Chaabani et al., 1984; Loveslati et al., 2001);
Iss: Niger (Lefèvre-Witier, 1982); Sio: Egypt (Dugoujon, unpublished results).
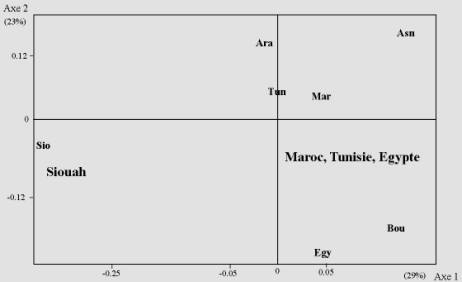
Figure 2. Analyse en Composantes Principales : les Berbères et les STR.
Références: Ara, Asn, Bou, Mar : Maroc (Bosch et
al., 2001 ; Perez-Lezaun et al., 2000 ; Coudray et al.,
soumis) ; Tun : Tunisie (Cherni et al., 2005 ; Brandt-Casadevall
et al., 2003) ; Egy, Sio : Egypte (Tahir et al., 2003 ;
Dugoujon, résultats non publiés).
Figure 2. Principal
Coordinate Analysis: the Berbers and the STR polymorphisms.
References: Ara, Asn, Bou, Mar: Morocco (Bosch et al., 2001; Perez-Lezaun et al., 2000; Coudray et al., submitted); Tun: Tunisia (Cherni et al., 2005; Brandt-Casadevall et al., 2003); Egy, Sio: Egypt (Tahir et al., 2003; Dugoujon, unpublished results).
L’analyse portant sur les 39 populations de la base de données GM permet de mettre en évidence 3 groupes de populations : (1) un groupe rapprochant les populations européennes et Berbères, caractérisé par des haplotypes GM communément rencontrés dans les populations européennes ; (2) un groupe constitué de toutes les populations sub-sahariennes et de la population berbère de Siouah, caractérisé par des haplotypes GM fréquemment rencontrés dans les groups sub-sahariens ; (3) un dernier groupe composé des 2 populations d’Afrique de l’Est et caractérisé par des haplotypes GM ayant une fréquence élevée dans les populations du Proche-Orient. La dernière analyse concernant les 18 populations de la base de données STR met aussi en évidence le rapprochement des populations européennes, berbérophones et arabophones, formant un groupe génétiquement différencié de celui des populations sub-sahariennes.
Les résultats font donc apparaître une nette correspondance entre les analyses de l’allotypie GM et des STRs. En effet, on constate, qu’à propos des relations de parenté entre les groupes berbères, il existe une nette différenciation génétique entre les populations du Nord-Ouest de l’Afrique et les Berbères d’Egypte. Ces derniers, situés dans la partie la plus orientale de l’aire de répartition des Berbères, auraient-ils une histoire génétique différente de celle des groupes nord-ouest africains ? Les découvertes archéologiques attestent l’occupation de l’oasis de Siouah au Paléolithique supérieur autour de 6700 - 8800 ans av J.-C. (Hassan, 1978 ; Fakhry, 1973) avec une industrie lithique particulière dite « libyco-capsienne ». Celle-ci se distingue nettement des industries lithiques contemporaines présentes au Maghreb et tendrait plutôt à se rapprocher de la culture Natoufienne du Proche-Orient. D’un point de vue linguistique, le Siwi est bien un dialecte berbère ayant une proche parenté avec les parlers berbères Libyens. D’un point de vue génétique, différentes analyses : système GM, chromosome Y, ADN mitochondrial (Dugoujon, résultats non publiés) s’accordent sur le fait que la population berbère de Siouah a une structure génétique particulière avec un fort pourcentage de marqueurs « sub-sahariens ». Elles mettent aussi en évidence une étroite relation entre Siouah et l’Afrique de l’Est (Dugoujon, résultats non publiés). Deux hypothèses sont actuellement émises sur l’origine du peuplement de Siouah : soit les premiers Berbères seraient venus de l’Est avec la migration des Proto-Méditerranéens depuis l’Egypte vers le Maghreb ; soit ils seraient arrivés depuis l’Ouest avec la migration secondaire de groupes berbères depuis le Maghreb jusqu’en Egypte. Bien que nous observions des structures génétiques différentes entre le Nord-Ouest et le Nord-Est de l’Afrique, nous n’avons pas encore suffisamment d’éléments pour statuer sur l’une ou l’autre de ces hypothèses.
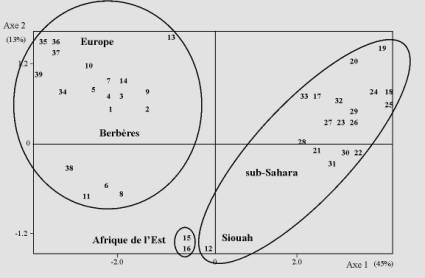
Figure 3. Analyse en Composantes Principales : les Berbères et la base de
données GM.
Références pour les populations nord-africaines :
1, 2, 3, 4, 5 : Maroc (Dugoujon et al., sous presse ; Coudray et al.,
sous presse) ; 6, 7, 8, 14 : Algérie (Dugoujon et al., 2004 ;
Dugoujon, résultats non publiés ; Lefèvre-Witier, 1982) ; 9, 10,
11 : Tunisie (Chaabani et al., 1984 ; Loveslati et al., 2001) ;
13 : Niger (Lefèvre-Witier, 1982) ; 12 : Egypte (Dugoujon,
résultats non publiés).
Références pour les populations est-africaines :
15 : Ethiopie et 16 : Djibouti (Dugoujon et al., 2004).
Références pour les populations sub-Sahariennes :
20, 21 : Sénégal (Blanc et al., 1990) ; 26 : Nigeria (Van Loghem
et al., 1978) ; 17 à 19 : Mali, 22 à 25 : Côte d’Ivoire, 27 à
32 : Cameroun et 33 : République Centrafrique (Dugoujon et al.,
2004).
Références pour les populations sud-ouest
européennes : 34 : Espagne (Giraldo et al., 1998 et 2001) et 35 à
39 : France (Hazout et al., 1991 ; Blanc et Ducos, 1986).
Figure 3. Principal
Coordinate Analysis: the Berbers and the GM database.
References for North-African populations: 1, 2, 3, 4,
5: Morocco (Dugoujon et al., In press; Coudray et al., In press); 6, 7, 8, 14:
Algeria (Dugoujon et al., 2004; Dugoujon, unpublished results; Lefèvre-Witier,
1982); 9, 10, 11: Tunisia (Chaabani et al., 1984; Loveslati et al., 2001); 13:
Niger (Lefèvre-Witier, 1982); 12: Egypt (Dugoujon, unpublished results).
References for East-African
populations: 15: Ethiopia and 16: Djibouti (Dugoujon et al., 2004).
References for sub-Saharan
populations: 20, 21: Senegal (Blanc et al., 1990); 26: Nigeria (Van Loghem et
al., 1978); 17 to 19: Mali, 22 to 25: the Ivory Coast, 27 to 32: Cameroon and
33: the Central African Republic (Dugoujon et al., 2004).
References for South-West
European populations: 34: Spain (Giraldo et al., 1998 and 2001) and 35 to 39:
France (Hazout et al., 1991; Blanc and Ducos, 1986).
Bien que les structures génétiques des populations Berbères soient différentes (pour certains marqueurs) de celles des populations avoisinantes, on remarque tout de même une certaine proximité des groupes nord-ouest africains avec les populations sud-ouest européennes. En effet, bien que nous n’ayons pas d’information précise sur l’origine géographique et temporelle des haplotypes GM, la proximité génétique entre le nord de l’Afrique et les groupes sud-ouest européens conduisent à l’hypothèse d’une origine commune entre ces populations. Deux hypothèses sont actuellement discutées (Barbujani et al, 1994; Myles et al., 2005). Cette origine commune pourrait dater du Paléolithique Supérieur avec l’expansion d’Hommes anatomiquement modernes depuis le Proche-Orient et s’étendant le long des deux rives de la Méditerranée (Straus, 1989; Ferembach, 1985). Elle pourrait aussi avoir eu lieu au cours de la diffusion Néolithique depuis le Proche-Orient, il y a 10 000 ans av. J.-C. (Ammerman et Cavalli-Sforza, 1984).
Au Maroc, on constate qu’il n’y a pas de distinction génétique entre les populations berbérophones et arabophones. Ceci laisserait alors supposer que la conquête arabe a été, sur les populations marocaines, un phénomène principalement culturel avec l’Islamisation et l’Arabisation de certains groupes. Quand est-il alors de l’impact génétique ? En fait, n’ayant pas à ce jour les outils pour mesurer l’impact génétique des migrants Arabes sur les populations nord-africaines, on pourrait penser que cette similarité génétique entre Arabes et Berbères était déjà présente avant la conquête Arabe. On peut aussi imaginer qu’elle résulte, pour partie, d’une homogénéisation du pool génique par échanges réciproques entre les diverses populations.
Finalement, toutes ces observations semblent en accord sur le fait que les Berbères actuels portent les traces d’un peuplement nord-africain ancien qui a du affronter et subir les différents évènements historiques, mais qui a su préserver dans les générations suivantes ses originalités culturelles (notamment ses coutumes et la langue).
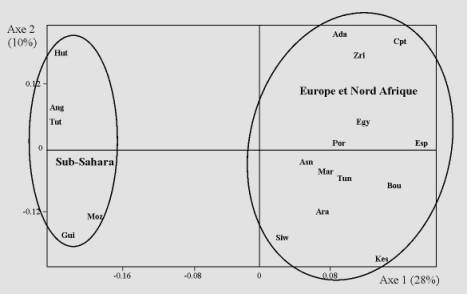
Figure 4. Analyse en Composantes Principales: les Berbères et la base de
données STR.
Références pour les populations nord-africaines :
Ara, Asn, Bou, Mar : Maroc (Bosch et al., 2001 ; Perez-Lezaun et al.,
2000 ; Coudray et al., soumis) ; Tun, Kes, Zri : Tunisie (Cherni
et al., 2005 ; Brandt-Casadevall et al., 2003) ; Ada, Cpt, Egy,
Siw : Egypte (Tahir et al., 2003 ; Dugoujon, résultats non publiés).
Références pour les populations sub-Sahariennes :
Guin : Guinée Bissau (Goncalves et al., 2002) ; Hut, Tut :
Rouanda (Regueiro et al., 2004 ; Tofanelli et al., 2003) ; Ang :
Angola (Beleza et al., 2004) ; Moz : Mozambique (Alves et al., 2004).
Références pour les populations sud-ouest européennes:
Esp : Espagne et Por : Portugal (Perez-Lezaun et al., 2000).
Figure 4. Principal
Coordinate Analysis: the Berbers and the STR database.
References for North-African populations: Ara, Asn,
Bou, Mar: Morocco (Bosch et al., 2001; Perez-Lezaun et al., 2000; Coudray et
al., submitted); Tun, Kes, Zri: Tunisia (Cherni et al., 2005; Brandt-Casadevall
et al., 2003); Ada, Cpt, Egy, Siw: Egypt (Tahir et al., 2003; Dugoujon,
unpublished results).
References for sub-Saharan populations: Guin: Guinea
Bissau (Goncalves et al., 2002); Hut, Tut: Rwanda (Regueiro et al., 2004;
Tofanelli et al., 2003); Ang: Angola (Beleza et al., 2004); Moz: Mozambique
(Alves et al., 2004).
References for South-West European populations: Esp:
Spain and Por: Portugal (Perez-Lezaun et al., 2000).
Remerciements. Nos remerciements s’adressent à toutes les personnes et infrastructures qui ont participé à la collecte des échantillons et l’acquisition des résultats. Ces recherches s’inscrivent dans le cadre du projet EUROCORES de l’ESF (European Science Foundation), contrat no. ERAS-CT-2003-980409 et bénéficient de financements du CNRS (Centre National de la Recherche Scientifique) et du Conseil Régional de Midi-Pyrénées (Toulouse, France).
Bibliographie
Alves, C., Gusmao, L., Damasceno,
A., Soares, B., Amorim, A., 2004, Contribution for an African autosomic STR
database (AmpF/STR Identifiler and Powerplex 16 System) and a report on
genotypic variations. Forensic Sci Int, 139, 201-205.
Ammerman, A.J., Cavalli-Sforza,
L.L., 1984, The Neolithic transition and the Genetics of populations in Europe
(Princeton: Princeton University Press).
Arredi, B., Poloni, E.S.,
Paracchini, S., Zerjal, T., Fathallah, D.M., Makrelouf, M., Pascali, V.L.,
Novelletto, A., Tyler-Smith, C., 2004, A predominantly neolithic origin for
Y-chromosomal DNA variation in North Africa. Am J Hum Genet, 75, 338-45.
Barbujani, G., Pilastro, A., De
Domenico, S., Renfrew, C., 1994, Genetic variation in North Africa and Eurasia:
Neolithic demic diffusion vs. Palaeolithic colonisation. Am J Phys Anthropol,
95, 137-154.
Beleza, S., Alves, C., Reis, F., Amorim, A., Cariacedo, A., Gusmao, L., 2004, 17 STR data (AmpF/STR Identifiler and Powerplex 16 System) from Cabinda (Angola). Forensic Sci Int, 141, 193-196.
Ben Hamed, M., Darlu, P., 2003, Origine et expansion de l’Afro-Asiatique: méthodologie pour une approche pluridisciplinaire. Bull Mem Soc Anthropol Paris, 15, 79-99.
Blanc, M., Ducos, J., 1986, Les allotypes des systèmes Gm et Km dans les provinces françaises. Dans Génétique des Populations, édité par E. Ohayon et A. Cambon-Thomsen, Paris: Colloque INSERM, 142, 207-226.
Blanc,
M., Sanchez-Mazas, A., Van Blyenburgh, N.H., Sevin, A., Pison, G., Langaney,
A., 1990, Interethnic genetic differentiation: Gm polymorphism in eastern
Senegal. Am J Hum
Genet, 46, 383-392.
Bosch, E., Calafell, F.,
Pérez-Lezaun, A., Clarimon, J., Comas, D., Mateu, E., Martinez-Arias, R.,
Morera, B., Brakez, Z., Akhayat, O., Sefiani, A., Hariti, G., Cambon-Thomsen,
A., Bertranpetit, J., 2000, Genetic structure of north-west Africa revealed by
STR analysis. Eur J Hum Genet, 8, 360-366.
Bosch, E., Clarimon, J.,
Perez-Lezaun, A., Calafell, F., 2001, STR data for 21 loci in northwestern
Africa. Forensic Sci Int, 116, 41-51.
Brandt-Casadevall, C., Ben Dhiab,
M., Taroni, F., Gehrig, C., Dimo-Simonin, N., Zemni, M., Mangin, P., 2002,
Tunisian population data on 15 PCR-based loci. Forensic Sci Int, 126, 272-274.
Cavalli-Sforza, L.L., Menozzi, P.,
Piazza, A., 1994, The history and geography of human genes (Princeton:
Princeton University Press).
Chaabani, H., Helal, A.N., Van
Loghem, E., Langaney, A., Benammar Elgaaied, A., Rivat Peran, L., Lefranc, G.,
1984, Genetic study of Tunisian Berbers. I. Gm, Am and Km immunoglobulin
allotypes and ABO blood groups. J Immunogenet, 11, 107-113.
Cherni, L., Loueslati Yaacoubi, B.,
Pereira, L., Alves, C., Khodjet El Kill, H., Ben Ammar El Gaaied, A., Amorim,
A., 2005, Data for 15 autosomal
STR markers (Powerplex 16 System) from two Tunisian populations: Kesra (Berber)
and Zriba (Arab). Forensic Sci Int, 147, 101-106.
Coudray, C., Guitard, E., Lemaire,
O., Cherkaoui, M., Baali, A., Hilali, K., Sevin, A., Kandil, M., Harich, N.,
Melhaoui, M., Larrouy, G., Moral, P., Dugoujon, J.M., 2004, Les allotypes Gm
des immunoglobulines chez les Berbères du Maroc. Antropo, 6, 63-69.
Coudray, C., Guitard, E., Kandil,
M., Harich, N., Melhaoui, M., Baali, A., Sevin, A., Moral, P., Dugoujon, J.M.,
Study of GM immunoglobulin allotypic system in Berbers and Arabs from Morocco.
Am J Hum Biol, sous presse.
Coudray, C., Guitard, E.,
Keyser-Tracqui, C., Melhaoui, M., Cherkaoui, M., Larrouy, G., Dugoujon, J.M.,
Population genetic data of 15 tetrameric short tandem repeats (STRs) in Berbers
from Morocco. Soumis.
Desanges, J., 1990, The
proto-Berbers. Dans General history of Africa, édité par G. Mokhtar (Paris:
Unesco) p 236-245.
Dugoujon, J.M., Hazout, S., Loirat,
F., Mourrieras, B., Crouau-Roy, B., Sanchez-Mazas, A., 2004, Gm haplotype
diversity of 82 populations over the world suggests a centrifugal model of
human migrations. Am J Phys Anthropol, 125, 175-192.
Dugoujon,
J.M., Lemaire, O., Guitard, E., Sevin, A., Larrouy, G., Moral, P., Sabir, B.,
Baali, A., Cherkaoui, M., Diversité des allotypes des immunoglobulines
(systèmes Gm et Km) d’une population berbère de la vallée du Tacchedirt (Haut
Atlas, Maroc). Comparaison
à d’autres populations africaines et européennes et histoire du peuplement de
l’Afrique du Nord. Bull Soc Anthropol Paris. Sous presse.
Edwards, A.W.F., 1992, The method of
maximum likelihood. Dans Likelihood, expanded edition. (Baltimore and London:
The Johns Hopkins University Press) p 70-102.
Fadhlaoui-Zid, K., Plaza, S.,
Calafell, F., Ben Amor, M., Comas, D., Elgaaied, A.B., 2004, Mitochondrial DNA
heterogeneity in Tunisian Berbers. Ann Hum Genet, 68, 222-233.
Fakhry, A., 1973, Siwa Oasis (The
American University in Cairo Press) 214p.
Ferembach, D., 1985, On the origin
of the Iberomaurusians (Upper Palaeolithic: North Africa). A new hypothesis. J
Hum Evol, 14, 393-397.
Field, L.L., Dugoujon, J.M., 1989,
Immunoglobulin allotyping (Gm and Km) of GAW5 families. Genet Epidemiol, 6,
31-34.
Giraldo, M.P., Vallet, M., Guitard,
E., Senegas, M.T., Sevin, A., Nogues, R.M., Aluja, M.P., Dugoujon, J.M., 1998,
Gm and Km immunoglobulin allotypes in one Spanish Pyrenean population: Val
d’Aran. Ann Hum Biol, 25, 453-465.
Giraldo, M.P., Esteban, E., Aluja,
M.P., Nogues, R.M., Backes-Duro, C., Dugoujon, J.M., Moral, P., 2001, Gm and Km
alleles in two Spanish Pyrenean populations (Andorra and Pallars Sobirà): a
review of Gm variation in the Western Mediterranean basin. Ann Hum Genet, 65,
537-548.
Goncalves, R., Jesus, J., Fernandes,
A.T., Brehm, A., 2002, Genetic profile of a multi-ethnic population from
Guine-Bissau (west African coast) using the new PowerPlex 16 System kit.
Forensic Sci Int, 129, 78-80.
Gonzalez-Pérez, E., Via, M.,
Esteban, E., Lopez-Alomar, A., Mazieres, S., Harich, N., Kandil, M., Dugoujon,
J.M., Moral, P., 2003, Alu insertions in the Iberian Peninsula and north west
Africa - genetic boundaries or melting pot? Coll Antropol, 27, 491-500.
Hassan, F.A., 1978, Archeological
explorations of the Siwa Oasis Region, Egypt. Curr Anthropol, 19, 146-148.
Hayward, R.J., 2000, Afroasiatic.
Dans African Languages. An Introduction, édité par B. Heine et D. Nurse
(Cambridge: Cambridge University Press) p 74-98.
Hazout, S., Dugoujon, J.M., Loirat,
F., Constans, J., 1991, Genetic similarity maps and immunoglobulin allotypes of
eleven populations from Pyrenees (France). Ann Hum Genet, 55, 161-174.
Lefèvre-Witier, P., 1982, Structure et dynamique génétiques d’une communauté rurale du Sahara central (Ideles). Ph.D., Université Paul Sabatier, Toulouse, France.
Lefranc, M.P., Lefranc, G., 1990,
Molecular genetics of immunoglobulin allotype expression. Dans The Human IgG
subclasses: molecular analysis of structure, function and regulation, édité par
F. Shakib (Oxford: Pergamanon Press) p 43-78.
Loveslati, B.Y., Sanchez-Mazas, A.,
Ennafaa, H., Marrakchi, R., Dugoujon, J.M., Lefranc, J.M., Elgaaied, A.B.,
2001, A study of Gm allotypes and immunoglobulin heavy gamma IGHG genes in
Berbers, Arabs and sub-Saharan Africans from Jerba Island, Tunisia. Eur J
Immunogenet, 28, 531-538.
Myles, S., Bouzekri, N., Haverfield,
E., Cherkaoui, M., Dugoujon, J.M., Ward, R., 2005, Genetic evidence in support
of a shared Eurasian-North African dairying origin. Hum Genet, 117, 34-42.
Perez-Lezaun, A., Calafell, F.,
Clarimon, J., Bosch, E., Mateu, E., Gusmao, L., Amorim, A., Benchemsi, N.,
Bertranpetit, J., 2000, Allele frequencies of 13 short tandem repeats in
population samples from the Iberian Peninsula and northern Africa. Int J Legal
Med, 113, 208-214.
Regueiro, M., Carril, J.C., Pontes,
M.L., Pinheiro, M.F., Luis, J.R., Caeiro, B., 2004, Allele distribution of 15
PCR-based loci in the Rwanda Tutsi population by multiplex amplification and
capillary electrophoresis. Forensic Sci Int, 143, 61-63.
Tofanelli, S., Boschi, I.,
Bertoneri, S., Coia, V., Taglioli, L., Franceschi, M.G., Destro-Bisol, G.,
Pascali, V., Paoli, G., 2003, Variation at 16 STR loci in Rwandans (Hutu) and
implications on profile frequency estimation in Bantu-speakers. Int J Legal
Med, 117, 121-126.
Renfrew, C., 1991, Before Babel:
speculations on the origins of linguistic diversity. Cambridge Archaeol J, 1,
3-23.
Straus, L.G., 1989, Age of the
modern Europeans. Nature,
342, 476-477.
Tahir, M.A., Herrera, R.J.,
el-Gohary, M., Granoff, M., Amjad, M., 2003, Allele frequency distribution of
Power Plex 1.2 and Profiler Plus short tandem repeats (STR) loci in Egyptian
population. J Forensic Sci, 48, 889-890.
Oudin, J., 1956, L’allotypie de certains antigènes protéiques du serum. C R Hebd Seances Acad Sci, 242, 2606-2608.
Van
Loghem, E., Salimonu, L., Williams, A.I., Osunkoya, B.O., Boyd, A.M., De Lange,
G., Nijenhuis, L.E., 1978, Immunoglobulin allotypes in African populations. I. Gm-Am haplotypes in a Nigerian
population. J Immunogenet, 5, 143-147.
Ward, R.H., 1963, Hierarchical
grouping to optimise an objective function. Am Stat Assoc, 58, 236-244.
