Calvo, L.,
Ricaut, F., Keyser, C., Grimoud, A. M., Ludes, B., Crubezy, E., Lesca, C.,
Lodter, J. Ph., 2001. Etude d’ADN ancien au niveau de la pulpe dentaire
de la série ostéologique de Saint Côme et Damien. Antropo,
1, 21-29. www.didac.ehu.es/antropo
Etude d’ADN ancien
au niveau de la pulpe dentaire de la série ostéologique de Saint
Côme et Damien.
L. Calvo1, F. Ricaut2, C. Keyser2, A. M. Grimoud1, B. Ludes2, E. Crubezy1, C. Lesca3, J. Ph. Lodter1
1 UMR 8555, 37 allées
Jules GUESDE 31000 Toulouse. ANNEMARIE. GRIMOUD@ wanadoo.fr
2 Institut de Médecine
Légale, Laboratoire de biologie moléculaire de l’ADN
ancien, Faculté de Médecine, 7, rue Ulmann 67000 Strasbourg
3 Institut de Pharmacologie et de Biologie Structurale, CNRS 205 route de Narbonne 31067 Toulouse cedex 4
Mots clef : ADN ancien, pulpe dentaire, PCR,
électrophorèse.
Résumé
De
nombreuses études soutiennent l’hypothèse de la
“non-possibilité” de contamination de pulpes dentaires par de
l’ADN exogène ou par des inhibiteurs de la réaction de
polymérisation en chaîne. Nous avons étudié les loci
STR de 20 dents appartenant à quatre sujets de la série
ostéologique de Saint Côme et Damien. L’ADN a
été extrait de pulpes dentaires anciennes recueillies
après fracture des 20 dents. Nous avons mis en évidence une
fragmentation très importante de l’ADN qui inhibe la
réaction de polymérisation en chaîne. Cette inhibition a
été limitée par dilution des volumes réactionnels
des échantillons, nous avons pu mettre alors en évidence des
profils alléliques malgré l’extrême
dégradation de l’ADN.
Abstract
Many
studies had supported that teeth offer to DNA an excellent protection against contamination,
with modern DNA, and against material inhibitif effects of polymerase chain
reaction. We have studied STR loci from four samples from Saint Côme and
Damien collection. We have extract DNA from pulp of broken teeth. We have shown
that the DNA was extremely fragmented, this fragmentation inhibits the
polymerase chain reaction. This inhibition was removed by dilution. We have
displayed allelic profile, in spite of DNA extremely degraded.
Introduction
L’utilisation
de la biologie moléculaire dans l’étude du passé a
commencé en 1984, grâce à l’avènement
d’une nouvelle technique: la réaction de polymérisation en chaîne (PCR) qui a permis
à Higuchi de mettre en évidence pour la première fois la
persistance d’ADN ancien sur des échantillons de tissus
prélevés sur les restes d’un Quagga taxidermisé
(Higuchi et al., 1984 a et b). Au début, l’étude de
l’ADN fossile était réalisée sur des tissus mous
(Paabo, 1985 a et b), mais dès le début des années 90, la
recherche est orientée vres l’étude des tissus durs: les os
(Cooper, 1994; Lee et al., 1991) et les dents (Ginther et al., 1992; Hanni et
al., 1990; Schwartz et al., 1991). La conservation de l’ADN semble
meilleure au niveau des tissus durs. Le problème majeur de
l’étude de l’ADN ancien, outre le fait d’en trouver,
est de prouver son authenticité. Le problème des contaminations
des échantillons par de l’ADN moderne est encore
d’actualité dans cette discipline jeune. L’os est un tissu
poreux qui ne met pas l’ADN à l’abri des contaminations, il
est possible de trouver de l’ADN contaminant en prélevant au
centre du tissu osseux (Hendrik et al. 1999). Les tissus durs de la dent
étant extrêmement minéralisés, l’organe
dentaire est de ce fait le plus dur de l’organisme et serait à
l’abri de contaminations par de l’ADN exogène et des
inhibiteurs de la PCR contenus dans les sols (Woodward et al., 1994). Lors de
l’analyse des échantillons un protocole d’asepsie rigoureux
doit être mis en place (Baker et al., 2001; Brown T.A. et Brown K.A.,
1992; Cone et Fairfax, 1992; Hanni, 1994). Seule l’étude des locus
STR permet de garantir l’authenticité de l’ancienneté
de l’ADN, en effet, cela permet de comparer les résultats obtenus
avec les génotypes de l’équipe de recherche et
d’établir les liens de parenté entre les individus
d’une même sépulture, par l’étude des
allèles (Hummel et al, 1999). Aujourd’hui, la dent est le meilleur
outil pour la recherche d’ADN ancien (Ginther et al., 1992), il semble
qu’elles fournissent une excellente protection de l’ADN quelles que
soient les conditions de conservation des échantillons, avec une
légère augmentation de la dégradation de l’ADN pour
les échantillons conservés dans l’eau (zoo placenta,
dilution, augmentation de l’hydrolyse) (Garcia et al., 1996; Schwartz et
al., 1991). Il est possible d’en extraire de l’ADN même
après un certain degrés d’incinération, ce qui
s’avère impossible au niveau osseux (Baker et al., 2001). Les
études effectuant des recherches d’ADN au niveau des dents
proposent deux méthodes de recueil du matériel, soit l’étude
de dents broyées (Baker et al., 2001; Burger et al., 1999; Di Benedetto
et al., 2000; Hanni et al., 1990; Hummel et al, 1999; Pfeiffer et al., 1999),
soit l’étude de dents sectionnées (Drancourt et al., 1998;
Garcia et al., 1996; Ohira et Yamada, 1999; Pfeiffer et al., 1999; Raoult et
al., 2000; Sivagami et al., 2000; Woodward et al., 1994). Les résultats
obtenus sont meilleurs si les dents sont sectionnées; lorsque
l’anatomie de la dent est préservée c’est la
méthode de choix (Garcia et al., 1996).
L’ADN
ancien subit dès le décès une dégradation rapide,
par différents facteurs qui fragmentent la molécule. L’ADN
ne pourrait pas être amplifié au-delà de 500 paires de
bases et ceci dès la première année suivant le
décès (Pfeiffer et al., 1999). Il apparaît en effet
difficile de trouver des fragments d’ADN au-delà de 200 paires de
base, (Di Benedetto et al., 2000).
L’étude
d’ADN ancien que nous présentons a été
réalisée à partir des dents appartenant à des
sujets de la série ostéologique du site de Saint Côme et
Damien, cimetière du XI au XV siècle, retrouvé lors de
travaux de voirie à Montpellier et dans lequel 600 squelettes ont
été mis à jour. Notre but était
d’établir un protocole de recueil de pulpes dentaires et
d’analyser les échantillons en biologie moléculaire.
Matériel et
méthode
Sélection des
échantillons
Nous avons
sélectionné 20 dents, présentes sur les mâchoires de
quatre individus différents, selon les critères suivants: apex
fermé, absence de fenestration au niveau des tables osseuses (perforation
de la corticale osseuse par la racine dentaire), de mini-fractures coronaires
ou radiculaires, d’abrasions importantes du bord incisif ou des faces
occlusales et de lésions carieuses. Les dents étaient toutes en
excellent état.
Préparation
des échantillons
Avulsions des
dents
Les
mâchoires, préalablement à l’avulsion des dents, sont
désinfectées au formaldéhyde et les avulsions
pratiquées au davier dans un environnement opératoire aseptique:
champs, daviers, tubes et compresses stériles. Dès leur avulsion
les dents sont conservées dans des tubes stériles jusqu’au
moment de recueil des pulpes dentaires.
Trajets
d’incisions
Les dents sont
préalablement lavées à l’eau bi-distillée,
séchées avec des compresses stériles puis exposées
sous un rayonnement ultra violet 30 minutes sur chaque face.
Les trajets
d’incision et de fracture sont réalisés en fonction de
l’anatomie des dents et de celle de la pulpe. L’incision
préfigure le trait de fracture qui permettra l’accès le
plus large possible à la chambre et aux canaux pulpaires. Les incisions
ont été pratiquées en regard de la pulpe sans jamais
atteindre la cavité pulpaire. Elles sont effectuées au moyen
d’un disque diamanté à usage unique monté sur
pièce à main à l’aide d’un mandrin
stérile.
- Pour les dents
mono-radiculées (incisives mandibulaires et maxillaires, canines
mandibulaires et maxillaires, prémolaires mandibulaires) et les
prémolaires supérieures, l’incision est
réalisée sur les faces vestibulaires et palatines ou linguales,
selon qu’il s’agit de dents maxillaires ou mandibulaires.
- Pour les molaires
maxillaires qui présentent trois racines, deux vestibulaires et une
palatine, l’incision vestibulaire passe entre les deux racines
vestibulaires et vient entamer la racine palatine sur la face vestibulaire, une
incision de la face palatine est aussi réalisée (Figures 1 et 2).
- Pour les molaires
inférieures, présentant deux racines, l’incision de la
racine mésiale passe entre les deux canaux mésiaux et entame la
racine distale sur sa face mésiale, le trajet d’incision se
poursuivant sur la face distale.
Fracture des
dents
La
dent est sectionnée sur toute sa longueur en utilisant un syndesmotome
droit placé dans le trait d’incision, la fracture est
réalisée en exerçant simultanément un petit mouvement
de luxation et une légère pression (Figure 3).
|
|
|
|
|||||||||
|
Figure
1.
Incision vestibulaire. Figure
1.
Vestibular incision. |
Figure
2.
Incision palatine. Figure
2.
Palatal incision |
Figure
3.
Molaire supérieure sectionnée. Figure
3.
Broken upper molar |
Recueil de la
pulpe dentaire
La pulpe est récupérée
par curetage, dans des tubes Ependorf de 2ml, au moyen d’une sonde
stérile. En moyenne, nous avons récupéré 7,5mg de
poudre par dent. En fonction de la quantité de pulpe recueillie dans
chacune des 20 dents étudiées nous avons regroupé les
dents dans 9 tubes : sujet 1 : tube 1 (2 molaires), tube 2 (2
prémolaires) tube 3 (1 molaire); sujet 2 : tube 4 (1 molaire et 1
prémolaire), tube 5 (4 molaires) ; sujet 3 : tube 6 (3
molaires), tube 7 (1 canine et 2 molaires) ; sujet 4 : tube 8 (1
prémolaire et 1 incisive), tube 9 (1 prémolaire).
Protocole d’analyse de l’ADN ancien
Extraction
Les pulpes dentaires ont été mises à
incuber dans 500ml de tampon d’extraction : EDTA 5mM, SDS 2%, Tris-HCL
10 mM, acétate de sodium 0,3M, protéinase K 1mg/ml et 25 ml de protéinase K,
à 50°C durant une nuit, sur un agitateur; les tubes sont
fermés hermétiquement avec du papier parafilm.
Purification
Après incubation, nous
ajoutons à la solution d’extraction 500ml de la solution:
phénol/chloroforme/alcool isoamylique (commercialisé sous cette
forme, le phénol et le chloroforme sont à volume égal,
l’alcool isoamylique à volume moindre : 25 – 24 –
1, le pH est de 7,9). Cette solution est centrifugée 5mn à 3000
rpm, à température ambiante. Au surnageant, prélevé
à l’aide de pipettes à embouts cotonnés, sont
ajoutés 1000ml de solution d’hybridration et 130ml de résine (protocole B
du Kit de purification d’ADN CLEANMIX).
Amplification
La PCR est
réalisée avec le Kit AmpF-STR® Profiler PlusTM, de
Perking Elmer. L’amplification est réalisée, à
partir de 10ml de chacun des 9 tubes,
dans le thermocycleur T3 de BIOMETRA.
L’amplification
suit les recommandations du fabricant, seul le nombre de cycles a
été modifié pour optimiser la réaction, 37 cycles
sont programmés au lieu des 28 mentionnés dans la fiche technique
Electrophorèse
Après
amplification, 2 ml sont
prélevés sur les 10 ml de volume
total de PCR. Le premier puits du gel contient l’échelle
allélique (Ladder STR) qui permet de vérifier la migration de
chacun des allèles étudiés (tableau 1) et de valider leur
taille. L’analyse du produit de la P.C.R. est effectuée sur gel de
polyacrylamide à 6%, sur le séquenceur: ABI Prism 373A Applied
Biosystems DNA sequencer.
a :La
classification par taille est la base de la reconnaissance des
différents
allèles
d’un même gène,
contenus dans AmpF-STR Allelic Ladders
b :Dans
quelques littératures ce locus est désigné : D6S502
c : R peut
représenter aussi bien les nucléotides A ou G
|
Loci |
Localisation |
Motif reconnu |
(pb)a |
coloration |
|
D3S1358 |
3p |
TCTA(TCTG)1-3(TCTA)n |
114-142 |
bleu |
|
VWA |
12p12-pter |
TCTA(TCTG)3-4(TCTA)n |
157-197 |
bleu |
|
FGA |
4q28 |
(TTTC)3TTTT TTCT |
219-267 |
bleu |
|
|
|
(CTTT)nCTCC(TTCC)2 |
|
|
|
Amélogénine X |
p22.1-22.3 |
|
107 |
vert |
|
|
Y :p11.2 |
|
113 |
|
|
D8S1179b |
8 |
(TCTR)nc |
128-168 |
vert |
|
D21S11 |
21 |
(TCTA)n(TCTG)n |
189-243 |
vert |
|
|
|
[(TCTA)3TA(TCTA)3TCA |
|
|
|
|
|
(TCTA)2TCCA
TA](TCTA)n |
|
|
|
D18S51 |
18q21.3 |
(AGAA)n |
273-341 |
vert |
|
D5S818 |
5q21-31 |
(AGAT)n |
135-171 |
jaune |
|
D13S317 |
13q22-31 |
(GATA)n |
206-234 |
jaune |
|
D7S820 |
q11.21-22 |
(GATA)n |
258-294 |
jaune |
Tableau 1. Amorces du Kit
AmpF-STR® Profiler PlusTM
Table
1. Primer of the Kit AmpF-STR® Profiler PlusTM
Résultats
Pour les 20 dents
analysées, le contenu de la chambre pulpaire, avait une coloration
brune. Nous récupérons, en moyenne, 7,5mg de matériau par
dent. La quantité de pulpe dentaire recueillie n’est pas
proportionnelle au volume de la chambre et des canaux de la dent, elle varie
pour un même type de dent.
Les résultats
présentés ont été obtenus au terme de 4
étapes comprenant huit PCR et les électrophorèses sur gel
de polyacrylamide.
1° étape :
amplification initiale
La PCR est réalisée
en utilisant le volume maximal conseillé par PERKIN ELMER, soit 4ml. Le test comprend un
témoin positif, et un témoin négatif. Le témoin
positif, fourni dans le Kit AmpFlSTR® Profiler PlusTM, permet de
valider la réaction. Ces volumes réactionnels ne nous ont pas
permis de visualiser les produits de la réaction d’amplification,
sur les électrophorégrammes. Le contrôle positif a
donné le résultat attendu.
2° étape :
évaluation de la quantité de matériel et test
d’inhibition
A ce stade de nos investigations, d’une part nous
vérifions si la quantité de matériel extrait n’est
pas insuffisante et d’autre part nous testons la présence de
facteurs d’inhibition.
Ainsi, nous rassemblons les trois tubes d’extraction
du sujet 1 en un seul, puis nous concentrons l’extrait sur Microcon
jusqu’à un volume de 20 µl. L’absence de pic à
l’électrophorégramme pour cet échantillon concentré,
conduit à vérifier l’hypothèse de la présence
de facteurs d’inhibition dans l’ADN ancien, nous réalisons
des tests d’inhibition (tableau 2). L’ADN ancien des sujets 1 et 2
inhibe l’amplification de l’ADN moderne provenant du témoin
positif.
|
|
ADN moderne |
ADN ancien |
TE |
MIX |
Electrophorégramme |
|
Témoin négatif |
0 µl |
0 µl |
4 µl |
6 µl |
Pas de pic |
|
Témoin positif |
0,5 µl |
0 µl |
3,5 µl |
6 µl |
Profil attendu |
|
Sujet 1 |
0,5 µl |
3,5 µl |
0 µl |
6 µl |
Pas de pic |
|
Sujet 1 |
0 µl |
4 µl |
0 µl |
6 µl |
Pas de pic |
|
Sujet 2 |
0,5 µl |
3,5 µl |
0 µl |
6 µl |
Pas de pic |
|
Sujet 2 |
0 µl |
4 µl |
0 µl |
6 µl |
Pas de pic |
Tableau 2. test
d’inhibition
Table 2. Inbition test
3° étape calcul du
seuil d’inhibition
Pour vérifier
l’hypothèse de la présence d’inhibiteur, il
était nécessaire de calculer le seuil d’inhibition. Pour
chacun des sujets 2, 3 et 4, nous disposons de 2 tubes, une amplification est
réalisée à 1µl pour un des tubes et 1,5µl pour
l’autre tube. L’échantillon du sujet 1 a subi une
amplification à 1µl et 1,5µl à partir du même
tube, issu d’une concentration préalablement
réalisée. Le volume d’extrait d’ADN étant
diminué, la Taq polymérase est en excès par rapport
à l’étape précédente.
Pour l’échantillon
concentré du sujet 1 issu de la concentration,
l’électrophorèse ne révèle pas de
réaction. Pour les sujets 2, 3 et 4, un pic apparaît à
l’électrophorégramme pour un volume d’1µl. Pour
le sujet 2, nous avons amplifié le locus correspondant au gène de
l’amélogénine X, d’une taille de 106 paires de bases.
Le sujet 3 présente à l’électrophorégramme un
pic situé à 205,48 paires de bases et correspondant au locus
D21S11, allèle 29. Pour le sujet 4 le locus D21S11, allèle 12,
d’une taille de 142,75 paires de bases est amplifié. Ces
résultats situent le seuil d’inhibition à 1µl, sauf
pour l’échantillon concentré du sujet 1.
4° étape :
amplification au seuil d’inhibition
Nous avons, dans un premier temps réalisé une
amplification à 1µl, pour chacun des tubes. Le sujet 3
présente un résultat pour le locus D21S11, allèle 30,2, d’une
taille de 211,11 paires de bases.
Par la suite, pour palier les variations intra-tubes et inter-tubes pour un même sujet, nous travaillons avec de plus petits volumes d’extrait allant de 0,5µl à 2µl; la faible quantité de matériel présent dans les tubes rendant les solutions non homogènes. Pour le sujet 2 nous amplifions les loci suivants : D3S1358 (allèle 17) de 133,7 paires de bases, VWA (allèle 17) de 179,46 paires de bases, amélogénine X de 106 paires de bases, D21S11 de 209,38 paires de bases (allèle 30) (Figure 4) et 223,48 paires de bases (allèle 33,2), FGA (allèle 22) de 233,36 paires de bases (Figure 4) et D7S820 (allèle 10) de 272,71 paires de bases (Figure 4). Pour le sujet 4 nous amplifions les loci : D21S11 (allèle 30,2) de 211,44 paires de bases, D3S1358 (allèle 14) de 121,77 paires de bases et amélogénine X de 106 paires de bases. La diminution du volume des extraits a permis de diminuer partiellement l’effet des inhibiteurs.
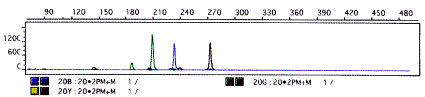
Figure 4. Électrophorégramme
des loci D21S11, FGA, D7S820
Figure 4. Elctrophoregram
of D21S11, FGA, D7S820 loci
Une PCR est effectuée sur
un volume de 4µl à partir d’extraits dilués au 1/10.
Le sujet 1 présente des pics d’amplification pour le locus D21S11
(allèle 32,2) de 219,48 paires de bases.
Au terme de ces quatre
étapes, nous avons pu mettre en évidence les profils
alléliques qui sont représentés dans le tableau 3
|
Sujets (tubes) |
Amélogénine |
D21S11 |
D3S1358 |
VWA |
FGA |
D7S820 |
|
1 (1, 2,3) |
|
32,2 |
|
|
|
|
|
2 (4) |
XX |
30/33,2 |
17 |
17 |
22 |
10 |
|
2 (5) |
XX |
|
|
17 |
|
|
|
3 (6) |
|
29 |
|
|
|
|
|
3 (7) |
|
30,2 |
|
|
|
|
|
4 (8) |
XX |
30,2 |
14 |
|
|
|
|
4 (9) |
|
12 |
|
|
|
|
Tableau 3. Allèles
identifiés
Tableau
3. Allelic
identification
Discussion
L’analyse
de l’ADN ancien étant directement liée, aux risques de
contamination, à la dégradation du matériel
génétique et à la difficulté de recueil des
échantillons, nous nous sommes attachés au cours de notre
étude à intégrer les répercussions associées
à ces différents facteurs et à définir des
conditions requises pour en palier les effets.
En ce qui
concerne la sélection et le recueil des échantillons, nous avons
limité tout risque de contaminations, d’une part en
sélectionnant des dents sur maxillaire et mandibule, présentant
après avulsion un apex fermé et exemptes de lésions
carieuses ou traumatiques sources de contaminations, et, d’autre part en
réalisant l’ensemble des manipulations de manière aseptique.
Selon la
technique de section des dents monoradiculées d’Aboudharam (Raoult
et al., 2000) nous avons mis au point une méthode d’incision et de
section étendue aux différents morphotypes de dents. Ainsi pour
les pluriradiculées qui présentent anatomiquement une morphologie
complexe nous avons défini des schémas d’incisions et de
fractures assurant le meilleur recueil d’échantillons. En effet,
la faible quantité de matériel présent dans la chambre et
les canaux pulpaires, nécessitait une technique permettant
d’utiliser tout type de dent et d’accéder à la totalité
de la pulpe pour optimiser le recueil des échantillons.
Les résultats que nous
obtenons, témoignent que notre protocole d’asepsie est efficace,
il évite les contaminations par de l’ADN moderne, l’absence
de fragments de grande taille et les résultats des témoins
négatifs tendent à prouver qu’aucune molécule
d’ADN moderne ne s’est glissée dans les échantillons
lors des différentes manipulations.
En ce qui
concerne la taille des fragments amplifiés, nos résultats sont en
accord avec ceux des études antérieures. Les allèles que
nous identifions sont généralement d’une taille
inférieure à 200 paires de bases (X: 107 paires de bases,
D8S1179: 130 à 163 paires de bases, D21S11: 190 à 240 paires de
bases, D3S1358: 110 à 140 paires de bases, VWA: 155 à 195 paires
de bases). Nous n’avons pu amplifier que deux allèles de plus de
200 paires de bases (FGA: 217 à 266 paires de bases et D7S820: 257-293
paires de bases). Di Benedetto affirme qu’il est extrêmement
difficile d’amplifier des fragments d’ADN ancien au-delà de
200 paires de bases (Di Benedetto et al. 2000). Ceci est confirmé par la
plupart des études dont les résultats varient de 95 à 500
paires de bases. Nos résultats confirment la présence d’une
multitude de petits fragments inférieurs à 100 paires de base.
Ceci tend à démontrer l’extrême dégradation de
l’ADN chez les sujets de Saint Côme et Damien.
Lors de la
fracture des dents, nous avions remarqué une coloration brune des pulpes
dentaires. Nous avons comparé cette coloration avec celle d’autres
sujets d’une autre série, nous avons pu observer des colorations
noires ou blanches. Ces colorations ne semblent en aucun cas avoir des
répercutions sur la qualité des résultats comme le
confirme l’étude de Raoult (Raoult et al., 2000).
La faible
quantité d’ADN et son importante dégradation rendent les
mélanges non homogènes dans un même du tube (Ramos et al.
1994; Burger et al. 1999). Ce phénomène permet de comprendre que
les quelques microlitres d’extraits prélevés pour
l’amplification, peuvent être différents qualitativement
pour un même tube. La qualité des résultats ne semble
liée ni au type de dents, ni à la quantité
d’échantillons. C’est ainsi qu’en raison de leur
volume pulpaire, nous pensions recueillir une quantité plus importante
d’échantillons au niveau des canines et des molaires. Par
ailleurs, nous notons aussi que la quantité de poudre recueillie est
sans rapport avec le type de dent. Les meilleurs résultats sont obtenus
pour le tube qui contenait la poudre de pulpe d’une molaire et
d’une prémolaire; alors que nous n’avons obtenu qu’un
seul locus pour les 5 pulpes du sujet 1 placées dans le même tube.
La
possibilité d’extraire et d’amplifier de l’ADN ancien
d’une pulpe dentaire est lié à la quantité de poudre
retrouvée au niveau d’une seule dent, ce qui est directement en
rapport avec le volume pulpaire et l’état de la pulpe au
décès de l’individu. Ceci confirmerait les
hypothèses avancées par Garcia (Garcia et al., 1996), selon
lesquelles la qualité des résultats pourrait varier en fonction
de l’âge du sujet à son décès, et des
antécédents pathologiques de l’organe dentaire, tout ceci
ayant des répercussions sur le volume et l’état
physiologique de la pulpe et donc sur le matériel génétique
qu’elle contient.
Nous
démontrons la présence d’inhibiteurs au sein de
l’extrait d’ADN ancien. Ce résultat va à
l’encontre de ce qui est habituellement reporté dans la
littérature sur les recherches d’ADN au niveau des dents.
Celles-ci sont considérées comme des coffres-forts pour
l’ADN ancien, le mettant à l’abri des contaminations (Scott
et al., 1994). En effet, bien que la dent soit l’organe le plus dur et le
plus dense de l’organisme, l’histologie démontre que cet
organe n’en est pas pour autant imperméable.
Lors de la
recherche d’inhibiteurs, lorsque nous nous diminuons la quantité
d’ADN, nous diminuons partiellement l’effet des inhibiteurs. Ceci
soulignerait que les inhibiteurs présents dans les échantillons
sont des inhibiteurs en “ trans ” (Orlando et Hanni,
2000). En diluant, nous avons encore diminué la quantité des
inhibiteurs, mais également la quantité d’ADN
engagée dans la réaction, de telle sorte qu’il n’y en
avait plus suffisamment pour l’amplification. Néanmoins, nous ne
pouvons pas envisager la présence d’inhibiteurs décrits
classiquement, comme les produits de Maillard (Hanni, 1994). Ceux-ci devraient
inhiber autant les petits fragments que les fragments de grande taille. Nous
pensons plutôt, que l’extrême dégradation de
l’ADN provoque une surabondance des petits fragments d’ADN qui
monopolisent les amorces et empêchent l’amplification des rares
fragments intacts. Nous ne pouvons pas parler “d’inhibiteurs”
au sens propre du terme puisque c’est le matériel lui-même
qui inhiberait la réaction de PCR.
Notre protocole
d’extraction a permis d’identifier des allèles, la
difficulté pour obtenir ces résultats nous conduit à
avancer l’hypothèse que l’ADN extrait des sujets de Saint
Côme et Damien est extrêmement dégradé.
En conclusion,
le protocole mis au point permet de recueillir la pulpe de différents
types de dents avec un maximum d’efficacité et dans des conditions
d’asepsie qui évite la contamination par de l’ADN moderne.
La phase d’extraction a permis d’identifier des allèles, les
résultats obtenus démontrent la dégradation de l’ADN
ancien dans les échantillons étudiés et les
difficultés d’amplifications associées. Une porte
s’ouvre au terme de cette étude sur la maîtrise de la
technique d’amplification de petits fragments d’ADN ancien.
Remerciements. Nous remercions pour
leur précieuse collaboration: Toute l’équipe du Professeur
B LUDES,Laboratoire d’Anthropologie moléculaire de
l’Institut de médecine légale de Strasbourg, 7, rue Ulmann,
67000 Strasbourg; Madame M. DEFAIS, Madame C. LESCA, IPBS, CNRS, 205 route de
Narbonne, 31400 Toulouse; Monsieur B. PAJOT, Madame S. DUCHENNE, UMR CNRS 8555,
37, allées Jules Guesde, 31000 Toulouse.
Bibliographie
Baker L.E., Mc Cormick W. F.,
Matteson K. J., 2001, A silica-based mitochondrial DNA extraction method
applied to forensic hair shafts and teeth. Journal of Forensic Sciences, 46,
126-130.
Brown T.A., Brown K.A., 1992,
Ancient DNA and the archeologist. Antiquity, 66, 10-23.
Burger J., Hummel S., Hermann B.,
Henke W., 1999, DNA preservation: A microsatellite-DNA study on ancient
skeletal remains. Electrophoresis, 20, 1722-1728.
Cooper, A., 1994, DNA from museum
specimens. in ancient DNA (New-York: Springer-Verlag), pp .
Cone, R. W., Fairfax, M. R.,
1993, Protocol for ultraviolet irradiation of surfaces to reduce PCR contamination.
PCR Methods and Applications, 9, 15-17.
Di Benedetto G., Nasidze I. S.,
Stenico M., Nigro L., Krings M., Lanzinger M., Vigilant L., Stoneking M., Paabo
S., Barbujani G., 2000, Mitochondrial DNA sequences in prehistoric human
sequences in prehistoric human remains from the alps. European Journal of Human
Genetic , 8, 669-677.
Drancourt M., Aboudharam G.,
Signoli M., Dutour O., Raoult D., 1998, Detection of 400-year-old Yersinia
pestis DNA in human dental pulp: An approach to the diagnosis ofn ancient
septicemia. Proc. Natl. Acad. Sci. USA, 95, 12637-12640.
Garcia A. A., Munoz I., Pestoni
C., Lareu M. V., Rodriguez-Calvo M. S., Carracedoa., 1996, Effect of
environmental factors on PCR-DNA analysis from dental pulp. International
Journal of Legal Medicine, 109, 125-129.
Ginther C., Issel-Tarver L., King
M.-C., 1992, Identifying individuals by sequencing mitochondrial DNA from
teeth. Nature genetics, 2, 135-138.
Hanni C., 1994,
Archéologie et paléontologie moléculaires :
application à l’étude des gisements. Thèse
Doctorale, MON 20074.
Hanni I C., Laudet V., Sakka M.,
Begue A., Stehelin D., 1990, Amplification of mitochondrial DNA fragments from
ancient humain teeth and bones. Compte Rendu. De l’Académie des
Sciences Paris, 310, Série III, 365-370.
Hendrik N., Poinar B.,
Stankiewicz B. Arthur M., 1999, Protein preservation and DNA retrieval from
ancient tissues. Proc. Natl. Acad. Sci. USA, 96, 8426-8431.
Higuchi R., Bowman B., Freiberger
M., Ryder O., Wilson A.C., 1984a, DNA sequences from the quagga, an extinct
member of the horse family. Nature, 312, 282-284.
Higuchi R., Wilson A. C., 1984b,
Recovery of DNA from extinct species. Fedn. Proc., 43, 1557-1960.
Hummel S., Schultes T., BramantiI
B., Herrmann B., 1999, Ancient DNA profiling by megaplex amplifications.
Electrophoresis, 20, 1717-1721.
Lee H., Pagliaro E., Berka K.,
Folk N., Anderson, Ruano G., Keith T., Phipps P., Herrin G., Garner D.,
Gaensslen R., 1991, Genetic markers in human bone, Dezoxyribonucleic acid (DNA)
analysis. JFSCA, 36, 320-330.
Ohira H., YAMADA Y., 1999,
Advantages of dental mitochondrial dna for detection and classification of the
sequence variation using restriction fragment length polymorphisms. American
Journal of Forensic Medecineand pathology, 20, 261-268.
Orlando L. et Hanni C., 2000, Du
nouveau pour l’ADN ancien. Société Française de
Génétique, 16, 1-16.
Paabo S., 1985a, Molecular
cloning of ancient egyptian mummy DNA. Nature, 314, 644-645.
Paabo S., 1985b, DNA in ancient
egyptian mummies. J. Arch. Science, 12, 411-417.
Pfeiffer H., Huhne J., Seitz B.,
Brinkmann B., 1999, Influence of soil storage and exposure period on DNA
recovery from teeth. International Journal of Legal Medicine., 112, 142-144.
Raoult D., Aboudharam G., Crubezy
E., Larrouy G., Ludes B., Drancourt M., 2000, Molecular identification by
“suicide PCR” of Yersinia pestis as the agent of medieval black
death. Proc. Nat. Acad. Sci. USA, 97, 12800-12803.
Schwartz T., Schwartz E.,
Mieszerski L., Mc Nally L., Kobitinsky L.,1991, Characterisation of
deoxyribonucleic acid (DNA) obtained from teeth subjected to various
environnemental conditions. Journal of Forensic Sciences, 36, 979-990.
SivagamiI A. V., Rajeswarao A.,
Varshney U., 2000, A simple and cost-effective method for preparing DNA from
the hard tooth tissue, and its use in polymerase chain reaction amplification
of amelogenin gene segment for sex determination in an indian population.
Forensic Science International, 110, 107-115.
Woodward S.R., King M.J., Chiu
.M, Kuchar M.J., Griggs W.1994, Amplification of ancient nuclear DNA from teeth
and soft tissues. PCR Methods and amplification Technical Tips, 3, 244-247.