Calò,
C. M., Varesi, L., Memmì, M., Mameli, G. E., Moral, P., Vona, G., 2001.
Analisi di 5 microsatelliti nelle tre maggiori isole del Mediterraneo
Occidentale. Antropo, 1, 7-19. www.didac.ehu.es/antropo
Analisi
di 5 microsatelliti nelle tre maggiori isole del Mediterraneo Occidentale
C.M.
Calò1, L. Varesi2, M. Memmì2,
G.E. Mameli1, P. Moral3, G. Vona1
1
Dipartimento di Biologia Sperimentale, Sezione di Scienze Antropologiche,
Università di Cagliari, Italy
2
Faculté de Sciences et Technique (Civaren), Université de Corse,
20250 Corte, France
3
Facultat de Biologia, Secciò d'Antropologia, Universitat de Barcelona,
0828 Barcelona, Spain.
Parole
Chiave:
microsatelliti, dinucleotide, Sardegna, Sicilia, Corsica
Riassunto
Sono stati esaminati 360 individui
provenienti dalle tre maggiori isole del Mediterraneo Occidentale: Sardegna
(Italia), Sicilia (Italia) e Corsica (Francia). Il DNA è stato
amplificato tramite PCR e i frammenti sono stati separati usando un gel di
poliacrilammide. I microsatelliti analizzati nel presente lavoro sono: GABRB3,
D15S108, LIPC, D13S115, D13S270. La distribuzione delle frequenze alleliche
hanno mostrato differenze significative tra le isole. I 5 microsatelliti
presentano un elevato potere di discriminazione (PD) e un elevato contenuto di
informazione polimorfica (PIC). Il potere di discriminazione più alto
è stato trovato per il GABRB3 e secondariamente per D13S115, che ha
mostrato anche il più alto valore del PIC. I confronti con le altre
popolazioni hanno evidenziato un netto raggruppamento delle popolazioni per
continenti, in particolare gli europei mostrano una più ridotta
eterogeneità genetica rispetto agli altri continenti. I risultati
ottenuti dimostrano che tali marcatori sono utili nello studio della
variabilità genetica delle popolazioni umane e nell’analisi
forense.
Abstract
About 360 unrelated individuals from the
three largest islands of the Western Mediterranean: Sardinia (Italy), Sicily
(Italy) and Corsica (France) were examined. The DNA was amplified by means of
PCR and the fragments were separated using a polyacrylamide gel. The
microsatellites analysed in the present work are: GABRB3, D15S108, LIPC,
D13S115, D13S270. The allele frequencies distribution showed significant
differences between the islands. The 5 microsatellites have a high
discrimination power (PD) and a high Polymorphic Information Content (PIC). The
highest discrimination power was found for GABRB3 and secondarily for D13S115,
which also showed the highest values of PIC. The comparison with other
populations underlined the populations from the same continent cluster
together. Particularly Europeans shows a less genetic heterogeneity than other
continents. The results demonstrate that these microsatellites are useful in
the study of genetic variability of the human populations and in forensic
analysis.
Introduzione
Tra i marcatori che vengono usati nelle
analisi genetiche, nelle diagnosi mediche e in medicina forense, gli STRs
(Short Tandem Repeat polymorphisms) hanno recentemente assunto una grande
importanza. Questi possono essere utilizzati con differenti scopi nel campo
della genetica, e si sono dimostrati di grande aiuto sia nello studio della
struttura genetica delle popolazioni sia in medicina forense. La
variabilità mostrata da molti degli STRs li rende estremamente
interessanti come marcatori antropologici (Harding, 1992).
Tecnicamente, gli STRs, in confronto con
altri marcatori, presentano il vantaggio di poter essere analizzati tramite PCR
e i loro alleli possono essere individuati senza ambiguità con un gel di
poliacrilamide e non utilizzando il radioattivo. Questa relativa
facilità di studio ha portato ad un rapido incremento nel numero degli
STRs esaminati e delle popolazioni studiate.
Al fine di dare un contributo alla
conoscenza della distribuzione delle frequenze alleliche degli STRs nelle
popolazioni della Corsica (Francia), Sardegna e Sicilia (Italia), le tre maggiori
isole del Mediterraneo Occidentale (Figura 1), sono stati studiati tre campioni
provenienti dalle tre isole.
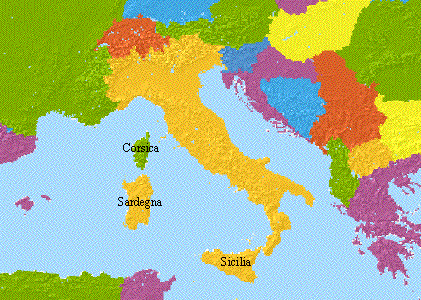
Figura 1. Localizzazione geografica delle
popolazioni esaminate.
Figure 1. Geographical localization of
the examined populations.
I cinque STRs utilizzati in questo studio
sono microsatelliti dinucleotidi (CA)n (GABRB3, D15S108, D13S115, D13S270) e
(CT)n (LIPC). La scelta dei questi marcatori è stata suggerita
dall’alto valore di eterozigosità e dall’alto contenuto di
informazione polimorfica (PIC) riscontrato da vari autori (Bowcock, 1993;
Bowcock et al., 1993; Gyapay et al., 1994) che li rendono adatti per
l’utilizzo in genetica di popolazioni e in medicina forense (Bowcock et
al., 1994).
La Sicilia (Italia), la più grande
isola del Mediterraneo Occidentale (25,707 km2), ha una
densità di 193 abitanti per km2. Per la sua particolare
posizione geografica, la Sicilia è stata per generazioni teatro di
numerose invasioni e dominazioni da parte di diverse popolazioni.
Dall’arrivo nell’isola dei primi gruppi (Sicani, Elimi e Siculi) la
Sicilia ha visto un’alternanza di Fenici, Greci, Romani, Vandali, Goti,
Arabi, Normanni e Spagnoli. L’importanza del contributo genetico di
questi gruppi sulla popolazione siciliana deve essere ancora chiarito (Vona et
al., 2000).
La popolazione della Sardegna (Italia)
è un esempio di isolato genetico, come dimostrato dalla ben documentata
unicità delle sue frequenze geniche (Piazza et al., 1985; Modiano et
al., 1986; Walter et al., 1991; Vona et al., 1992). La Sardegna è
rimasta isolata per lungo tempo in due modi: dal resto d’Italia e dalle
regioni mediterranee ed all’interno del suo stesso territorio. La ragione
di questo isolamento può essere spiegata dall’insularità e
dalla posizione geografica, che non favoriscono vaste migrazioni, dalle
condizioni geomorfologiche interne, che creano barriere tra le comunità
frammentando la popolazione, e dalla scarsa densità di popolazione, che
attualmente è 70 abitanti per km2, in un territorio ampio
(24.000 km2). Diversi studi sulla struttura genetica e matrimoniale,
sulla consanguineità e sulla kinship dei cognomi hanno mostrato un alto
grado di isolamento interno ed esterno (Cappello et al., 1996; Floris e Vona,
1984; Moroni et al., 1972, Vona et al., 1996; Vona, 1997).
La Corsica (Francia), la terza isola del
Bacino del Mediterraneo (8,682 km2; 28.8 abitanti per km2),
è caratterizzata da una storia estremamente complessa. L’isola fu
sottoposta a svariati insediamenti da parte di differenti popolazioni: Liguri,
Fenici, Greci, Romani, Bizantini, Pisani, e Francesi. Un alto grado di
isolamento è stato suggerito dalle sue caratteristiche geomorfologiche,
i suoi piccoli villaggi (spesso con meno di 400 abitanti),
l’unicità delle sue frequenze geniche per alcuni polimorfismi e
una forte eterogeneità se confrontata con le altre popolazioni
mediterranee (Vona et al., 1995; Moral et al., 1996; Memmì et al., 1998;
Varesi et al., 2000)
Materiali
e Metodi
Sono stai esaminati 360 campioni
provenienti dai centri trasfusionali e dagli ospedali della Corsica (Francia),
Sardegna e Sicilia (Italia), precisamente 130 erano corsi del distretto di
Corte e Ajaccio (centro e sud-ovest dell’isola), 130 siciliani
provenienti dalle province di Palermo e Trapani (Sicilia occidentale) e 100 sardi
delle province di Cagliari (sud Sardegna) e Nuoro (centro Sardegna)
Il campione è costituito da
individui di entrambi i sessi, apparentemente sani, originari delle tre isole
da almeno tre generazioni.
Il DNA è stato isolato da sangue
intero, periferico, con il metodo tradizionale di digestione con proteinasi K
ed estrazione con fenolo-cloroformio, nel Dipartimento di Biologia Sperimentale
dell’Università di Cagliari (Italia).
L’amplificazione è stata
condotta utilizzando i primers riportati in tabella 1.
|
GABRB3 |
L:
5’-CTCTTGTTCCTGTTGCTTTCAATACAC-3’ R:
5’-CACTGTGCTAGTAGATTCAGCTC-3’ |
(Beckmann et al., 1993) |
|
D15S108 |
MFD102L: 5’-ATTCTTAACAGGAAGTGAGGG-3’ MFD102R:
5’-AACATGAGTTTCAGAGGGG-3’ |
(Beckmann et al., 1993) |
|
LIPC |
HLIP1: 5’-ATGTGATGTCAGTGCTGCCAGTCCA-3’ HLIP2:5’-ACTGACATTTGAAAGATACGACCAC-3’ |
(Bhattacharrya et al., 1991) |
|
D132115 |
MS34L: 5’-TGTAAGGAGAGAGAGATTTCGACA-3’ MS34R:
5’-TCTTAGCTGCTGGTGGTGG-3’ |
(Bowcock et al.,1993) |
|
D13S270 |
MS34L: 5’-TGTAAGGAGAGAGAGATTTCGACA-3’ MS34R:
5’-TCTTAGCTGCTGGTGGTGG-3’ |
(Gyapay et al., 1994) |
Tabella 1. Primers utilizzati per
l’amplificazione e fonti bibliografiche
Table 1. Primers used for the amplification and
references.
Tutti i microsatelliti sono stati amplificati
in un volume totale di 12.5 ml contenente
Buffer, MgCl2 1,5 mM, dNTP 2,5 mM ognuno, 10 mM of ogni primer
e 0.6 unità di Taq polimerasi.
La PCR è stata portata avanti con
i seguenti cicli: denaturazione iniziale a 94°C per 7', seguita da 30 cicli
costituiti da denaturazione (94°C per 30"), ibridazione (55°C per
30") ed estensione (72°C per 30"). L’amplificazione
è stata completata con un’estensione finale a 72°C per 7'. I
prodotti amplificati sono stati sottoposti ad elettroforesi verticale in un gel
di poliacrilamide all’8%. Dopo l’elettroforesi, le bande sono state
visualizzate con una colorazione di nitrato d’argento. I genotipi sono
stati determinati usando marcatori di peso molecolare disponibili in commercio
e alcuni marcatori prodotti nel nostro laboratorio dalla combinazione di
più campioni precedentemente sequenziati.
Analisi
statistiche
Le frequenze alleliche sono state
calcolate mediante la conta degli alleli. La possibile divergenza
dall’equilibrio di Hardy-Weinberg è stata determinata con tre
differenti metodi statistici: il likelihood ratio test, il test esatto della
catena di Markov, in accordo con Guo e Thompson (1992) e l’Fis che
dà una stima del coefficiente di inbreeding e rappresenta
l’ampiezza della deviazione dall’equilibrio di Hardy-Weinberg
La stima dell’eterozigosità
attesa è stata calcolata mediante il metodo descritto da Nei e
Roychoudhury (1974). Il linkage disequilibrium per i loci situati sullo stesso
cromosoma è stato verificato attraverso il test esatto della catena di
Markov. E’ stata anche studiata l’eterogeneità genetica tra
le popolazioni esaminate seguendo il metodo di Fisher. Tutti i test sono stati
eseguiti con il programma GENEPOP, versione 1.2 (Raymond e Rousset, 1995).
I valori del contenuto di informazione polimorfica
(PIC) sono stati ottenuti in accordo con Weber (1990) e il potere di
discriminazione (PD) è stato calcolato secondo la formula di Fisher
(Fisher, 1951). I livelli di diversità genetica intra- e
inter-popolazione sono stati stimati tramite l’eterozigosità e i
coefficienti di Nei (1973) e la significatività dei valori del Gst
è stata testata utilizzando il metodo suggerito da Paoli e Franceschi
(1990).
Le distanze genetiche tra le popolazioni
sono state calcolate con il metodo di Reynolds et al. (Reynolds et al., 1983)
attraverso il programma Phylip 3.5 (Felsenstein, 1989), utilizzando come
confronti i dati gentilmente concessi dal Prof. L.L. Cavalli-Sforza e dal Prof.
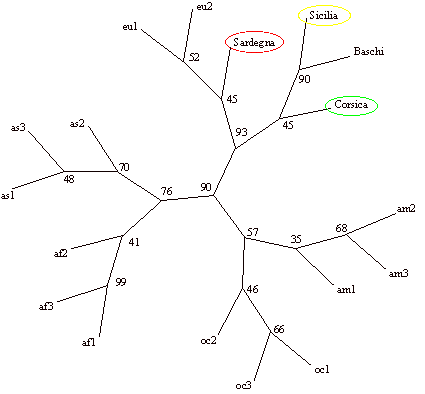
P. Moral per la tesi di dottorato di uno di noi (Calò, 1997). Per la costruzione
del dendrogramma dalla matrice delle distanze genetiche è stato
utilizzato il metodo neighbor-joining (Saitou e Nei 1987). Le popolazioni prese
in esame per i confronti sono: EU1=Nord Italia; EU2=Nord Europa; BAS= Baschi;
AF1=Pigmei Biaka dell’Africa centrale; AF2= Lisongo del Gabon; AF3=
Pigmei Mbuti dello Zaire; AS1=Cina; AS2= Giappone; AS3= Cambogia; AM1= Maya
dello Yucatan, Messico; AM2= Karitiana dell’Amazzonia, Brasile; AM3=
Surui dell’Amazzonia, Brasile; OC1= Melanesiani di Bouganville, Isole
Salomone; OC2= Nuova Guinea; OC3= Australia.
Risultati
Distribuzione
delle frequenze alleliche
Le frequenze alleliche dei 5 STRs
esaminati nelle popolazioni delle tre isole mediterranee con i loro errori
standard sono mostrate nella tabella 2.
Locus GABRB3
Per il microsatellite GABRB3 sono stati
trovati complessivamente 13 alleli, con alcune particolarità, e la
taglia degli alleli varia da 175 a 210. La distribuzione delle frequenze appare
estremamente variabile, ma in tutte le popolazioni l’allele più
frequente è risultato il 179, seguito dal 181 nei sardi e dal 183 nei
Siciliani e nei Corsi. La distribuzione delle frequenze appare bimodale in
Sardegna e in Sicilia e multimodale in Corsica. Nella popolazione sarda
è stata riscontrata la presenza di un allele raro, il 175.
Locus D15S108
La taglia degli alleli per il marcatore
D15S108 ha un range di 141-163 e sono stati trovati 12 alleli, sebbene alcuni
di essi (141, 147, 149) presentano frequenze molto basse e sono presenti solo
in alcune popolazioni. L’allele 157 si è dimostrato essere il
più frequente in tutte le popolazioni, con una frequenza massima (0.583)
nel campione siciliano. La distribuzione delle frequenze appare bimodale con i
picchi più alti in corrispondenza degli alleli 143 e 157.
Locus LIPC
Il marcatore LIPC ha un range di 165-177
bp e mostra 6 differenti alleli, con una distribuzione bimodale e con i picchi
maggiori sugli alleli 167 e 171 in tutte le popolazioni. L’allele con
frequenze maggiori in tutte le popolazioni esaminate è risultato il 167,
con un range di frequenza tra 0.567 nel campione siciliano e 0.649 nel campione
sardo.
Locus D13S115
Per il locus D13S115 sono stati trovati
complessivamente 8 alleli (161-175 bp). L’allele 165 è risultato
essere il più frequente in tutte le popolazioni esaminate. La
distribuzione delle frequenze appare unimodale.
Locus D13S270
Sono stati riscontrati 9 alleli per il
locus D13S270, con una taglia di 79-97. La distribuzione delle frequenze,
sebbene variabile nelle diverse popolazioni, risulta bimodale, con i picchi
sugli alleli 79 e 89. Alcuni alleli mostrano frequenze estremamente basse e
sono presenti solo in alcune popolazioni: gli alleli 81, 85 e 97 solo nel
campione sardo e l’allele 93 è stato trovato solo nei corsi. Non
è stato trovato l’allele 83 nei nostri campioni.
|
GABRB3 |
Sardegna |
Sicilia |
Corsica |
|
LIPC |
Sardegna |
Sicilia |
Corsica |
|
175 |
0.005 ± 0.005 |
0.000 ± 0 |
0.000 ± 0 |
|
165 |
0.102 ± 0.023 |
0.133 ± 0.024 |
0.093 ± 0.019 |
|
179 |
0.437 ± 0.048 |
0.442 ± 0.041 |
0.434 ± 0.042 |
|
167 |
0.649 ± 0.058 |
0.567 ± 0.049 |
0.613 ± 0.050 |
|
181 |
0.116 ± 0.025 |
0.085 ± 0.018 |
0.053 ± 0.015 |
|
169 |
0.015 ± 0.009 |
0.033 ± 0.012 |
0.061 ± 0.016 |
|
183 |
0.074 ± 0.020 |
0.100 ± 0.020 |
0.094 ± 0.020 |
|
171 |
0.224 ± 0.034 |
0.212 ± 0.030 |
0.202 ± 0.029 |
|
185 |
0.037 ± 0.014 |
0.085 ± 0.018 |
0.041 ± 0.013 |
|
173 |
0.010 ± 0.007 |
0.050 ± 0.014 |
0.032 ± 0.011 |
|
187 |
0.032 ± 0.013 |
0.031 ± 0.011 |
0.049 ± 0.014 |
|
177 |
0.000 ± 0 |
0.004 ± 0.004 |
0.000 ± 0 |
|
189 |
0.021 ± 0.011 |
0.023 ± 0.009 |
0.012 ± 0.007 |
|
D13S115 |
Sardegna |
Sicilia |
Corsica |
|
191 |
0.079 ± 0.020 |
0.062 ± 0.015 |
0.074 ± 0.017 |
|
161 |
0.021 ± 0.010 |
0.030 ± 0.011 |
0.025 ± 0.010 |
|
193 |
0.089 ± 0.022 |
0.062 ± 0.015 |
0.090 ± 0.019 |
|
163 |
0.021 ± 0.010 |
0.004 ± 0.004 |
0.025 ± 0.010 |
|
195 |
0.063 ± 0.018 |
0.054 ± 0.014 |
0.037 ± 0.012 |
|
165 |
0.546 ± 0.053 |
0.515 ± 0.044 |
0.425 ± 0.042 |
|
197 |
0.026 ± 0.012 |
0.058 ± 0.015 |
0.078 ± 0.018 |
|
167 |
0.201 ± 0.032 |
0.263 ± 0.031 |
0.271 ± 0.034 |
|
199 |
0.005 ± 0.005 |
0.019 ± 0.009 |
0.012 ± 0.007 |
|
169 |
0.129 ± 0.026 |
0.078 ± 0.017 |
0.175 ± 0.027 |
|
201 |
0.016 ± 0.009 |
0.008 ± 0.006 |
0.025 ± 0.010 |
|
171 |
0.026 ± 0.012 |
0.026 ± 0.010 |
0.058 ± 0.016 |
|
D15S108 |
Sardegna |
Sicilia |
Corsica |
|
173 |
0.046 ± 0.015 |
0.063 ± 0.015 |
0.017 ± 0.008 |
|
141 |
0.000 ± 0 |
0.004 ± 0.004 |
0.000 ± 0 |
|
175 |
0.010 ± 0.007 |
0.022 ± 0.009 |
0.017 ± 0.008 |
|
143 |
0.085 ± 0.021 |
0.150 ± 0.025 |
0.141 ± 0.023 |
|
D13S270 |
Sardegna |
Sicilia |
Corsica |
|
145 |
0.030 ± 0.012 |
0.013 ± 0.007 |
0.050 ± 0.014 |
|
79 |
0.364 ± 0.044 |
0.468 ± 0.043 |
0.446 ± 0.043 |
|
147 |
0.000 ± 0 |
0.000 ± 0 |
0.011 ± 0.006 |
|
81 |
0.005 ± 0.005 |
0.000 ± 0 |
0.000 ± 0 |
|
149 |
0.005 ± 0.005 |
0.000 ± 0 |
0.000 ± 0 |
|
85 |
0.038 ± 0.014 |
0.000 ± 0 |
0.000 ± 0 |
|
151 |
0.015 ± 0.009 |
0.008 ± 0.006 |
0.000 ± 0 |
|
87 |
0.190 ± 0.032 |
0.039 ± 0.012 |
0.004 ± 0.004 |
|
153 |
0.050 ± 0.016 |
0.046 ± 0.014 |
0.031 ± 0.011 |
|
89 |
0.386 ± 0.046 |
0.205 ± 0.028 |
0.244 ± 0.032 |
|
155 |
0.115 ± 0.024 |
0.083 ± 0.019 |
0.107 ± 0.020 |
|
91 |
0.011 ± 0.008 |
0.276 ± 0.033 |
0.285 ± 0.034 |
|
157 |
0.580 ± 0.054 |
0.583 ± 0.049 |
0.542 ± 0.045 |
|
93 |
0.000 ± 0 |
0.000 ± 0 |
0.004 ± 0.004 |
|
159 |
0.090 ± 0.021 |
0.088 ± 0.019 |
0.080 ± 0.017 |
|
95 |
0.000 ± 0 |
0.008 ± 0.006 |
0.017 ± 0.008 |
|
161 |
0.020 ± 0.010 |
0.025 ± 0.010 |
0.027 ± 0.010 |
|
97 |
0.005 ± 0.005 |
0.000 ± 0 |
0.000 ± 0 |
|
163 |
0.010 ±0.007 |
0.000 ±0 |
0.012 ± 0.007 |
|
|
|
|
|
Tabella 2. Frequenze alleliche dei 5 marcatori
esaminati con relativi errori standard.
Table 2. Allele frequencies for the 5 markers
examined with standard errors.