

Romagno, D., Chiarelli, B., Sineo, L., 2001. Studio dell'evoluzione dei cromosomi dell'uomo attraverso il mappaggio di loci specifici nei primati non umani: il caso del cromosoma 15. Antropo, 0, 45-52. www.didac.ehu.es/antropo
Studio dell'evoluzione dei cromosomi dell'uomo attraverso il mappaggio di loci specifici nei primati non umani: il caso del cromosoma 15
Romagno Daniela1, Chiarelli Brunetto1, Luca Sineo2
1-Dipartimento di Biologia animale e Genetica "Leo Pardi". Laboratori di Antropologia. Università di Firenze, Italia.
2-Dipartimento di Biologia animale. Università di Palermo.
Corrispondenza: Luca Sineo. Dipartimento di Biologia animale. Università di Palermo. Via Archirafi 18, 90123, Palermo, Italia. Tel: 0039 - 91 3 6230139. E-mail: llsineo@unipa.it
Parole chiave: Cromosoma 15, Sonde locus specifiche, Primati, Cercopithecoidea, Hominoidea
La tecnica del painting cromosomico ha permesso di definire la forte conservazione delle sintenie geniche del cromosoma 15 dell'uomo nei primati non umani. Nonostante questo, sebbene un omologo con bandeggiamento simile o identico al cromosoma 15 sia presente nelle scimmie antropomorfe, non c'è una chiara controparte di questo cromosoma negli altri primati. Infatti, il painting cromosomico è uno strumento molto potente per mettere in evidenza riarrangiamenti intercromosomici ma inadeguato per il rilevamento di riarrangiamenti intracromosomici. In questo lavoro viene descritta la localizzazione cromosomica dei loci GABRB3 e PML, che nell'uomo mappano rispettivamente in 15q11-13 e 15q22, in Macaca fascicularis e Cercopithecus aethiops. L'ibridazione ha messo in evidenza la posizione pericentromerica del locus PML e la posizione subtelomerica nei cromosomi omologhi dei primati esaminati. Sulla base dei risultati ottenuti viene proposto uno schema filogenetico per il cromosoma 15 dell'uomo negli Hominoidea e nei Cercopithecoidea. Dal momento che esiste una associazione sintenica dei cromosomi 14 e 15 molto conservata nei mammiferi, la ricostruzione coinvolge anche il cromosoma 14. L'utilizzo di sonde locus specifiche si dimostra, quindi, un utile strumento per lo studio dei riarrangiamenti intracromosomici e dell'evoluzione cromosomica in generale.
Chromosome painting approach defined a strong conservation of HSA 15 syntenies in non-human Primates, In spite of this evidence, although a homologous to human chromosome 15 with similar or identical banding pattern is present in great apes, there is not clear counterpart of this chromosome in the other primates. In fact,
chromosome painting is a powerful tool in case of interchrosomal rearrangements, but not in case of intrachromosomal rearrangements.
We report the in situ mapping of GABRB3 and PML single locus probes on Macaca fascicularis and Cercopithecus aethiops chromosomes. In situ hybridisation indicates a PML pericentromeric localisation and a GABRB3 subtelomeric localisation in the homologous chromosomes of the non-human primates tested. In man GABRB3 and PML map respectively at 15q11-13 and 15q22.
A reconstruction of HSA 15 chromosome phylogeny in Hominoidea and in Cercopithecoidea is presented. As the 14/15 syntenic association is a conservative syntenic association in mammals, this recontruction involved also chromosome 14.
Single locus and subchromosomal in situ hybridisation is a useful tool in the investigation of intrachromosomal rearrangements and in chromosomal evolution.
La citogenetica molecolare si è dimostrata uno strumento molto valido per la comparazione cromosomica interspecifica e per la definizione del ruolo dei riarrangiamenti cromosomici nell'evoluzione animale (O'Brien et al., 1999; O'Brien and Stanyon, 1999).
Il cosiddetto "painting" cromosomico consente di inviduare estese omologie tra specie filogeneticamente molto distanti ed una efficace verifica delle ipotesi proposte mediante la comparazione dei "patterns" di bandeggiamento (O'Brien et al., 1999; Wienberg et al., 1990). Questo approccio ha permesso di stabilire una forte conservatività dei gruppi sintenici nei mammiferi ma è indicato solo per la rilevazione di riarrangiamenti intercromosomici; non può, invece, essere di alcun aiuto nella rilevazione di riarrangiamenti intracromosomici. In questo caso un approccio più efficace ma ancora poco applicato è quello che prevede l'uso di sonde subcromosomiche o locus specifiche (Wienberg et al., 1990; Romagno et al., 2000).
L'utilizzo di sonde locus specifiche o comunque di piccole librerie subcromosomiche nello studio dell'evoluzione dei cromosomi umani è stato fondamentalmente limitato alle antropomorfe, nel tentativo di verificare i riarrangiamenti proposti sulla base dell'analisi dei pattern di bandeggiamento (Samonte et al., 1997; Verma e Luke, 1994; Verma et al., 1996; Luke e Verma, 1995; Muller et al., 1996; Wienberg et al., 1994; Haaf e Bray-Ward, 1996; Tanabe et al., 1996; Mc Conkey et al., 1996; McConkey, 1997). Dati su altri primati si limitano al mappaggio fisico di alcuni loci in Aotus (Ma e Gerhard, 1992; Ma e Lin, 1992), alla localizzazione del gene RB1 (Retinoblastoma ) in Macaca, Cercopithecus aethiops e Pithecia pithecia (Tihy et al., 1996) ed al riconoscimento di un complesso riarrangiamento cromosomico, in Macaca, ottenuto mediante una sonda subcromosomica del cromosoma 3 umano (Muller et al., 1996).
Relativamente al cromosoma 15 dell'uomo la comparazione dei "patterns" di bandeggiamento ha consentito di stabilire che il cromosoma dell'uomo e gli omologhi in Gorilla e Pongo possono essere considerati ancestrali per gli Hominoidea mentre il cromosoma di Pan può essere derivato da una inversione pericentrica (Yunis e Prakash, 1982); non è stata, tuttavia, ritrovata una chiara controparte di questo cromosoma in altri primati. Con la tecnica del "painting" sono state confermate le omologie ipotizzate sulla base del bandeggiamento (Jauch et al., 1992) e sono stati individuati i segmenti cromosomici omologhi al cromosoma 15 in molti altri primati; è stata anche evidenziata la conservatività della sintenia 15/14 nei mammiferi (Haig, 1999). Questo approccio non ha, tuttavia, permesso di risalire, a partire dalle omologie individuate tramite il "painting" nelle specie più strettamente imparentate, ai cambiamenti cromosomici che hanno portato alla forma ancestrale del cromosoma 15 di uomo, Pan Gorilla e Pongo.
Nel presente lavoro abbiamo affrontato questo problema mediante la localizzazione dei loci GABRB3 (Recettore b-3 dell'Acido Gamma aminobutirrico) e PML (Leucemia Promielocitica) in Macaca fascicularis e Cercopithecus aethiops. La scelta dei loci, che nell9uomo mappano rispettivamente in 15q11-13 e 15q22, è stata condizionata dal fatto che questi erano già stati localizzati nelle antropomorfe in un precedente lavoro nel quale era stata confermata l'occorrenza della inversione pericentrica, ipotizzata sulle basi del bandeggiamento, in Pan (Luke e Verma, 1995). La scelta di un rappresentante del genere Macaca è stata fatta in considerazione del fatto che i generi Macaca e Papio, cromosomicamente molto conservativi, sono quelli più spesso utilizzati come "outgroup" negli studi sull'evoluzione cromosomica degli Hominoidea mentre Cercopithecus aethiops è stato scelto come rappresentante di un genere caratterizzato da una notevole variabilità cromosomica. Le scelte operate hanno consentito, inoltre, di conoscere la localizzazione sui cromosomi di due primati non umani abbondantemente utilizzati nell'ambito delle ricerca biomedica, di due loci implicati, nell'uomo, nelle sindromi di Prader-Willi/Angelman (GABRB3) e nella leucemia promielocitica (PML).
Colture cellulari e colorazioni differenziali.
Colture a breve termine di sangue periferico di Macaca fascicularis (di seguito MFA) e Cercopithecus aethiops (di seguito CAE) sono state allestite secondo le correnti metodologie (Small et al., 1985). I vetrini sono stati "invecchiati" per 2-4 ore a 60°C e successivamente bandeggiati con la tecnica GTG per permettere un adeguato riconoscimento dei cromosomi ibridati.
Ibridazione in situ con la libreria del cromosoma 15 e con le sonde GABRB3 e PML.
I vetrini sono stati deidratati in serie crescente di etanolo (70%, 80%, 100%) per 2 minuti, a temperatura ambiente; sono stati quindi denaturati per 2 minuti a 70°C in a 70% Formamide /2xSSC, pH 7.3 e quindi nuovamente deidratati in serie crescente di etanolo. Le sonde biotinate (Oncor, Inc. Gaithersburg MD, USA) dopo un preriscandamento a 37°C per 10 minuti, sono state applicate (10 ml) sui vetrini preriscaldati. Dopo l'incubazione per 16-20 ore a 37°C in camera umida i vetrini sono stati lavati per 15 minuti in 50% formamide/2xSSC, a 43°C e per 8 minuti in PBD a 37°C. Dopo aver aggiunto 60 ml di anti-digoxigenina /FITC i vetrini sono stati posti al buio ad una temperatura di 37°C . Dopo l'incubazione i vetrini sono stati sottoposti a tre successivi lavaggi di due minuti in 1xPBD seguiti da controclororazione con ioduro di propidio. I vetrini sono stati montati per l'osservazione in una soluzione di Fenilendiamina /PBS/Glicerina.
Ibridazione in situ con sonda PML
In questo caso è stata utilizzata una sonda - LSI ® Locus Specific Identifier DNA probe - Spectrum Orange (Vysis Inc. Downers Grove, IL, USA). Una mistura di sonda fluorescente e DNA bloccante è stata denaturata a 73°C ; 10 ml di sonda sono stati quindi applicati su vetrini preriscaldati a 37 °C. Dopo una ibridazione di 16 ore in camera umida a 37°C si è proceduto ad un lavaggio per 10 minuti in 50% Formamide/2xSSC a 46°C, seguito da un lavaggio in 2xSSC e quindi in 2XSSC/0.1% NP-40 46°C per 5 minuti. I vetrini sono stati controcolorati con 10 ml di DAPI.
Tutti gli esperimenti di ibridazione con le varie sonde sono stati condotti in parallelo con controlli di Homo sapiens (di seguito HSA ).
Cattura del segnale e analisi del dato.
I segnali di ibridazione sono stati catturati con una telecamera COHU CCD Camera montata su microscopio a luce fluorescente Zeiss Axiophot.
I cromosomi di MFA sono stati identificati secondo Small et al., (1985) e Wienberg et al., (1992); per i cromosomi di CAE si è utilizzata la classificazione di Sineo et al. (1986) e Finelli et al. (1999).
1) L'ibridazione con la sonda "painting" 15 dell'uomo produce due segnali su due diversi acrocentrici in CAE. In pratica si ha una ibridazione pressoché totale con il cromosoma 26 ed una ibridazione in corrispondenza della porzione prossimale del cromosoma 29.
2) L'ibridazione con le sonde GABRB3 e PML produce due segnali che in MFA sono localizzati in prossimità del centromero ed in posizione subtelomerica sul braccio corto del cromosoma 7 ed in CAE nelle stesse posizioni sul cromosoma 26.
3) L'ibridazione con la sonda del solo PML produce di un segnale in prossimità del centromero sul braccio corto del cromosoma 7 in MFA e nella stessa posizione sul cromosoma 26 di CAE.
Il cromosoma 15 di Homo (HSA 15) ed i suoi omologhi in Gorilla e Pongo sono considerati ancestrali per gli Hominoidea, mentre in Pan l'omologo è derivato da una inversione pericentrica (Yunish e Prahash, 1982; Luke e Verma, 1995). In Macaca e Papio, cromosomicamente conservativi e spesso utilizzati come "outgroup" nell'analisi citogenetica comparata delle Hominoidea, l'uso di sonde "painting" ha dimostrato che il cromosoma 15 umano è omologo a parte del cromosoma 7, e precisamente all'intero braccio p e parte del q; la restante parte del 7q ibridizza con la libreria umana del cromosoma 14 (Wienberg et al., 1992; Morescalchi et al., 1998; Best et al., 1998). L'analisi del bandeggio del cromosoma 7 dei Papionini permette, infatti, di identificare una chiara analogia con il 14 umano, mentre risulta di difficile interpretazione l9analogia della rimanente parte con il bandeggio del 15 umano. Appare chiaro, quindi, che riarrangiamenti complessi sono intervenuti nella porzione che ibridizza con la sonda umana 15, riarrangiamenti che non vengono evidenziati con il "painting".
Nel Cercopithecus i risultati del "painting" prodotti in questo lavoro coincidono con quelli di Finelli et al. (1999): la sonda umana del 15 identifica i cromosomi 26 e 29, con l'eccezione della porzione telomerica che ibridizza con la sonda umana 14, sonda che marca anche l'intero cromosoma 24. Il bandeggio di CAE 26 è identico a quello di MFA 7p mentre il bandeggio di CAE 29 non risulta informativo; per quanto riguarda CAE 24 l'analogia del bandeggio con gran parte di MFA 7q è piuttosto evidente.
L'analisi della posizione delle sonde GABRB3 e PML in macaca e cercopiteco, unitamente ai dati del bandeggio e del "painting" consente di ricostruire i riarrangiamenti complessi che hanno portato al cromosoma HSA 15 che, come esposto precedentemente, può essere considerato ancestrale per gli Hominoidea; inoltre, unitamente ai dati presenti in letteratura sui cariotipi bandaggiati e sul "painting" di varie specie, consente di proporre uno schema evolutivo dei cromosomi corrispondenti nei Cercopithecoidea. In conseguenza della sintenia 15/14 sono state ottenute informazioni anche riguardo l'evoluzione di quest'ultimo cromosoma.
Filogenesi negli Hominoidea.
A partire da un cromosoma simile al 7 di macaca, una inversione pericentrica avrebbe portato alla formazione di un cromosoma metacentrico caratterizzato da tutto il materiale genetico relativo a HSA 15 sul braccio p ed il materiale genetico relativo a HSA 14 sul braccio q. Un fenomeno di fissione avrebbe creato due cromosomi acrocentrici, omologhi a HSA 15 e HSA 14. Per spiegare il "pattern" di bandeggiamento e la posizione dei loci mappati in Homo sapiens è necessario ipotizzare l'insorgenza di una vasta inversione paracentrica, avvenuta prima o dopo l'evento di fissione (Fig. 1). Un' ipotesi alternativa implica, sempre a partire da un cromosoma simile a MFA 7, l'occorrenza di una fissione e la attivazione di un centromero latente nel braccio q, in corrispondenza del limite tra i domini delle sonde 14 e 15: in seguito a questi riarrangiamenti si sarebbero originati due cromosomi acrocentrici corrispondenti ai 14 e 15 umani; in seguito sul cromosoma corrispondente al 15 si sarebbe avuta la trasposizione del centromero in posizione q terminale. L'attivazione e lo shift centromerico sono eventi poco descritti in letteratura e quindi questa ipotesi alternativa, peraltro meno parsimoniosa, appare improbabile rispetto alla precedente.
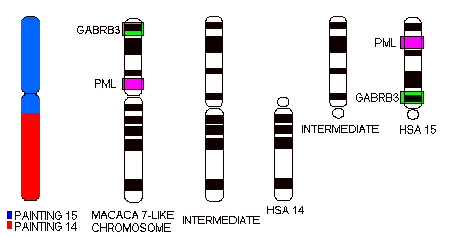
Figura 1. Filogenesi del cromosoma 15 nelle Hominoidea (vedere il testo).
Figure 1. Phylogeny of chromosome 15 in Hominoidea (see text)
Filogenesi nei Cercopithecoidea
I Papionini sono caratterizzati da una forte conservatività sia del numero e della morfologia cromosomica che del bandeggio. Un cromosoma praticamente identico a MFA 7 si ritrova in tutte le specie esaminate (M. mulatta, M. nemestrina, M. sylenus, M. radiata, M. fuscata, M. arctoides, Papio anubis, P. ursinus, P. hamadryas, P. cynocephalus, Mandrillus sphynx, M. leucophaeus, Theropithecus gelada, Cercocebus aterrhimus) (Stanyon et al., 1988; Fantini comunicazione personale). Relativamente alle Colobinae esaminate (Presbytis rubicundus, P. crystata, Colobus guereza, Nasalis larvatus), se si esclude Nasalis, dove non è stata evidenziata una omologia sicura, è stato ritrovato un cromosoma praticamente identico al cromosoma 7 dei Papionini (Fantini, comunicazione personale). Sia nel caso dei Papionini che nel caso delle Colobinae i dati disponibili sul "painting" cromosomico confermano le osservazioni fatte sulle basi del bandeggio (Bigoni et al., 1997a; Bigoni et al., 1997b; Morescalchi et al., 1997; Wienberg et al.; Best et al.).
Per quanto riguarda i Cercopithecini, oltre a C. aethiops, sono stati esamiminati i dati presenti in letteratura relativi ai cariotipi bandeggiati di: C. mitis (maesi), C. albogoularis e C. nictitans (stampflii) (Sineo, 1990); Allenopithecus nigroviridis, Erythrocebus patas, Miopithecus talapoin, Cercopithecus diana, C. mona, C. petaurista, C. cephus (Clemente et al., 1990); Cercopithecus l'hoesti, C. ascanius, C. petaurista, C. hamlyni e C. diana (Sineo, 1986). Da questa analisi è stato rilevato che Allenopithecus nigroviridis è caratterizzato da un submetacentrico identico al 7 dei Papionini (Clemente et al., 1990) e che in Erythrocebus patas e Miopithecus talapoin sono presenti un acrocentrico (EPA 25, MTA 24 nella numerazione proposta da Clemente et al., 1990) e un submetacentrico (EPA 11 and MTA 9) apparentemente omologhi di Macaca 7p e 7q. Per quanto riguarda le altre specie non è stato possibile trovare chiare omologie né con il cromosoma 7 dei Papionini né con i cromosomi 24, 26 e 29 di CAE.
Sulla base di questa analisi proponiamo un cromosoma simile a MFA 7 come cromosoma ancestrale per i Cercopithecoidea, dato che si ritrova immutato nei Papionini, nelle Colobinae ed in Allenopithecus nigroviridis, considerato un rappresentante ancestrale dei Cercopithecini (Napier, 1981; Hill, 1966). A partire da questo cromosoma si sarebbe verificata una fissione che ha dato origine a due cromosomi acrocentrici: uno corrispondente a CAE 26, EPA 25 e MTA 24, l'altro corrispondente a CAE 26+29. Una inversione pericentrica in quest'ultimo cromosoma avrebbe dato origine EPA 11 e MTA 9. Una seconda fissione avrebbe generato i cromosomi CAE 26 e CAE 29 (Fig. 2).
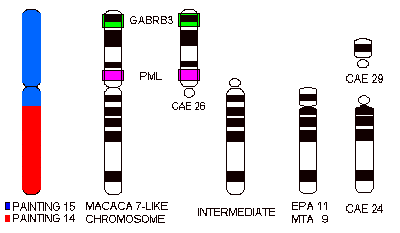
Figura 2. Filogenesi del cromosoma 15 nella Cercopithecoidea (vedere il testo)
Figure 2. Phylogeny of Chromosome 15 in Cercopithecoidea (see text)
Lo studio qui riportato dimostra chiaramente l'utilità dell'uso di sonde locus specifiche nello studio dell'evoluzione cromosomica. Il "painting" mediante sonde cromosomiche o subcromosomiche è infatti utilizzabile solo per la rilevazione di riarrangiamenti intercromosomici ma non è informativo nella verifica di riarrangiamenti intracromosomici ipotizzati in base all'analisi dei "patterns" di bandeggiamento né tantomeno nei casi in cui si sospetta esistano dei riarrangiamenti intracromosomici complessi non definibili con la sola analisi del bandeggiamento.
L'estensione di questo approccio ad un maggior numero di specie utilizzando un gran numero di sonde risulterà, a nostro avviso, fondamentale nella comprensione dei meccanismi fini di evoluzione cromosomica.
Ringraziamenti. Gli autori ringraziano Maria Luisa Giovannucci-Uzielli (Az. Osp. Meyer e Università di Firenze) che ha messo a disposizione le sonde cromosomiche, Roscoe Stanyon (National Cancer Inst., Fredericks, MA, USA) per gli utili suggerimenti sul testo, Giandonato Tartarelli (Scuola Normale Superiore, Università di Pisa) per l'analisi delle immagini e Camilla Fantini (Firenze) per i suoi dati sulla citogenetica dei Papionini.
Il lavoro è stato finanziato con fondi M.U.R.S.T. accordati a L. Sineo e B. Chiarelli. Daniela Romagno è fruitrice di un Assegno di Ricerca finanziato dalla società Prosperius srl di Firenze.
Best RG, Diamond D, Crawford E, Grass FS, Janish C, Lear TL, Soenksen D, Szalay AA, Moore CM, 1998, Baboon/human homologies examined by spectral karyotiping (SKY): a visual comparision. Cytogenet Cell Genet 82, 83-87.
Bigoni F, Stanyon R, Koheler U, Morescalchi AM, Wienberg J, 1997a, Mapping homology between human and Black and White Colobine monkey chromosomes by fluorescent in situ hybridization. Am J of Primatology 42, 289-298.
Bigoni F, Koheler U, Stanyon R, Ishida T, Wiemberg J, 1997b, Fluorescence in situ hybridization estabilishes homology between human and sylvered leaf monkey chromosomes, reveals reciprocal translocations between chromosomes homologous to human Y/5, 1/9, 6/16 and delineates an X1X2Y1Y2/X1X1X2X2 sex-chromosome system. Am J Physical Anthropology 23, 315-327.
Clemente IC, Ponsà M, Garcia M, Egozcue J, 1990, Chromosome Evolution in the Cercopithecidae and its relationship to Human fragile sites and neoplasia. Int J Primatology 11(4), 377-399.
Finelli P, Stanyon R, Plesker R, Ferguson-Smith MA, O'Brien PCM, Wienberg J, 1999, Reciprocal chromosome "painting" shows that the great difference in diploid number between human and African green monkey is mostly due to non-Robertsonian fissions. Mammalian Genome 10, 713-718.
Haaf T, Bray-Ward P, 1996, Region-specific YAC banding and painting probes for comparative genome mapping: implications for the evolution of human chromosome 2. Chromosoma 104(8), 537-544.
Haig D , 1999, A brief history of Human autosomes. Phil. Trans R Soc Lond B 354, 1447-1470.
Hill WCO, 1966, Primates. Comparative anatomy and Taxonomy. Vol VI Catarrhini, Cercopithecoidea. (Edimburgh University Press).
Jauch A, Wienberg J, Stanyon R, Arnold N, Tofanelli S, Ishida T, Cremer T, 1992, Reconscruction of genomic rearrangements in great apes and gibbons by chromosome painting. Proc Natl Acad Sci USA 89, 8611-8615.
Luke S., Verma RS, 1995, The genomic sequence for Prader-Willi/Angelman syndromes' loci of human is apparently conserved in the great apes. Journal Molecular Evolution 41, 250-252.
Ma NS, Gerard DS, 1992, Mapping of five human chromosome 19 DNA markers to owl monkey chromosomes. Cytogenet Cell Genet 59(1), 57-62.
Ma NS, Lin KC, 1992, Chromosome mapping of the owl monkey CSF1R and IL5 genes. Genomics 13(4), 1174-1177.
McConkey EH, Menon R, Williams G, Baker E. Sutherland GR, 1996, Assignementof the gene for beta-casein (CSN2) to 4q13-q21 in humans and 3p13-p12 in chimpanzee. Cytogenet Cell Genet 72(1), 60-62.
McConkey EH, 1997, The origin of human chromosome 18 from a human/ape ancestor. Cytogenet Cell Genet 76(3-4), 189-191.
Morescalchi AM, Camperio Ciani A, Stanyon R, 1998, Chromosome banding and molecular cytogenetics of the Barbary macaque, Macaca Sylvanus. Ital J Zool 65, 101-107.
Muller S, Koheler U, Wienberg J, Marzella R, Finelli P, Antonacci R, Rocchi M, Archidiacono N, 1996, Comparative fluorescence in situ hybridization mapping of primate chromosomes with Alu polymerase chain reaction generated probes from human/rodent somatic cell hybrids. Chromosome Res 4(1), 38-42.
Napier PH, 1981, Catalogue of primates in British Museum (Nat. Hist.) Part II: Family Cercopithecida, subfamily Cercopithecinae. London British Museum.
O'Brien SJ, Stanyon R , 1999, Ancestral primate viewed. Nature 402, 365-366.
O'Brien SJ, Menotti - Raymond M, Murphy WJ, Nash WG, Wienberg J, Stanyon R, Copeland NG, Jenkins NA, Womack JE, Marshall Graves J, 1999, The promise of comparative genomics in mammals. Science 286, 458-481.
Romagno D, Chiarelli B, Guarducci S, Giovannucci Uzielli ML, Sineo L, 2000, Chromosome mapping of GABRB3 and PML loci in Macaca and Cercopithecus indicates the mechanism of evolution of human chromosome 15. Cromomosome Res 8, 747-749.
Samonte RV, Conte RA, Verma RS, 1997, Comparative mapping of the estrogen receptor (ESR) and the Kallmann (KAL) regions to the chromosomes of the great apes. Am J Phys Anthropol 103(4), 561-563.
Sineo L, Stanyon R, Chiarelli B, 1986, Chromosomes of the Cercopithecus aethiops species group: Cercopithecus aethiops (Linnaeus 1758), Cercopithecus cynosurus (Scopoli 1786), Cercopithecus pygerythrus (Cuvier 1821) and Cercopithecus sabaeus (Linnaeus 1776). Int J Primatol 7, 569-582.
Sineo L, 1986, Cariologia ed evoluzione del genere Cercopithecus. Tesi di Dottorato Università di Firenze.
Sineo L, 1990, The banded karyotypes of Cercopithecus mitis maesi compared with the karyotype of C. albogularis samango and C. nictitans stampflii. Int J Primatol 11 (6), 541-552.
Small MF, Stanyon R, Smith DG, Sineo L, 1985, High resolution chromosomes of Rhesus macaques (Macaca mulatta). Am J Primatol 9, 63-67.
Stanyon R, Fantini C, Camperio-Ciani A, Chiarelli B, Ardito G, 1988, Banded karyotypes of 20 Papionini species reveal no necessary correlation with speciation. Am J Primatol 16, 3-17 .
Tanabe H, Ishida T, Ueda S, Sofuni T, Mizusawa H, 1996, Molecular anatomy of human chromosome 9: comparative mapping of the immunoglobulin processed pseudogene C epsilon 3 (IGHEP2) in primates. Cytogenet Cell Genet 76(1-2), 92-96.
Tihy F, Lemieux N, Lombard M, Dutrillaux B , 1996, Comparative RB1 gene mapping in Homo sapiens, Pithecia pithecia, Macaca sylvana, and Cercopithecus aethiops tantalus. Cytogenet Cell Genet 72, 9-11.
Verma RS, Luke S , 1994, Evolutionary divergence of human chromosome 9 as revealed by the position of the ABL protooncogene in higher primates. Mol Gen Genet 243, 369-373
Verma RS, Ramesh KH, Samonte RV, Conte RA, 1996, Mapping the homolog of the human Rb1 gene to chromosome 14 of higher primates. Mamm Genome 7(8), 591-592.
Wienberg J, Jauch A, Stanyon R, Cremer T, 1990, Molecular cytotaxonomy of primates by chromosomal in situ suppression hybridization. Genomics 8, 347-350.
Wienberg J, Stanyon R, Jauch A, Cremer T, 1992, Homologies in human and Macaca fuscata chromosomes revealed by in situ suppression hybridization with human chromosomes specific DNA libraries. Chromosoma 101(5-6), 265-270.
Wienberg J, Jauch A, Ludecke HJ, Sanger G, Horsthemke B, Claussen U, Cremer T, Arnold N, Lengauer C , 1994, The origin of chromosome 2 analyzed by comparative chromosome mapping with a DNA microlibrary. Chromosome Res 2(5), 405-410.
Yunish J, Prakash O, 1982, The origin of Man: a chromosomal pictorial legacy. Science, 215, 1525-1529.